solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1chma2 ( 157 ) miKsaeehvmIrhGA
d1pv9a2 ( 125 ) ktkeeieiIekAC
d1qxya- ( 1 ) mivkteeelqaLkeIG
d1qzya2 ( 110 ) kvqtdppsvpIcdlypngvFpkgqeceypeekkalDqaseeiWndFReAA
d1wl6a2 ( 177 ) speeiavLrrAG
d1xgsa2 ( 1 ) mdteklmkAG
d2ggca- ( 2 ) aisiKtpediekMrvAG
d3tava1 ( 3 ) glfgrkktVeqrtpgeLdaMaaAG
d3tb5a- ( 1 ) mitlkspreiemMdeSG
aaaaaaaaaaa
60 70 80 90 100
d1chma2 ( 172 ) rIAdiGGaaVveaLgdqvpEyeValhAtqaMvraIa---dtfedvelmdT
d1pv9a2 ( 138 ) eIAdkAVmaAieeItegkrEreVaakVeylMkmngA---e-------kpA
d1qxya- ( 17 ) yICAkVrntMqaaTkpgitTkeLDniAkelFee-ygAiSAPihden--Fp
d1qzya2 ( 174 ) eAHrqVRkyVmswIkpgmtMieICekLedCSrkLIken-----glnaglA
d1wl6a2 ( 189 ) eITAmAHtrAMekCrpgmfEyhLegeIhhEFnrhgA---r-------ypS
d1xgsa2 ( 11 ) eIAkkVrekAiklArpgmlLleLaesIekmImel-----------gGkpA
d2ggca- ( 19 ) rlAAeVLemIepyVkpgvsTGeLDriCndyIvneQhAvSAclgyhg--Yp
d3tava1 ( 27 ) siVGaALvaVrdaAkagvsTleLdqvAesvIra--gAvPSflgyhg--Fp
d3tb5a- ( 18 ) elLAdVHrhLrtfIkpgitSwdIEvfVrdfIesh-gGvAaqigyeg--Yk
aaaaaaaaaaaaa aaaaaaaaaaaaaa
110 120 130 140 150
d1chma2 ( 219 ) w-TwFQSginTdga-hnpvtt---rkVnkgdILSLNCFPMIagyyTALeR
d1pv9a2 ( 178 ) fdTIIASghrSAlp-hGvAsd---krIergdLVVIdLGAlynhyNSDITR
d1qxya- ( 64 ) gqTcISVne---EVahGiPsk---rvIregdLVnIdVSAlkngyYADTGi
d1qzya2 ( 219 ) fpTgCSlnn---cAahytPnagdttvLqyddICKIdFGThisGrIIDCAF
d1wl6a2 ( 229 ) ynTIVGSgenGcil-hYteNe---ceMrdgdLVLIDAGCeykgyAGDITR
d1xgsa2 ( 50 ) fpVnLSine---iaahytPykgDttvLkegDyLKIdVGVhidGfIADTAv
d2ggca- ( 67 ) kSVcISine---vVChgiPdd--aklLkdgdIVNIdVTVikdgFhGDTSk
d3tava1 ( 74 ) ASICSSvnd---qVVhGiPsa--tavLadgdLVSIdCGAildgwhGDSAw
d3tb5a- ( 65 ) yATCCSind---eIchGfPrk---kvLkdgdLIKVdMCVdlkgaISDSCW
bbbb bbbbbbbbbb bb bbb
160 170 180 190 200
d1chma2 ( 264 ) TLFld-hCsddhlrlWqVNveVHeaGlklIkpgarCsdIAreLneiFlkh
d1pv9a2 ( 224 ) Tivvg-spnekqreIyeIVleAQkrAveaAkpgmtAkeLDsiAreIIkey
d1qxya- ( 108 ) SfVVgesddpmkqkVCdVAtmAFenAiakVkpgtkLsnIGkaVhnTArqn
d1qzya2 ( 266 ) Tvtfn----pkYdtLlkAVkdATntGIkcAGidvrLcdVGeaIqevMesy
d1wl6a2 ( 275 ) TFPVnGkFtqaqreIYdiVleSLetSlrlYrpgTsIleVtgeVvriMVsg
d1xgsa2 ( 97 ) TvrVg----meedeLMeAAkeALnaAisvAragveIkeLGkaIeneIrkr
d2ggca- ( 112 ) MfiVgkpt-imGerLCrITqeSLylALrmVkpginLreIGaaIqkfVeae
d3tava1 ( 119 ) TfaVgtvi-psDeaLseATrlSMeaGiaaMipgnrLtdVShaIelgTraA
d3tb5a- ( 109 ) SyvVgest-peIdrLmeVTkkALylGIeqAqvgnrigdIGhaIqtyVege
bbb aaaaaaaaaaaaaaaa aaaaaaaaaaaaaa
210 220 230 240 250
d1chma2 ( 313 ) --------------------dvlqyrtfgYGhSFGtLShyygreagLELr
d1pv9a2 ( 273 ) --------------------gygdyFihsLGhGVGlei----hew-PrIS
d1qxya- ( 158 ) ---d---------------LkVIk--nlt-GHGVGl----slheapahVl
d1qzya2 ( 312 ) ---eVeIdgk------tyqVkPIr--nln-GhsIgq---yrihagk-tVp
d1wl6a2 ( 325 ) LvklgIlkgdvdeliaqn--aHrpfFmhgLShwLgldv----hdv-gvyg
d1xgsa2 ( 143 ) ---g---------------fkPiv--nls-GHkIer---yklhAgi-sIp
d2ggca- ( 161 ) ---g---------------fsVVr--eyc-GhGIGr----gfheeP-qVl
d3tava1 ( 168 ) ---eqfd----------raFgIVd--gyG-GhgIGr----sMhldP-fLp
d3tb5a- ( 158 ) ---g---------------YgVVr--dfv-GhGIgp----tihesp-mIp
bbb bb
260 270 280 290 300
d1chma2 ( 343 ) e----didtvLepgMVVSMEPMImlpe---------g-----lpGagGYR
d1pv9a2 ( 298 ) q----ydetvLkegmVITIePGIYipk------------------lGGVR
d1qxya- ( 183 ) NyfdpkdktlLtegmVLAIePFISsnasfVtegknewAfeTs-dkSfVAQ
d1qzya2 ( 346 ) Iik-ggeatrMeegevYAIeTFGSTGkGvvDik-----------gSyTAQ
d1wl6a2 ( 368 ) q----drsriLepgmVLTVePGLYIap---------daeVpeqYrgiGIR
d1xgsa2 ( 168 ) NiyrphdnyvLkegdvFAIePFATigar----------------ngiVAQ
d2ggca- ( 185 ) HydsretnvvLkpgMTFTIePMVNagkkeirtmkdgwtVkTk-drslSAQ
d3tava1 ( 198 ) NegapgkgplLavGSVLAIePMLTlgttqTrvladdwTVvTt-dgsrAAH
d3tb5a- ( 182 ) HygeagkglrLkegMVITIePMVNtgtwrmkmdpngwTAyTe-dgglSCQ
bbbb bbb bbb
310 320 330
d1chma2 ( 375 ) EHDILIVnengAenITk-fpyGpekNii-r
d1pv9a2 ( 326 ) IEDTVlItengAkrLTk-ter
d1qxya- ( 232 ) iEhTVIVtkdgpilTTki
d1qzya2 ( 458 ) FEHTILLRPtcKEVVSrgddy
d1wl6a2 ( 405 ) IEDDIVItetgnenLTasVvkkPeeI---ealMvaarkq
d1xgsa2 ( 279 ) fEhTIIVekdsVivTTe
d2ggca- ( 234 ) yEHTIVVtdnGCeILTlRkdDt-Ipaiishd-------e
d3tava1 ( 247 ) wEHTVAVteagpriLTmrp
d3tb5a- ( 231 ) YEHSLAItkegpriLTsQgeel-ty
bbbb bb
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Creatinase, catalytic (C-terminal) domain | Creatinase/aminopeptidase | Pseudomonas putida [TaxId: 303] | 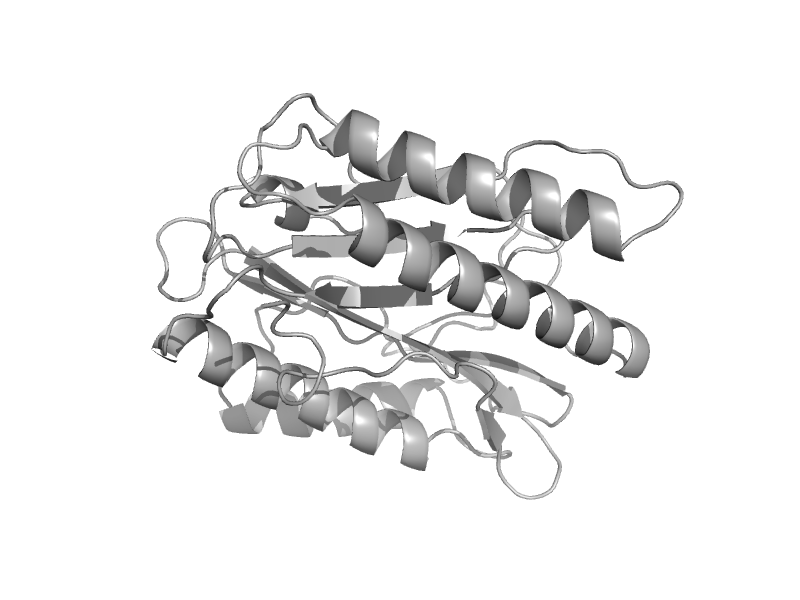 | view | |
| Aminopeptidase P, C-terminal domain | Creatinase/aminopeptidase | Pyrococcus furiosus [TaxId: 2261] | 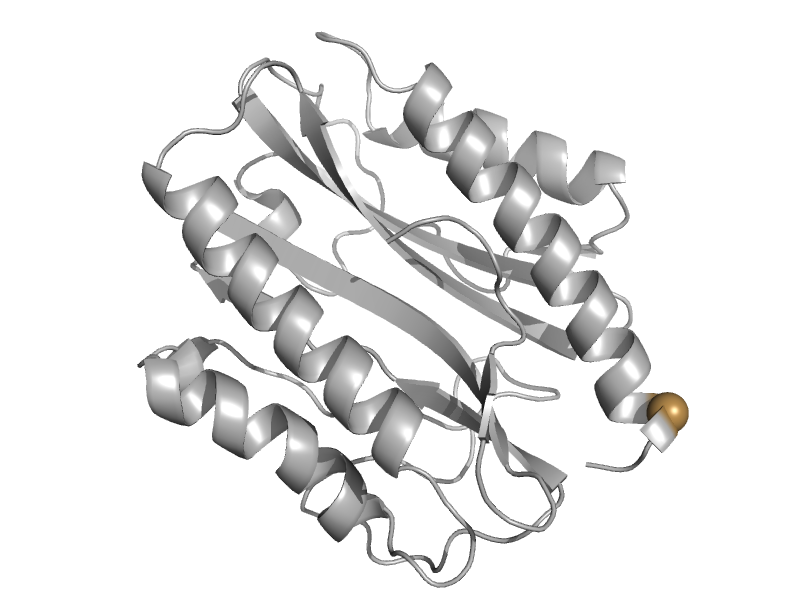 | view | |
| Methionine aminopeptidase | Creatinase/aminopeptidase | Staphylococcus aureus [TaxId: 1280] | 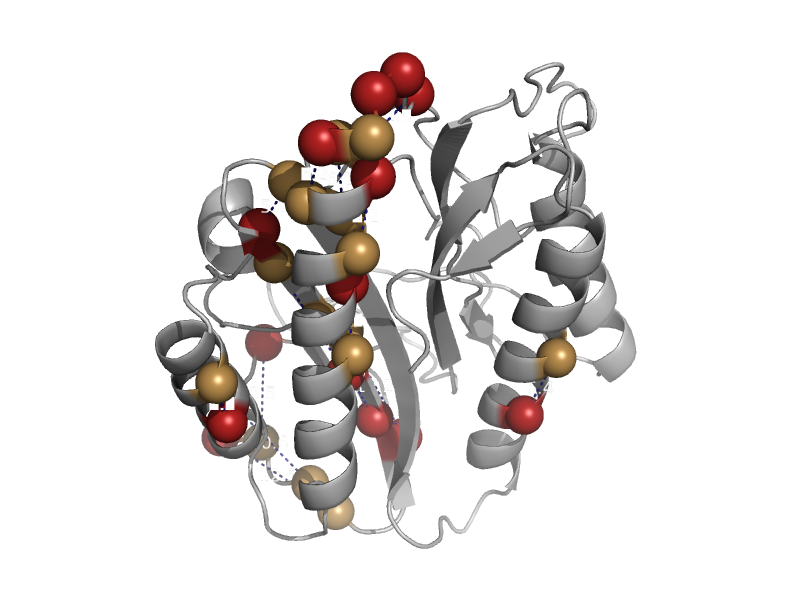 | view | |
| Methionine aminopeptidase | Creatinase/aminopeptidase | Human (Homo sapiens) [TaxId: 9606] | 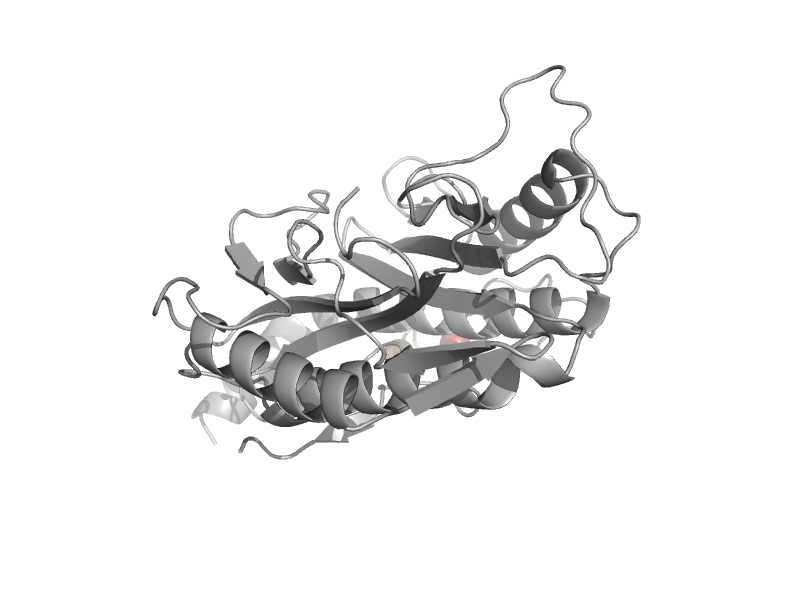 | view | |
| Aminopeptidase P, C-terminal domain | Creatinase/aminopeptidase | Escherichia coli [TaxId: 562] | 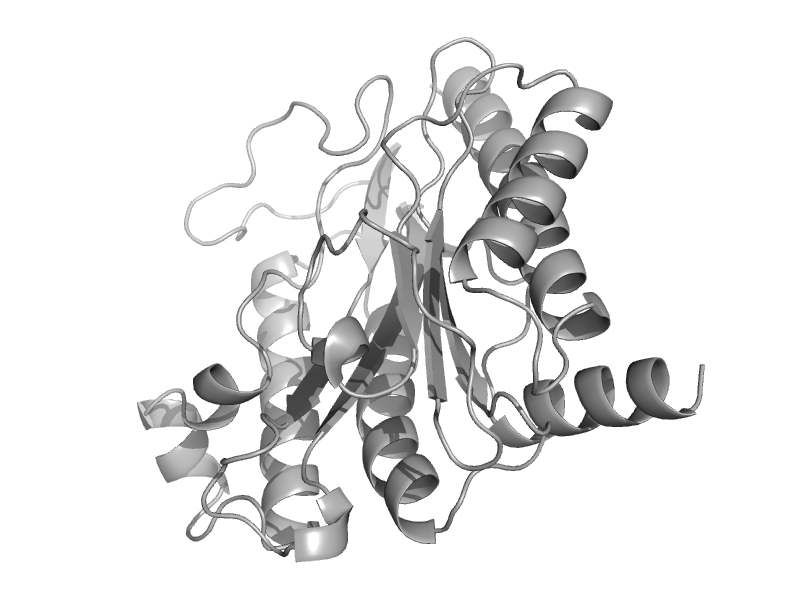 | view | |
| Methionine aminopeptidase | Creatinase/aminopeptidase | Pyrococcus furiosus [TaxId: 2261] |  | view | |
| Methionine aminopeptidase | Creatinase/aminopeptidase | Escherichia coli K-12 [TaxId: 83333] |  | view | |
| automated matches | automated matches | Mycobacterium abscessus [TaxId: 561007] |  | view | |
| automated matches | automated matches | Enterococcus faecalis [TaxId: 565646] |  | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | 230 | 240 | 250 | 260 | 270 | 280 | 290 | 300 | 310 | 320 | 330 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | I | K | S | A | E | E | H | V | M | I | R | H | G | A | R | I | A | D | I | G | G | A | A | V | V | E | A | L | G | D | Q | V | P | E | Y | E | V | A | L | H | A | T | Q | A | M | V | R | A | I | A | - | - | - | D | T | F | E | D | V | E | L | M | D | T | W | - | T | W | F | Q | S | G | I | N | T | D | G | A | - | H | N | P | V | T | T | - | - | - | R | K | V | N | K | G | D | I | L | S | L | N | C | F | P | M | I | A | G | Y | Y | T | A | L | E | R | T | L | F | L | D | - | H | C | S | D | D | H | L | R | L | W | Q | V | N | V | E | V | H | E | A | G | L | K | L | I | K | P | G | A | R | C | S | D | I | A | R | E | L | N | E | I | F | L | K | H | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | V | L | Q | Y | R | T | F | G | Y | G | H | S | F | G | T | L | S | H | Y | Y | G | R | E | A | G | L | E | L | R | E | - | - | - | - | D | I | D | T | V | L | E | P | G | M | V | V | S | M | E | P | M | I | M | L | P | E | - | - | - | - | - | - | - | - | - | G | - | - | - | - | - | L | P | G | A | G | G | Y | R | E | H | D | I | L | I | V | N | E | N | G | A | E | N | I | T | K | - | F | P | Y | G | P | E | K | N | I | I | - | R | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | T | K | E | E | I | E | I | I | E | K | A | C | E | I | A | D | K | A | V | M | A | A | I | E | E | I | T | E | G | K | R | E | R | E | V | A | A | K | V | E | Y | L | M | K | M | N | G | A | - | - | - | E | - | - | - | - | - | - | - | K | P | A | F | D | T | I | I | A | S | G | H | R | S | A | L | P | - | H | G | V | A | S | D | - | - | - | K | R | I | E | R | G | D | L | V | V | I | D | L | G | A | L | Y | N | H | Y | N | S | D | I | T | R | T | I | V | V | G | - | S | P | N | E | K | Q | R | E | I | Y | E | I | V | L | E | A | Q | K | R | A | V | E | A | A | K | P | G | M | T | A | K | E | L | D | S | I | A | R | E | I | I | K | E | Y | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | Y | G | D | Y | F | I | H | S | L | G | H | G | V | G | L | E | I | - | - | - | - | H | E | W | - | P | R | I | S | Q | - | - | - | - | Y | D | E | T | V | L | K | E | G | M | V | I | T | I | E | P | G | I | Y | I | P | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | G | G | V | R | I | E | D | T | V | L | I | T | E | N | G | A | K | R | L | T | K | - | T | E | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | I | V | K | T | E | E | E | L | Q | A | L | K | E | I | G | Y | I | C | A | K | V | R | N | T | M | Q | A | A | T | K | P | G | I | T | T | K | E | L | D | N | I | A | K | E | L | F | E | E | - | Y | G | A | I | S | A | P | I | H | D | E | N | - | - | F | P | G | Q | T | C | I | S | V | N | E | - | - | - | E | V | A | H | G | I | P | S | K | - | - | - | R | V | I | R | E | G | D | L | V | N | I | D | V | S | A | L | K | N | G | Y | Y | A | D | T | G | I | S | F | V | V | G | E | S | D | D | P | M | K | Q | K | V | C | D | V | A | T | M | A | F | E | N | A | I | A | K | V | K | P | G | T | K | L | S | N | I | G | K | A | V | H | N | T | A | R | Q | N | - | - | - | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | K | V | I | K | - | - | N | L | T | - | G | H | G | V | G | L | - | - | - | - | S | L | H | E | A | P | A | H | V | L | N | Y | F | D | P | K | D | K | T | L | L | T | E | G | M | V | L | A | I | E | P | F | I | S | S | N | A | S | F | V | T | E | G | K | N | E | W | A | F | E | T | S | - | D | K | S | F | V | A | Q | I | E | H | T | V | I | V | T | K | D | G | P | I | L | T | T | K | I | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| K | V | Q | T | D | P | P | S | V | P | I | C | D | L | Y | P | N | G | V | F | P | K | G | Q | E | C | E | Y | P | E | E | K | K | A | L | D | Q | A | S | E | E | I | W | N | D | F | R | E | A | A | E | A | H | R | Q | V | R | K | Y | V | M | S | W | I | K | P | G | M | T | M | I | E | I | C | E | K | L | E | D | C | S | R | K | L | I | K | E | N | - | - | - | - | - | G | L | N | A | G | L | A | F | P | T | G | C | S | L | N | N | - | - | - | C | A | A | H | Y | T | P | N | A | G | D | T | T | V | L | Q | Y | D | D | I | C | K | I | D | F | G | T | H | I | S | G | R | I | I | D | C | A | F | T | V | T | F | N | - | - | - | - | P | K | Y | D | T | L | L | K | A | V | K | D | A | T | N | T | G | I | K | C | A | G | I | D | V | R | L | C | D | V | G | E | A | I | Q | E | V | M | E | S | Y | - | - | - | E | V | E | I | D | G | K | - | - | - | - | - | - | T | Y | Q | V | K | P | I | R | - | - | N | L | N | - | G | H | S | I | G | Q | - | - | - | Y | R | I | H | A | G | K | - | T | V | P | I | I | K | - | G | G | E | A | T | R | M | E | E | G | E | V | Y | A | I | E | T | F | G | S | T | G | K | G | V | V | D | I | K | - | - | - | - | - | - | - | - | - | - | - | G | S | Y | T | A | Q | F | E | H | T | I | L | L | R | P | T | C | K | E | V | V | S | R | G | D | D | Y | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | P | E | E | I | A | V | L | R | R | A | G | E | I | T | A | M | A | H | T | R | A | M | E | K | C | R | P | G | M | F | E | Y | H | L | E | G | E | I | H | H | E | F | N | R | H | G | A | - | - | - | R | - | - | - | - | - | - | - | Y | P | S | Y | N | T | I | V | G | S | G | E | N | G | C | I | L | - | H | Y | T | E | N | E | - | - | - | C | E | M | R | D | G | D | L | V | L | I | D | A | G | C | E | Y | K | G | Y | A | G | D | I | T | R | T | F | P | V | N | G | K | F | T | Q | A | Q | R | E | I | Y | D | I | V | L | E | S | L | E | T | S | L | R | L | Y | R | P | G | T | S | I | L | E | V | T | G | E | V | V | R | I | M | V | S | G | L | V | K | L | G | I | L | K | G | D | V | D | E | L | I | A | Q | N | - | - | A | H | R | P | F | F | M | H | G | L | S | H | W | L | G | L | D | V | - | - | - | - | H | D | V | - | G | V | Y | G | Q | - | - | - | - | D | R | S | R | I | L | E | P | G | M | V | L | T | V | E | P | G | L | Y | I | A | P | - | - | - | - | - | - | - | - | - | D | A | E | V | P | E | Q | Y | R | G | I | G | I | R | I | E | D | D | I | V | I | T | E | T | G | N | E | N | L | T | A | S | V | V | K | K | P | E | E | I | - | - | - | E | A | L | M | V | A | A | R | K | Q |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | D | T | E | K | L | M | K | A | G | E | I | A | K | K | V | R | E | K | A | I | K | L | A | R | P | G | M | L | L | L | E | L | A | E | S | I | E | K | M | I | M | E | L | - | - | - | - | - | - | - | - | - | - | - | G | G | K | P | A | F | P | V | N | L | S | I | N | E | - | - | - | I | A | A | H | Y | T | P | Y | K | G | D | T | T | V | L | K | E | G | D | Y | L | K | I | D | V | G | V | H | I | D | G | F | I | A | D | T | A | V | T | V | R | V | G | - | - | - | - | M | E | E | D | E | L | M | E | A | A | K | E | A | L | N | A | A | I | S | V | A | R | A | G | V | E | I | K | E | L | G | K | A | I | E | N | E | I | R | K | R | - | - | - | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | F | K | P | I | V | - | - | N | L | S | - | G | H | K | I | E | R | - | - | - | Y | K | L | H | A | G | I | - | S | I | P | N | I | Y | R | P | H | D | N | Y | V | L | K | E | G | D | V | F | A | I | E | P | F | A | T | I | G | A | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | G | I | V | A | Q | F | E | H | T | I | I | V | E | K | D | S | V | I | V | T | T | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | I | S | I | K | T | P | E | D | I | E | K | M | R | V | A | G | R | L | A | A | E | V | L | E | M | I | E | P | Y | V | K | P | G | V | S | T | G | E | L | D | R | I | C | N | D | Y | I | V | N | E | Q | H | A | V | S | A | C | L | G | Y | H | G | - | - | Y | P | K | S | V | C | I | S | I | N | E | - | - | - | V | V | C | H | G | I | P | D | D | - | - | A | K | L | L | K | D | G | D | I | V | N | I | D | V | T | V | I | K | D | G | F | H | G | D | T | S | K | M | F | I | V | G | K | P | T | - | I | M | G | E | R | L | C | R | I | T | Q | E | S | L | Y | L | A | L | R | M | V | K | P | G | I | N | L | R | E | I | G | A | A | I | Q | K | F | V | E | A | E | - | - | - | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | F | S | V | V | R | - | - | E | Y | C | - | G | H | G | I | G | R | - | - | - | - | G | F | H | E | E | P | - | Q | V | L | H | Y | D | S | R | E | T | N | V | V | L | K | P | G | M | T | F | T | I | E | P | M | V | N | A | G | K | K | E | I | R | T | M | K | D | G | W | T | V | K | T | K | - | D | R | S | L | S | A | Q | Y | E | H | T | I | V | V | T | D | N | G | C | E | I | L | T | L | R | K | D | D | T | - | I | P | A | I | I | S | H | D | - | - | - | - | - | - | - | E |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | L | F | G | R | K | K | T | V | E | Q | R | T | P | G | E | L | D | A | M | A | A | A | G | S | I | V | G | A | A | L | V | A | V | R | D | A | A | K | A | G | V | S | T | L | E | L | D | Q | V | A | E | S | V | I | R | A | - | - | G | A | V | P | S | F | L | G | Y | H | G | - | - | F | P | A | S | I | C | S | S | V | N | D | - | - | - | Q | V | V | H | G | I | P | S | A | - | - | T | A | V | L | A | D | G | D | L | V | S | I | D | C | G | A | I | L | D | G | W | H | G | D | S | A | W | T | F | A | V | G | T | V | I | - | P | S | D | E | A | L | S | E | A | T | R | L | S | M | E | A | G | I | A | A | M | I | P | G | N | R | L | T | D | V | S | H | A | I | E | L | G | T | R | A | A | - | - | - | E | Q | F | D | - | - | - | - | - | - | - | - | - | - | R | A | F | G | I | V | D | - | - | G | Y | G | - | G | H | G | I | G | R | - | - | - | - | S | M | H | L | D | P | - | F | L | P | N | E | G | A | P | G | K | G | P | L | L | A | V | G | S | V | L | A | I | E | P | M | L | T | L | G | T | T | Q | T | R | V | L | A | D | D | W | T | V | V | T | T | - | D | G | S | R | A | A | H | W | E | H | T | V | A | V | T | E | A | G | P | R | I | L | T | M | R | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | I | T | L | K | S | P | R | E | I | E | M | M | D | E | S | G | E | L | L | A | D | V | H | R | H | L | R | T | F | I | K | P | G | I | T | S | W | D | I | E | V | F | V | R | D | F | I | E | S | H | - | G | G | V | A | A | Q | I | G | Y | E | G | - | - | Y | K | Y | A | T | C | C | S | I | N | D | - | - | - | E | I | C | H | G | F | P | R | K | - | - | - | K | V | L | K | D | G | D | L | I | K | V | D | M | C | V | D | L | K | G | A | I | S | D | S | C | W | S | Y | V | V | G | E | S | T | - | P | E | I | D | R | L | M | E | V | T | K | K | A | L | Y | L | G | I | E | Q | A | Q | V | G | N | R | I | G | D | I | G | H | A | I | Q | T | Y | V | E | G | E | - | - | - | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Y | G | V | V | R | - | - | D | F | V | - | G | H | G | I | G | P | - | - | - | - | T | I | H | E | S | P | - | M | I | P | H | Y | G | E | A | G | K | G | L | R | L | K | E | G | M | V | I | T | I | E | P | M | V | N | T | G | T | W | R | M | K | M | D | P | N | G | W | T | A | Y | T | E | - | D | G | G | L | S | C | Q | Y | E | H | S | L | A | I | T | K | E | G | P | R | I | L | T | S | Q | G | E | E | L | - | T | Y | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
