solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1di2a- ( 112 ) mpvgsL
d1ekza- ( 1 ) mdegdkkspisqv
d1fjge2 ( 5 )
d1kn0a- ( 25 ) cfgqcqytaeeyqaiqkalrqrLgpeyissrmagggqkvcyIeghrVinl
d1o0wa2 ( 168 ) dyktaL
d1t4oa- ( 363 ) tdkLdmnAkrqL
d1uila1 ( 8 ) leseevdlnaglhgnwtlenAkarl
d1whna1 ( 350 ) sgiikmairFdrrayppqitPkmcl
d1whqa1 ( 4 ) iknfl
d1x47a1 ( 8 ) efvinpngk---sevcil
d1x48a1 ( 8 ) yiglv
d2dixa1 ( 8 ) ktpiqvl
d2dmya1 ( 8 ) rkildskaidlmnA
d2ez6a2 ( 151 ) dyktiL
d2ljha- ( 48 ) sdpkkkmckeripqp--------knt
d5npaa- ( 8 ) igwl
d6sdya1 ( 205 ) npisrl
aaaa
60 70 80 90 100
d1di2a- ( 118 ) qelAvqkgwr--lPeytvaqes-gp-----phk----reftItCrVe---
d1ekza- ( 14 ) heiGikrn---mtvhfkvlree-gp-----ahm----knfitACivgs--
d1fjge2 ( 5 ) dfeekmilir-rtarmqa---ggrrfrfGAlVvVgdrq
d1kn0a- ( 75 ) AnemFg----ynGwahsitqqnvdfvdlnn---gkfyVGVcAfVrVqlkd
d1o0wa2 ( 174 ) qeiVqseh--kvpPeyilvrte-kn-----dgd----rifVVeVrVn-g-
d1t4oa- ( 375 ) yslIGya---slrLhyvtvkkp-t------avd----pnSiVeCrVgdg-
d1uila1 ( 33 ) nqyFqkek---iqgeYkytqvgp-d------hn----rsfiAeMtiyikq
d1whna1 ( 375 ) lewCrreklpqPvyetvqrt-----------id----rmfCSvvtva---
d1whqa1 ( 9 ) yawCgkrk---mtPayeiravgn-k------nr----qkfmCevrve---
d1x47a1 ( 23 ) heyMqrvl--kvrPvynffece--n-----pse----p-fGASvtid-g-
d1x48a1 ( 13 ) nsfAqkkkl---svnyeqcepnsel-----p------qrfiCkCkig---
d2dixa1 ( 15 ) heyGmktk---niPvyecersdv-q-----ihv----ptftfrvtvg---
d2dmya1 ( 22 ) lmrlnqirp---glqykllsqs-gp-----vha----pvftMsvdvd---
d2ez6a2 ( 157 ) qeiTqkrwke--rPeyrlisve-gp-----hhk----kkfiVeAkIk---
d2ljha- ( 66 ) vamlnelrh---gliyklesqt-gp-----vha----plftiSvevd---
d5npaa- ( 12 ) qemCmqrrwp--pPsyetetev-gl-----phe----rlftiACsil---
d6sdya1 ( 211 ) aqiqqakkek--ePeytllte-rgl-----prr----refvMqvkvg---
aaaaaa bbbbbb bbbbbbb
110 120 130 140 150
d1di2a- ( 153 ) ---tfvetG--sgt----------skqvAkrvAAekLltk---fk-----
d1ekza- ( 49 ) ----ivteg--eGn----------gkkvSkkrAAekMlv-----elqkl
d1fjge2 ( 39 ) grvGlgfGkAp-------------evplAvqkAGyyArrnmvevplqn
d1kn0a- ( 118 ) gsyhedvGyGvSeglkskalSlekArkeAvtdGlkrALrsfg-naLGnCI
d1o0wa2 ( 210 ) ---ktiatG--kgr----------tkkeAekeAAriAye-----kll
d1t4oa- ( 410 ) ---tvLgtG--vgr----------nikiAGirAAenAlrd---kkmldfy
d1uila1 ( 69 ) lgrrifAre--hgs----------nkklAAqsCAlslvrq---lyhlgvi
d1whna1 ( 407 ) ---eqkYqStlwdk----------skklAeqtAA--ivCl---rsqglpe
d1whqa1 ( 42 ) -gfnyaGmG--nst----------nkkdAqsnAArdFvny---lvrinev
d1x47a1 ( 57 ) ---vtyGsg--tas----------skklAknkAAratle-----ilipdf
d1x48a1 ( 46 ) ---qtmYgt-Gsgv----------tkqeAkqlAAkeAyqk---llksppk
d2dixa1 ( 49 ) ---ditctG--egt----------skklAkhrAAeaAini---lkana
d2dmya1 ( 56 ) ---gttyeA--sgp----------skktAklhvAvkvlqa---mg-----
d2ez6a2 ( 192 ) ---eyrtlg--egk----------skkeAeqrAAeeLikl---Le-----
d2ljha- ( 100 ) ---gqkylg--qgr----------skkvArieAAatAlrs---fi-----
d5npaa- ( 47 ) ---nyremg--kgk----------skkiAkrlAAhrMwmr---lq-----
d6sdya1 ( 246 ) ---nhtAeg--tgt----------nkkvAkrnAAenMlei---lgfk
bbbbb aaaaaaaaaaaaaa a
160 170 180 190
d1di2a- ( 180 ) ---t
d1ekza-
d1fjge2
d1kn0a- ( 167 ) ldkdylrslnklprqlplevdltkakrqdlepsveearynsc
d1o0wa2
d1t4oa- ( 442 ) ak
d1uila1 ( 104 ) e---ays
d1whna1 ( 439 ) grlgeespslnkrkreapdqdpggpr
d1whqa1 ( 76 ) kseevpavg------------ivppp
d1x47a1 ( 87 ) vk--------------------qtse
d1x48a1 ( 79 ) ta----------------------gt
d2dixa1
d2dmya1 ( 83 ) ---y-ptgfdadi
d2ez6a2 ( 219 ) ---es
d2ljha- ( 127 ) ---qfkdg-----avlsplkpa
d5npaa- ( 74 ) ---e
d6sdya1
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Double-stranded RNA-binding protein A, second dsRBD | Double-stranded RNA-binding domain (dsRBD) | African clawed frog (Xenopus laevis) [TaxId: 8355] | 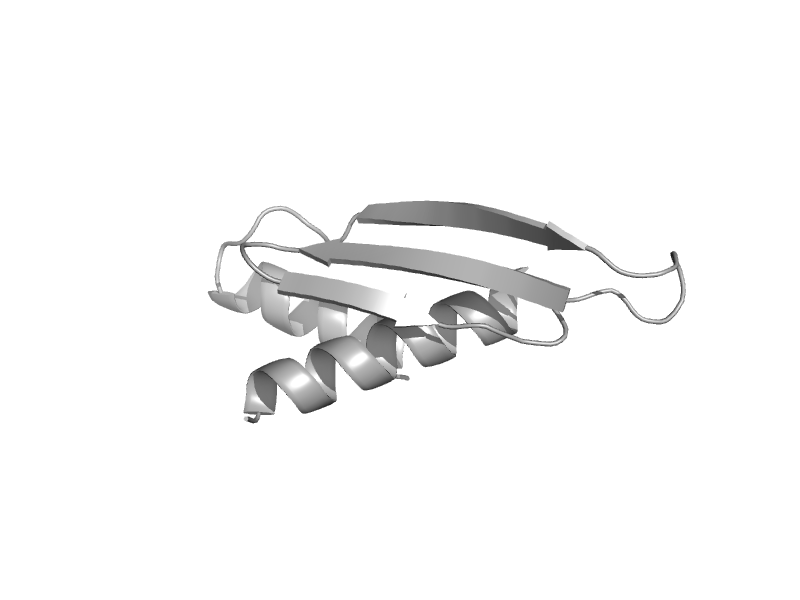 | view | |
| Staufen, domain III | Double-stranded RNA-binding domain (dsRBD) | Fruit fly (Drosophila melanogaster) [TaxId: 7227] |  | view | |
| Ribosomal S5 protein, N-terminal domain | Ribosomal S5 protein, N-terminal domain | Thermus thermophilus [TaxId: 274] |  | view | |
| The homologous-pairing domain of Rad52 recombinase | The homologous-pairing domain of Rad52 recombinase | Human (Homo sapiens) [TaxId: 9606] |  | view | |
| RNase III, C-terminal domain | Double-stranded RNA-binding domain (dsRBD) | Thermotoga maritima [TaxId: 2336] |  | view | |
| RNase III, C-terminal domain | Double-stranded RNA-binding domain (dsRBD) | Baker's yeast (Saccharomyces cerevisiae) [TaxId: 4932] |  | view | |
| ATP-dependent RNA helicase A, Dhx9 | Double-stranded RNA-binding domain (dsRBD) | Mouse (Mus musculus) [TaxId: 10090] | 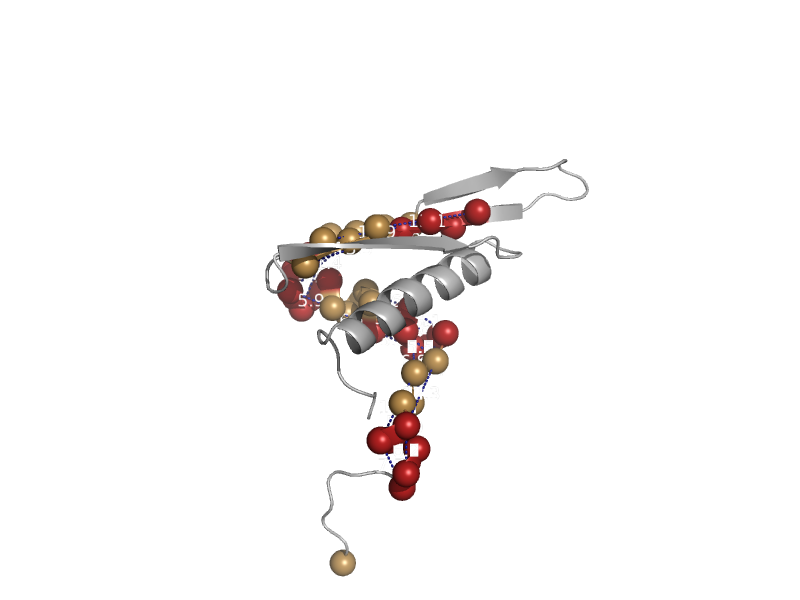 | view | |
| tRNA-dihydrouridine synthase 2-like, Dus2l (2310016k04Rik) | Double-stranded RNA-binding domain (dsRBD) | Mouse (Mus musculus) [TaxId: 10090] |  | view | |
| ATP-dependent RNA helicase A, Dhx9 | Double-stranded RNA-binding domain (dsRBD) | Mouse (Mus musculus) [TaxId: 10090] | 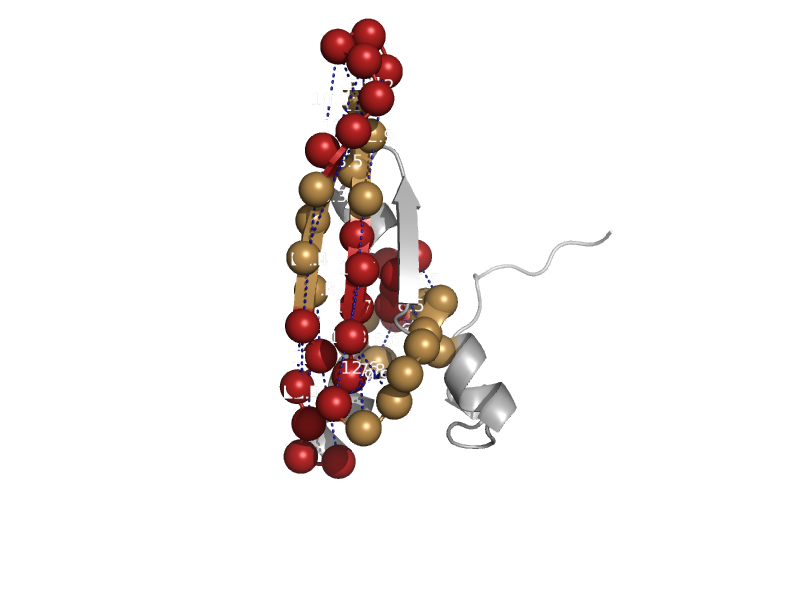 | view | |
| Dgcr8 protein | Double-stranded RNA-binding domain (dsRBD) | Human (Homo sapiens) [TaxId: 9606] | 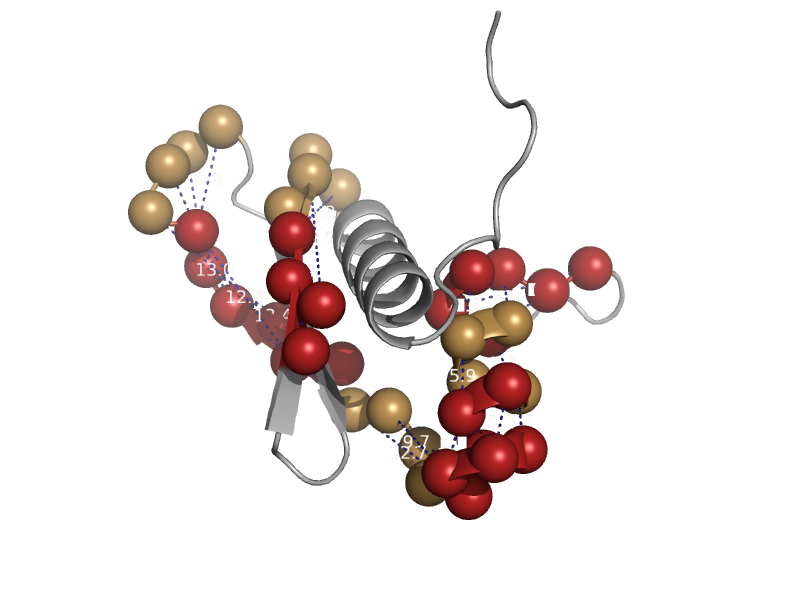 | view | |
| dsRNA-dependent protein kinase pkr | Double-stranded RNA-binding domain (dsRBD) | Mouse (Mus musculus) [TaxId: 10090] |  | view | |
| Interferon-inducible double stranded RNA-dependent protein kinase activator A | Double-stranded RNA-binding domain (dsRBD) | Human (Homo sapiens) [TaxId: 9606] |  | view | |
| Spermatid perinuclear RNA-binding protein | Double-stranded RNA-binding domain (dsRBD) | Human (Homo sapiens) [TaxId: 9606] |  | view | |
| RNase III, C-terminal domain | Double-stranded RNA-binding domain (dsRBD) | Aquifex aeolicus [TaxId: 63363] |  | view | |
| automated matches | automated matches | Fruit fly (Drosophila melanogaster) [TaxId: 7227] | 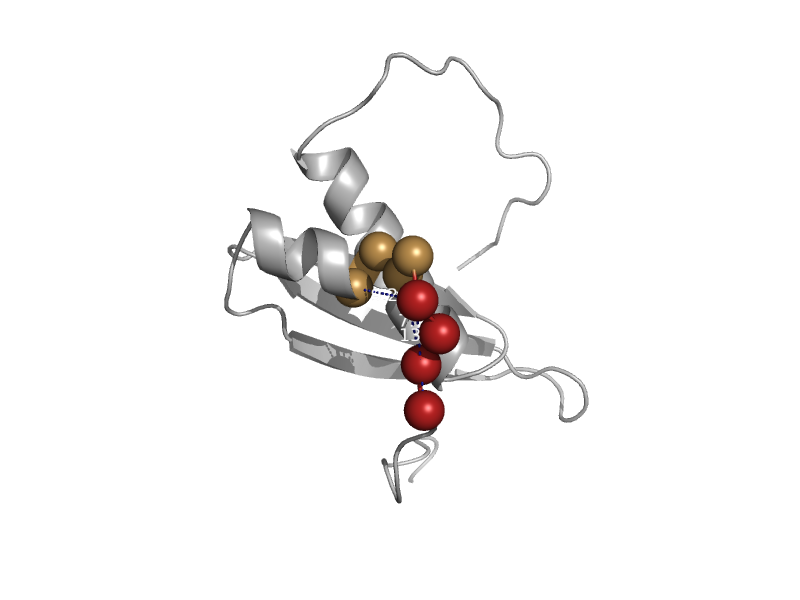 | view | |
| automated matches | automated matches | Fruit fly (Drosophila melanogaster) [TaxId: 7227] | 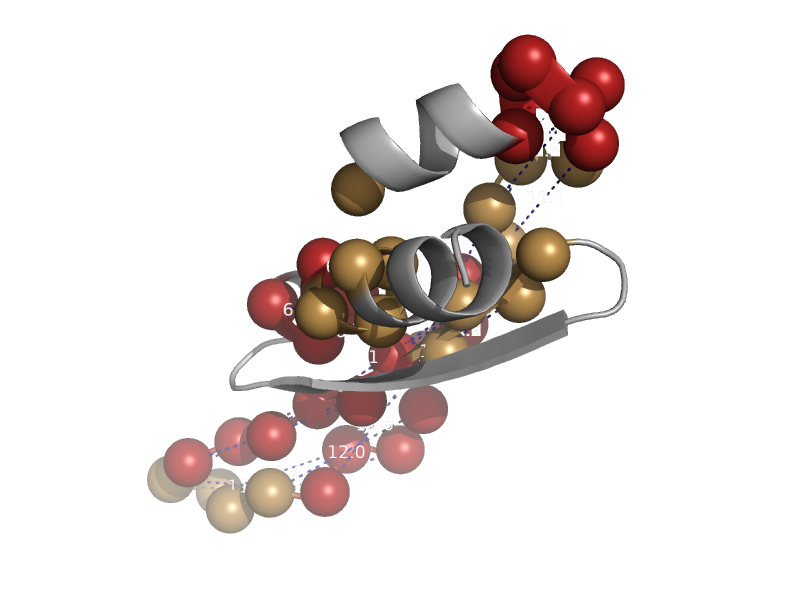 | view | |
| automated matches | automated matches | Human (Homo sapiens) [TaxId: 9606] | 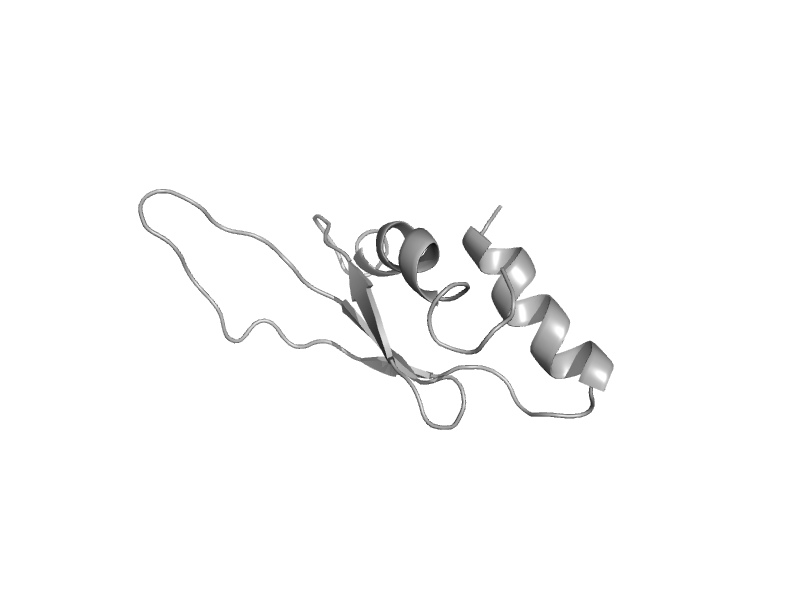 | view |
| Domain ID | Name |
|---|---|
| d1x49a1 | 1x49 A:8-91 |
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | P | V | G | S | L | Q | E | L | A | V | Q | K | G | W | R | - | - | L | P | E | Y | T | V | A | Q | E | S | - | G | P | - | - | - | - | - | P | H | K | - | - | - | - | R | E | F | T | I | T | C | R | V | E | - | - | - | - | - | - | T | F | V | E | T | G | - | - | S | G | T | - | - | - | - | - | - | - | - | - | - | S | K | Q | V | A | K | R | V | A | A | E | K | L | L | T | K | - | - | - | F | K | - | - | - | - | - | - | - | - | T | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | D | E | G | D | K | K | S | P | I | S | Q | V | H | E | I | G | I | K | R | N | - | - | - | M | T | V | H | F | K | V | L | R | E | E | - | G | P | - | - | - | - | - | A | H | M | - | - | - | - | K | N | F | I | T | A | C | I | V | G | S | - | - | - | - | - | - | I | V | T | E | G | - | - | E | G | N | - | - | - | - | - | - | - | - | - | - | G | K | K | V | S | K | K | R | A | A | E | K | M | L | V | - | - | - | - | - | E | L | Q | K | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | F | E | E | K | M | I | L | I | R | - | R | T | A | R | M | Q | A | - | - | - | G | G | R | R | F | R | F | G | A | L | V | V | V | G | D | R | Q | G | R | V | G | L | G | F | G | K | A | P | - | - | - | - | - | - | - | - | - | - | - | - | - | E | V | P | L | A | V | Q | K | A | G | Y | Y | A | R | R | N | M | V | E | V | P | L | Q | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| C | F | G | Q | C | Q | Y | T | A | E | E | Y | Q | A | I | Q | K | A | L | R | Q | R | L | G | P | E | Y | I | S | S | R | M | A | G | G | G | Q | K | V | C | Y | I | E | G | H | R | V | I | N | L | A | N | E | M | F | G | - | - | - | - | Y | N | G | W | A | H | S | I | T | Q | Q | N | V | D | F | V | D | L | N | N | - | - | - | G | K | F | Y | V | G | V | C | A | F | V | R | V | Q | L | K | D | G | S | Y | H | E | D | V | G | Y | G | V | S | E | G | L | K | S | K | A | L | S | L | E | K | A | R | K | E | A | V | T | D | G | L | K | R | A | L | R | S | F | G | - | N | A | L | G | N | C | I | L | D | K | D | Y | L | R | S | L | N | K | L | P | R | Q | L | P | L | E | V | D | L | T | K | A | K | R | Q | D | L | E | P | S | V | E | E | A | R | Y | N | S | C |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | Y | K | T | A | L | Q | E | I | V | Q | S | E | H | - | - | K | V | P | P | E | Y | I | L | V | R | T | E | - | K | N | - | - | - | - | - | D | G | D | - | - | - | - | R | I | F | V | V | E | V | R | V | N | - | G | - | - | - | - | K | T | I | A | T | G | - | - | K | G | R | - | - | - | - | - | - | - | - | - | - | T | K | K | E | A | E | K | E | A | A | R | I | A | Y | E | - | - | - | - | - | K | L | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | T | D | K | L | D | M | N | A | K | R | Q | L | Y | S | L | I | G | Y | A | - | - | - | S | L | R | L | H | Y | V | T | V | K | K | P | - | T | - | - | - | - | - | - | A | V | D | - | - | - | - | P | N | S | I | V | E | C | R | V | G | D | G | - | - | - | - | T | V | L | G | T | G | - | - | V | G | R | - | - | - | - | - | - | - | - | - | - | N | I | K | I | A | G | I | R | A | A | E | N | A | L | R | D | - | - | - | K | K | M | L | D | F | Y | A | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | E | S | E | E | V | D | L | N | A | G | L | H | G | N | W | T | L | E | N | A | K | A | R | L | N | Q | Y | F | Q | K | E | K | - | - | - | I | Q | G | E | Y | K | Y | T | Q | V | G | P | - | D | - | - | - | - | - | - | H | N | - | - | - | - | R | S | F | I | A | E | M | T | I | Y | I | K | Q | L | G | R | R | I | F | A | R | E | - | - | H | G | S | - | - | - | - | - | - | - | - | - | - | N | K | K | L | A | A | Q | S | C | A | L | S | L | V | R | Q | - | - | - | L | Y | H | L | G | V | I | E | - | - | - | A | Y | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | G | I | I | K | M | A | I | R | F | D | R | R | A | Y | P | P | Q | I | T | P | K | M | C | L | L | E | W | C | R | R | E | K | L | P | Q | P | V | Y | E | T | V | Q | R | T | - | - | - | - | - | - | - | - | - | - | - | I | D | - | - | - | - | R | M | F | C | S | V | V | T | V | A | - | - | - | - | - | - | E | Q | K | Y | Q | S | T | L | W | D | K | - | - | - | - | - | - | - | - | - | - | S | K | K | L | A | E | Q | T | A | A | - | - | I | V | C | L | - | - | - | R | S | Q | G | L | P | E | G | R | L | G | E | E | S | P | S | L | N | K | R | K | R | E | A | P | D | Q | D | P | G | G | P | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | I | K | N | F | L | Y | A | W | C | G | K | R | K | - | - | - | M | T | P | A | Y | E | I | R | A | V | G | N | - | K | - | - | - | - | - | - | N | R | - | - | - | - | Q | K | F | M | C | E | V | R | V | E | - | - | - | - | G | F | N | Y | A | G | M | G | - | - | N | S | T | - | - | - | - | - | - | - | - | - | - | N | K | K | D | A | Q | S | N | A | A | R | D | F | V | N | Y | - | - | - | L | V | R | I | N | E | V | K | S | E | E | V | P | A | V | G | - | - | - | - | - | - | - | - | - | - | - | - | I | V | P | P | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | F | V | I | N | P | N | G | K | - | - | - | S | E | V | C | I | L | H | E | Y | M | Q | R | V | L | - | - | K | V | R | P | V | Y | N | F | F | E | C | E | - | - | N | - | - | - | - | - | P | S | E | - | - | - | - | P | - | F | G | A | S | V | T | I | D | - | G | - | - | - | - | V | T | Y | G | S | G | - | - | T | A | S | - | - | - | - | - | - | - | - | - | - | S | K | K | L | A | K | N | K | A | A | R | A | T | L | E | - | - | - | - | - | I | L | I | P | D | F | V | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | T | S | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Y | I | G | L | V | N | S | F | A | Q | K | K | K | L | - | - | - | S | V | N | Y | E | Q | C | E | P | N | S | E | L | - | - | - | - | - | P | - | - | - | - | - | - | Q | R | F | I | C | K | C | K | I | G | - | - | - | - | - | - | Q | T | M | Y | G | T | - | G | S | G | V | - | - | - | - | - | - | - | - | - | - | T | K | Q | E | A | K | Q | L | A | A | K | E | A | Y | Q | K | - | - | - | L | L | K | S | P | P | K | T | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | T | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | T | P | I | Q | V | L | H | E | Y | G | M | K | T | K | - | - | - | N | I | P | V | Y | E | C | E | R | S | D | V | - | Q | - | - | - | - | - | I | H | V | - | - | - | - | P | T | F | T | F | R | V | T | V | G | - | - | - | - | - | - | D | I | T | C | T | G | - | - | E | G | T | - | - | - | - | - | - | - | - | - | - | S | K | K | L | A | K | H | R | A | A | E | A | A | I | N | I | - | - | - | L | K | A | N | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | K | I | L | D | S | K | A | I | D | L | M | N | A | L | M | R | L | N | Q | I | R | P | - | - | - | G | L | Q | Y | K | L | L | S | Q | S | - | G | P | - | - | - | - | - | V | H | A | - | - | - | - | P | V | F | T | M | S | V | D | V | D | - | - | - | - | - | - | G | T | T | Y | E | A | - | - | S | G | P | - | - | - | - | - | - | - | - | - | - | S | K | K | T | A | K | L | H | V | A | V | K | V | L | Q | A | - | - | - | M | G | - | - | - | - | - | - | - | - | Y | - | P | T | G | F | D | A | D | I | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | Y | K | T | I | L | Q | E | I | T | Q | K | R | W | K | E | - | - | R | P | E | Y | R | L | I | S | V | E | - | G | P | - | - | - | - | - | H | H | K | - | - | - | - | K | K | F | I | V | E | A | K | I | K | - | - | - | - | - | - | E | Y | R | T | L | G | - | - | E | G | K | - | - | - | - | - | - | - | - | - | - | S | K | K | E | A | E | Q | R | A | A | E | E | L | I | K | L | - | - | - | L | E | - | - | - | - | - | - | - | - | E | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | D | P | K | K | K | M | C | K | E | R | I | P | Q | P | - | - | - | - | - | - | - | - | K | N | T | V | A | M | L | N | E | L | R | H | - | - | - | G | L | I | Y | K | L | E | S | Q | T | - | G | P | - | - | - | - | - | V | H | A | - | - | - | - | P | L | F | T | I | S | V | E | V | D | - | - | - | - | - | - | G | Q | K | Y | L | G | - | - | Q | G | R | - | - | - | - | - | - | - | - | - | - | S | K | K | V | A | R | I | E | A | A | A | T | A | L | R | S | - | - | - | F | I | - | - | - | - | - | - | - | - | Q | F | K | D | G | - | - | - | - | - | A | V | L | S | P | L | K | P | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | I | G | W | L | Q | E | M | C | M | Q | R | R | W | P | - | - | P | P | S | Y | E | T | E | T | E | V | - | G | L | - | - | - | - | - | P | H | E | - | - | - | - | R | L | F | T | I | A | C | S | I | L | - | - | - | - | - | - | N | Y | R | E | M | G | - | - | K | G | K | - | - | - | - | - | - | - | - | - | - | S | K | K | I | A | K | R | L | A | A | H | R | M | W | M | R | - | - | - | L | Q | - | - | - | - | - | - | - | - | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | P | I | S | R | L | A | Q | I | Q | Q | A | K | K | E | K | - | - | E | P | E | Y | T | L | L | T | E | - | R | G | L | - | - | - | - | - | P | R | R | - | - | - | - | R | E | F | V | M | Q | V | K | V | G | - | - | - | - | - | - | N | H | T | A | E | G | - | - | T | G | T | - | - | - | - | - | - | - | - | - | - | N | K | K | V | A | K | R | N | A | A | E | N | M | L | E | I | - | - | - | L | G | F | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
