solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1kxpd1 ( 17 ) lergrdyeknkvçkefshlgkedFtslSLVlySrkfpsGtfeqVsqlVke
d1kxpd2 ( 215 ) lsnrvçsqyaaygekkSrlSnLIklAqkVPtAdLedVlpLAed
d1kxpd3 ( 405 ) elçadysentfteYkkklaerlkaklpdatpkelaklvnk
d6hn1a1 ( 1 ) dthkSeIAhrfndlgeenFqGlVlIaFSqyLQqCpfdeHvkLvke
d6hn1a2 ( 196 ) r--lrçasiqkfgeraLkawSVArLSQKFPkAdftdVtkIvtd
d6hn1a3 ( 388 ) k--knçelfekhgeygFQnaLIvryTrKaPqVstptLveISrs
aaaaaaaaa aaaaaaaaaaaaaaa aaaaaaaaaa
60 70 80 90 100
d1kxpd1 ( 67 ) vvsLTeaÇÇa----egad---pdçydtrts---alsak--sces--nSpF
d1kxpd2 ( 258 ) iTnILskÇçesase----dçmakeLpeHTvklçdnLst--knskFedÇÇq
d1kxpd3 ( 445 ) rSdfAsnÇÇsi---nspplyçdseidaelkni
d6hn1a1 ( 46 ) LTefAktÇvadeshagÇdkslhtlfGdeLçkv-atLre--tygdMAdÇçe
d6hn1a2 ( 237 ) lTkVhkeçÇh-----gdlleÇaddradlAkyIçdhqdt--LSskLkeÇÇd
d6hn1a3 ( 429 ) LGkvGtkÇÇakpes-ermpçtedylsliLnrLÇvlhektpvSekVtkÇçt
aaaaaaaa aaaaaaaaa
110 120 130 140 150
d1kxpd1 ( 103 ) P-vhp------lkhqpqe-----fptyv---eptndeIçeafrkdpkeya
d1kxpd2 ( 302 ) ektamdvfvçtyfMpaa--qlpe-lpdv--elptnkdVçdp---gntkvm
d1kxpd3
d6hn1a1 ( 93 ) k-qepeRneçFlkhkddsP----dLpkl---kpepdtlÇaeFkadekkFw
d6hn1a2 ( 280 ) k-pvlekShçiaeidkd--avpenlppltadFAedkeVçknYqeakdvfl
d6hn1a3 ( 478 ) e-slvnrrpçFsdltlDetyvpk--pfdgesftfhadIçtlpdtekqikk
aaaa
160 170 180 190 200
d1kxpd1 ( 157 ) nqFmweYSTnygqaplslLvsyTksYlsMVgsÇÇtsasptvçFlkerlql
d1kxpd2 ( 344 ) dkYTFelSrrt-hLpevfLSkvLeptlksLgeÇçdvedsttçFnakgpll
d1kxpd3
d6hn1a1 ( 135 ) gkyLYevArrhPyFyapeLlyyAnkyngvFqeÇÇqaedkgaçLlpkietm
d6hn1a2 ( 327 ) gsFLYeYSrrhPeyavsvLlrLakeYeatLedÇÇakedphaçYatVfdkL
d6hn1a3 ( 525 ) QtalVelLkhkpkAtdeqLktVmenFvafvdkÇçaaddkegçFlleGpkl
aaaaaaaa aaaaaaaaaaaaaaaa aaaaa aaaaa
210
d1kxpd1 ( 207 ) khlsllTt
d1kxpd2 ( 393 ) kkelssfidkGq
d1kxpd3
d6hn1a1 ( 185 ) rekvlassarq
d6hn1a2 ( 377 ) khlvdepqnl-i
d6hn1a3 ( 575 ) vastqaaLa
aaaaa
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Vitamin D binding protein | Serum albumin-like | Human (Homo sapiens) [TaxId: 9606] |  | view | |
| Vitamin D binding protein | Serum albumin-like | Human (Homo sapiens) [TaxId: 9606] |  | view | |
| Vitamin D binding protein | Serum albumin-like | Human (Homo sapiens) [TaxId: 9606] |  | view | |
| automated matches | automated matches | Goat (Capra hircus) [TaxId: 9925] |  | view | |
| automated matches | automated matches | Goat (Capra hircus) [TaxId: 9925] | 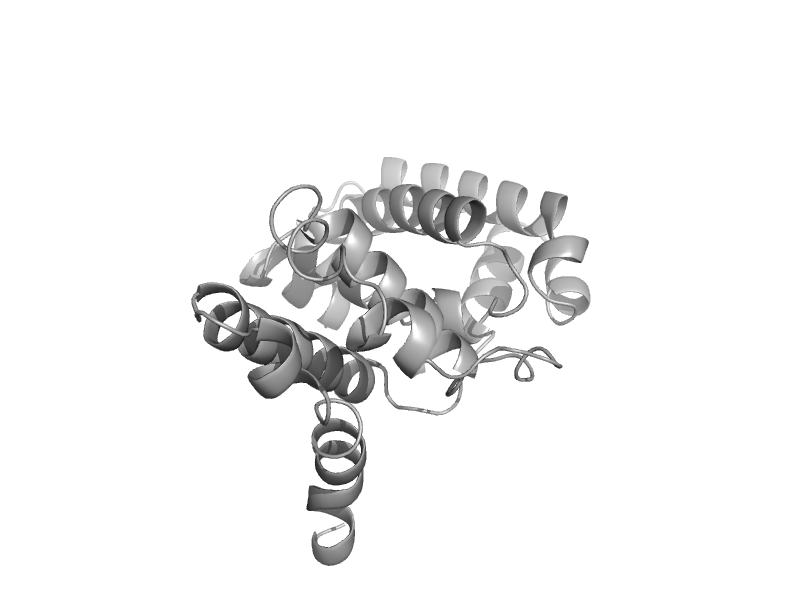 | view | |
| automated matches | automated matches | Goat (Capra hircus) [TaxId: 9925] | 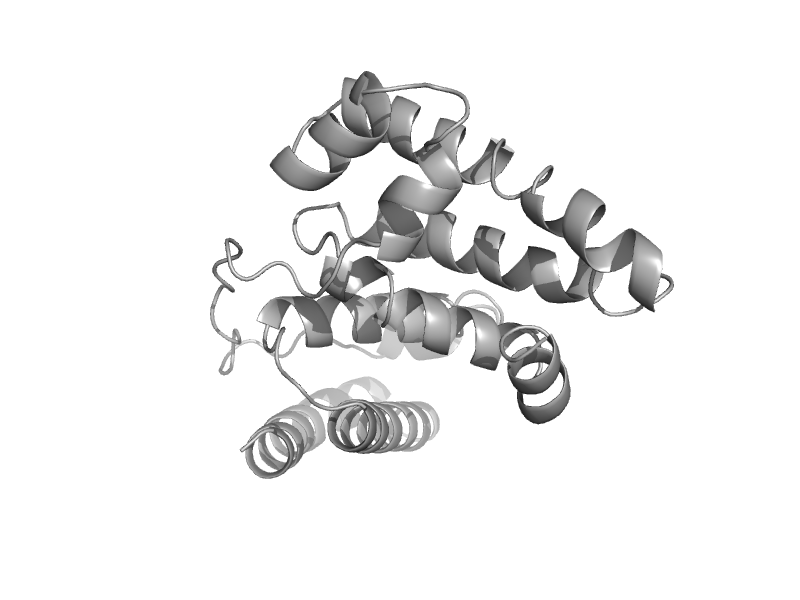 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| L | E | R | G | R | D | Y | E | K | N | K | V | C | K | E | F | S | H | L | G | K | E | D | F | T | S | L | S | L | V | L | Y | S | R | K | F | P | S | G | T | F | E | Q | V | S | Q | L | V | K | E | V | V | S | L | T | E | A | C | C | A | - | - | - | - | E | G | A | D | - | - | - | P | D | C | Y | D | T | R | T | S | - | - | - | A | L | S | A | K | - | - | S | C | E | S | - | - | N | S | P | F | P | - | V | H | P | - | - | - | - | - | - | L | K | H | Q | P | Q | E | - | - | - | - | - | F | P | T | Y | V | - | - | - | E | P | T | N | D | E | I | C | E | A | F | R | K | D | P | K | E | Y | A | N | Q | F | M | W | E | Y | S | T | N | Y | G | Q | A | P | L | S | L | L | V | S | Y | T | K | S | Y | L | S | M | V | G | S | C | C | T | S | A | S | P | T | V | C | F | L | K | E | R | L | Q | L | K | H | L | S | L | L | T | T | - | - | - | - |
| - | - | - | - | - | - | - | L | S | N | R | V | C | S | Q | Y | A | A | Y | G | E | K | K | S | R | L | S | N | L | I | K | L | A | Q | K | V | P | T | A | D | L | E | D | V | L | P | L | A | E | D | I | T | N | I | L | S | K | C | C | E | S | A | S | E | - | - | - | - | D | C | M | A | K | E | L | P | E | H | T | V | K | L | C | D | N | L | S | T | - | - | K | N | S | K | F | E | D | C | C | Q | E | K | T | A | M | D | V | F | V | C | T | Y | F | M | P | A | A | - | - | Q | L | P | E | - | L | P | D | V | - | - | E | L | P | T | N | K | D | V | C | D | P | - | - | - | G | N | T | K | V | M | D | K | Y | T | F | E | L | S | R | R | T | - | H | L | P | E | V | F | L | S | K | V | L | E | P | T | L | K | S | L | G | E | C | C | D | V | E | D | S | T | T | C | F | N | A | K | G | P | L | L | K | K | E | L | S | S | F | I | D | K | G | Q |
| - | - | - | - | - | - | - | - | - | - | E | L | C | A | D | Y | S | E | N | T | F | T | E | Y | K | K | K | L | A | E | R | L | K | A | K | L | P | D | A | T | P | K | E | L | A | K | L | V | N | K | R | S | D | F | A | S | N | C | C | S | I | - | - | - | N | S | P | P | L | Y | C | D | S | E | I | D | A | E | L | K | N | I | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | D | T | H | K | S | E | I | A | H | R | F | N | D | L | G | E | E | N | F | Q | G | L | V | L | I | A | F | S | Q | Y | L | Q | Q | C | P | F | D | E | H | V | K | L | V | K | E | L | T | E | F | A | K | T | C | V | A | D | E | S | H | A | G | C | D | K | S | L | H | T | L | F | G | D | E | L | C | K | V | - | A | T | L | R | E | - | - | T | Y | G | D | M | A | D | C | C | E | K | - | Q | E | P | E | R | N | E | C | F | L | K | H | K | D | D | S | P | - | - | - | - | D | L | P | K | L | - | - | - | K | P | E | P | D | T | L | C | A | E | F | K | A | D | E | K | K | F | W | G | K | Y | L | Y | E | V | A | R | R | H | P | Y | F | Y | A | P | E | L | L | Y | Y | A | N | K | Y | N | G | V | F | Q | E | C | C | Q | A | E | D | K | G | A | C | L | L | P | K | I | E | T | M | R | E | K | V | L | A | S | S | A | R | Q | - |
| - | - | - | - | - | - | - | R | - | - | L | R | C | A | S | I | Q | K | F | G | E | R | A | L | K | A | W | S | V | A | R | L | S | Q | K | F | P | K | A | D | F | T | D | V | T | K | I | V | T | D | L | T | K | V | H | K | E | C | C | H | - | - | - | - | - | G | D | L | L | E | C | A | D | D | R | A | D | L | A | K | Y | I | C | D | H | Q | D | T | - | - | L | S | S | K | L | K | E | C | C | D | K | - | P | V | L | E | K | S | H | C | I | A | E | I | D | K | D | - | - | A | V | P | E | N | L | P | P | L | T | A | D | F | A | E | D | K | E | V | C | K | N | Y | Q | E | A | K | D | V | F | L | G | S | F | L | Y | E | Y | S | R | R | H | P | E | Y | A | V | S | V | L | L | R | L | A | K | E | Y | E | A | T | L | E | D | C | C | A | K | E | D | P | H | A | C | Y | A | T | V | F | D | K | L | K | H | L | V | D | E | P | Q | N | L | - | I |
| - | - | - | - | - | - | - | K | - | - | K | N | C | E | L | F | E | K | H | G | E | Y | G | F | Q | N | A | L | I | V | R | Y | T | R | K | A | P | Q | V | S | T | P | T | L | V | E | I | S | R | S | L | G | K | V | G | T | K | C | C | A | K | P | E | S | - | E | R | M | P | C | T | E | D | Y | L | S | L | I | L | N | R | L | C | V | L | H | E | K | T | P | V | S | E | K | V | T | K | C | C | T | E | - | S | L | V | N | R | R | P | C | F | S | D | L | T | L | D | E | T | Y | V | P | K | - | - | P | F | D | G | E | S | F | T | F | H | A | D | I | C | T | L | P | D | T | E | K | Q | I | K | K | Q | T | A | L | V | E | L | L | K | H | K | P | K | A | T | D | E | Q | L | K | T | V | M | E | N | F | V | A | F | V | D | K | C | C | A | A | D | D | K | E | G | C | F | L | L | E | G | P | K | L | V | A | S | T | Q | A | A | L | A | - | - | - |
