solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1irxa1 ( 320 ) glgilnlydefEkveriyfgvegeelrrtYelsmpkkperlvAQApFrFL
d1j09a1 ( 306 ) dleklrwmnGkyIr-evlsleeVaerVkpfLreagLswe-----
d2cfoa2 ( 312 ) dwdklnwlnrqyIq--qlepeeFLaeLiplWqgagyaFd--e--
d2o5ra2 ( 313 ) dp-klkw-nGyylr-n--pieklaelAkpfFekagIkIi-----
d3afha2 ( 316 ) dyqklewvnGkhMr--ridledLKreFiewAkyagkeIp--s--
d4g6za2 ( 305 ) dhnklnwlnnhyIk--eaddarLaglAkpfFaalgId----a--
d4gria2 ( 315 ) dyhkldffnsyyir--ekkdedLfnlLlpfFqk-gyVskpst--
d6b1pa2 ( 300 ) swlnwlnahylqs--------------------------aLl
d7k86a2 ( 299 ) dm-algwvnqhfLk--tedvaaIvphLvyqLqklgldva--a--
aaaaaaaa aaaaaaaaaaaa
60 70 80 90 100
d1irxa1 ( 375 ) avLVqLphlteedIInvLikqghIprdlskedverVklRInlArnWvkky
d1j09a1 ( 344 ) ---------seayLrrAVeLMrp--------rFdtlkeFPekArYLFte-
d2cfoa2 ( 350 ) -------erdrpWLfdLAqlLqp--------gLntlreAidqGavFFip-
d2o5ra2 ( 350 ) ---------deeyFkkVLeiTke--------rVevlseFPeeSrFFfe--
d3afha2 ( 354 ) --------vderyFseTLrICre--------kvntlsqLydiMyPFMnd-
d4g6za2 ( 341 ) -------gaigpdLvsVMglmk---------dAstvkeIAenSAmFYra-
d4gria2 ( 355 ) -------leenqkLklLIplIks--------rI-klsdAlnmTkfFye--
d6b1pa2 ( 322 ) elLpfSf-sdlpalldalk------------eSqtlkeLAlkIdeVliap
d7k86a2 ( 337 ) -------gpa---pedVVvALre--------rVqtlkeMAekAvvWYqp-
aaaaaaaa aaaaa
110 120 130 140 150
d1irxa1 ( 425 ) ApedvkFsilekppeveVsedVreAMneVAewLenheefsveeFnniLfe
d1j09a1 ( 376 ) ----dYpvsekAqrklee---glplLkeLyprLraqeewteaaLealLrg
d2cfoa2 ( 384 ) ----svtfdseAma-qLgqpqSatILayLlehLpaepaLtvamGqqlIqq
d2o5ra2 ( 381 ) ----dpapv-------eIpee-keVfsqL-eeLqnv-rwt-eeItpvFkk
d3afha2 ( 387 ) ----dYeYekdYvekfLkreeAerVLeeAkkaFkdlnswnmeeIekTLrd
d4g6za2 ( 377 ) ----patps-----------------------------------------
d4gria2 ( 388 ) ------dIwnefe----------cilelkp----------------grse
d6b1pa2 ( 371 ) yeefn-------------------almplLefa---------------ee
d7k86a2 ( 368 ) ----lteydeaAvakhF-kagaevaLgkArelLaalp-wtaesVgvALhd
160 170 180 190 200
d1irxa1 ( 475 ) VakrrgIssreWfstLyrLFIgkergprLAsFLASLd--------rsfVi
d1j09a1 ( 419 ) FAaekgvklgqVaqPLrAALTGsleTpgLfeILalLgk-erALrRLeral
d2cfoa2 ( 429 ) AAkaAgvkkgaTmrTLrAALTGaVhgpdLmaAWqILHqrgwDepRLaaAl
d2o5ra2 ( 419 ) VLkqhgvkpkeFy-tLrrVLTgreeGpelvnIiplLgk-eiFlrRierSl
d3afha2 ( 433 ) LsekglaskkvVfqLIrGAVTGklvTPgLfeTIevLgk-erTlkRLerTL
d4g6za2 ( 449 ) -----------------------------davlllfgvvsra
d4gria2 ( 429 ) nda---------glpiriAALGs-vsPpLfdSlklIg------skvferi
d6b1pa2 ( 419 ) ia-------gsfmqpLrlALlgg--ggigleAlfILg--------kesRi
d7k86a2 ( 413 ) AaaaleigmgkVaqPLrVAITGtqvspdishTVyLAgr-eqAl-RIdvAi
aaa aaaaa aaaaaaa aaaa
d1irxa1 ( 517 ) kRLrleg
d1j09a1 ( 468 ) a
d2cfoa2 ( 479 ) kqAqtts
d2o5ra2 ( 468 ) g
d3afha2 ( 482 ) qflkk
d4g6za2
d4gria2 ( 483 ) Aqfrn
d6b1pa2 ( 459 ) nFl
d7k86a2 ( 462 ) tkVa
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| C-terminal domain of class I lysyl-tRNA synthetase | C-terminal domain of class I lysyl-tRNA synthetase | Pyrococcus horikoshii [TaxId: 53953] | 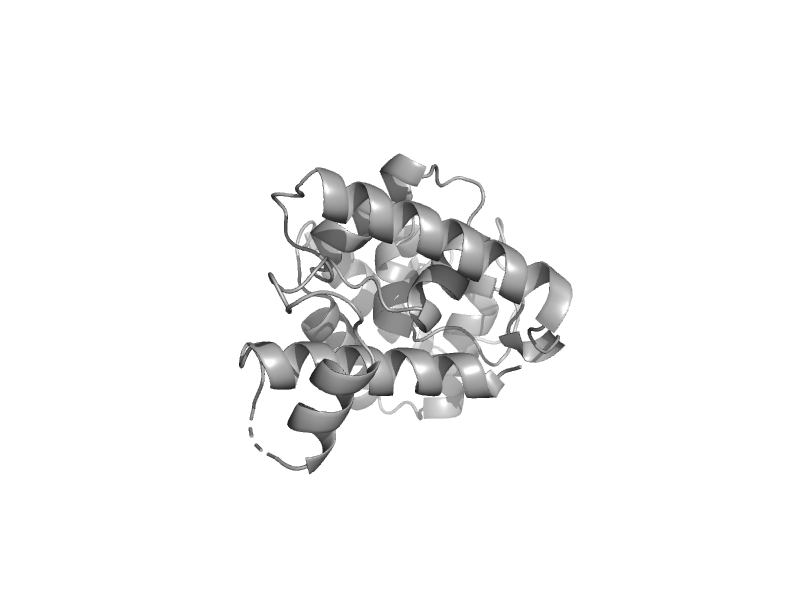 | view | |
| C-terminal domain of glutamyl-tRNA synthetase (GluRS) | C-terminal domain of glutamyl-tRNA synthetase (GluRS) | Thermus thermophilus [TaxId: 274] | 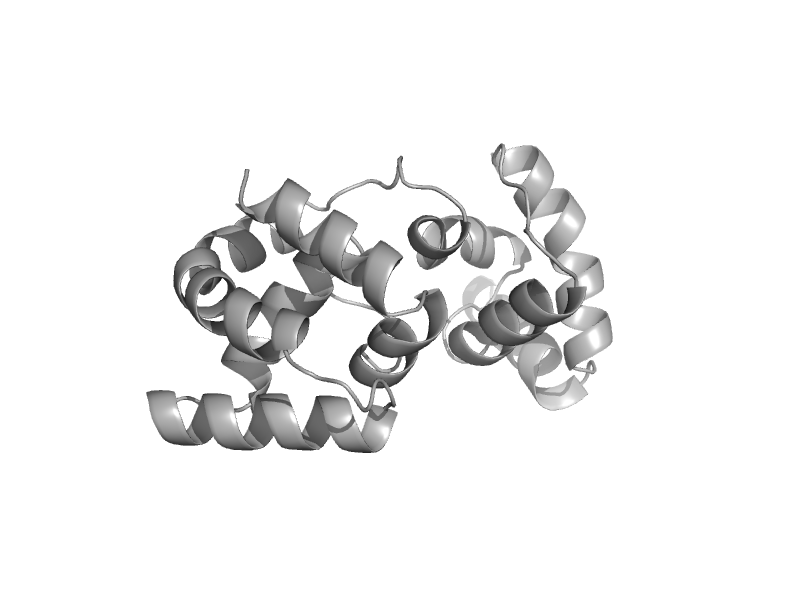 | view | |
| automated matches | automated matches | Synechococcus elongatus [TaxId: 32046] | 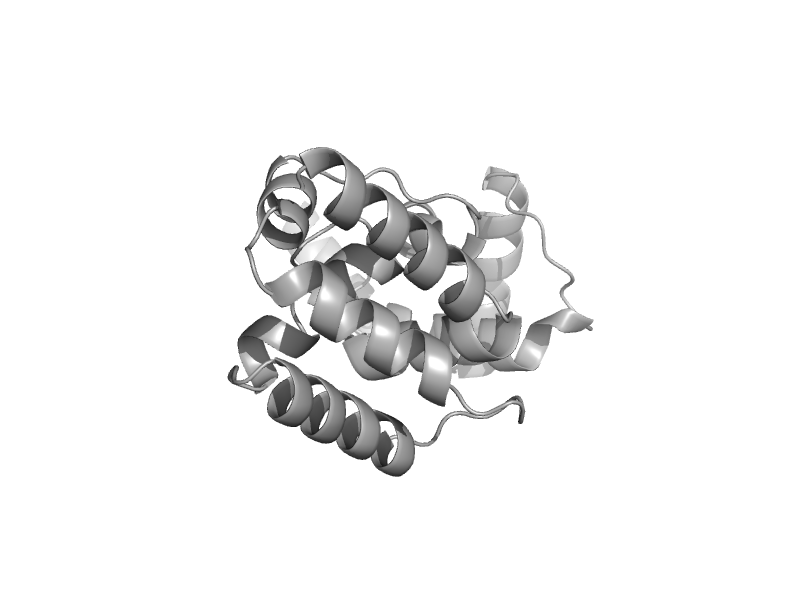 | view | |
| automated matches | automated matches | Thermotoga maritima [TaxId: 2336] | 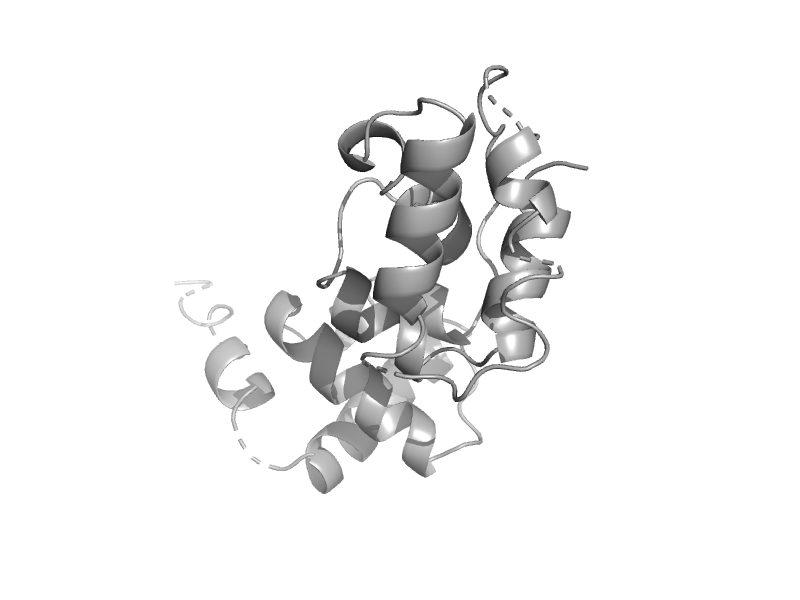 | view | |
| automated matches | automated matches | Thermotoga maritima [TaxId: 2336] | 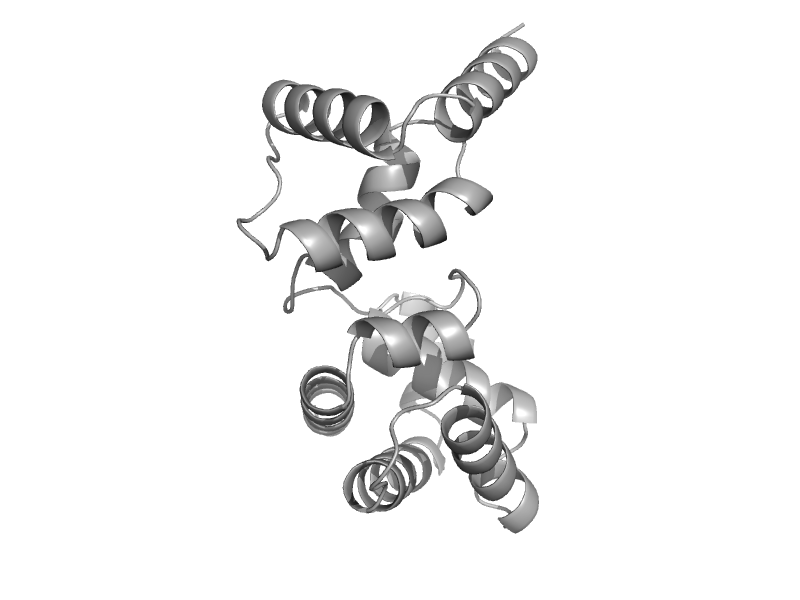 | view | |
| automated matches | automated matches | Burkholderia thailandensis [TaxId: 271848] | 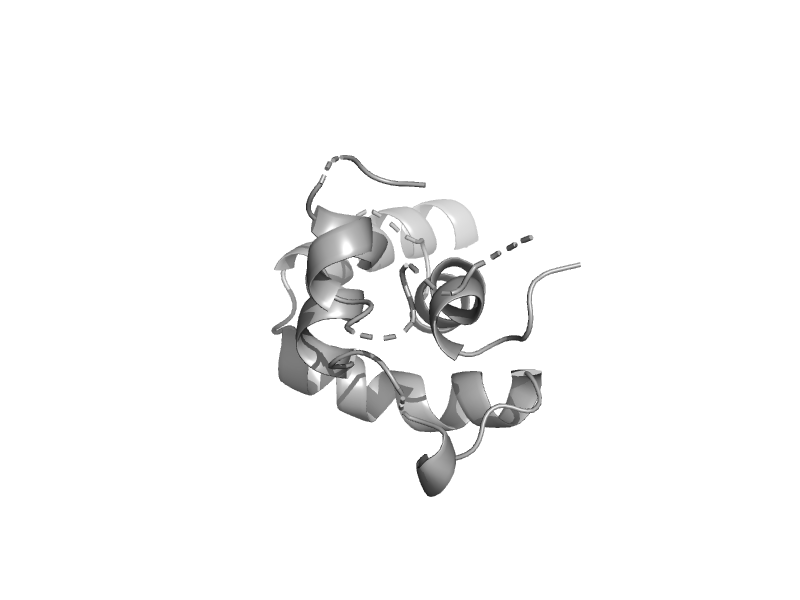 | view | |
| automated matches | automated matches | Borrelia burgdorferi [TaxId: 224326] | 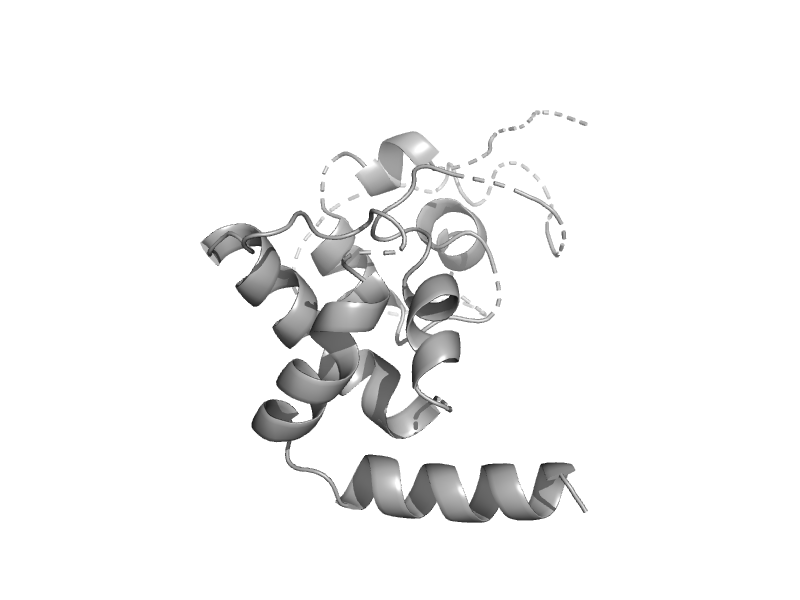 | view | |
| automated matches | automated matches | Helicobacter pylori [TaxId: 563041] | 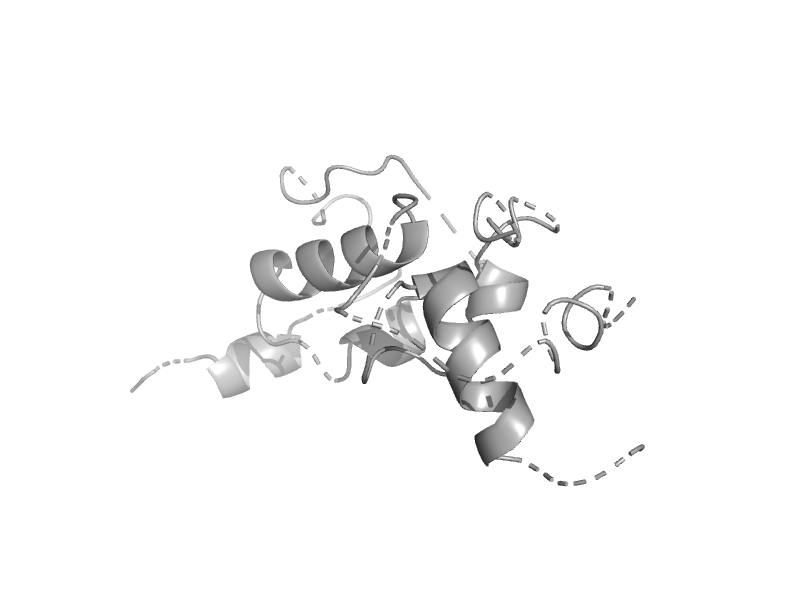 | view | |
| automated matches | automated matches | Stenotrophomonas maltophilia [TaxId: 522373] |  | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| G | L | G | I | L | N | L | Y | D | E | F | E | K | V | E | R | I | Y | F | G | V | E | G | E | E | L | R | R | T | Y | E | L | S | M | P | K | K | P | E | R | L | V | A | Q | A | P | F | R | F | L | A | V | L | V | Q | L | P | H | L | T | E | E | D | I | I | N | V | L | I | K | Q | G | H | I | P | R | D | L | S | K | E | D | V | E | R | V | K | L | R | I | N | L | A | R | N | W | V | K | K | Y | A | P | E | D | V | K | F | S | I | L | E | K | P | P | E | V | E | V | S | E | D | V | R | E | A | M | N | E | V | A | E | W | L | E | N | H | E | E | F | S | V | E | E | F | N | N | I | L | F | E | V | A | K | R | R | G | I | S | S | R | E | W | F | S | T | L | Y | R | L | F | I | G | K | E | R | G | P | R | L | A | S | F | L | A | S | L | D | - | - | - | - | - | - | - | - | R | S | F | V | I | K | R | L | R | L | E | G |
| - | - | - | - | - | - | D | L | E | K | L | R | W | M | N | G | K | Y | I | R | - | E | V | L | S | L | E | E | V | A | E | R | V | K | P | F | L | R | E | A | G | L | S | W | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | E | A | Y | L | R | R | A | V | E | L | M | R | P | - | - | - | - | - | - | - | - | R | F | D | T | L | K | E | F | P | E | K | A | R | Y | L | F | T | E | - | - | - | - | - | D | Y | P | V | S | E | K | A | Q | R | K | L | E | E | - | - | - | G | L | P | L | L | K | E | L | Y | P | R | L | R | A | Q | E | E | W | T | E | A | A | L | E | A | L | L | R | G | F | A | A | E | K | G | V | K | L | G | Q | V | A | Q | P | L | R | A | A | L | T | G | S | L | E | T | P | G | L | F | E | I | L | A | L | L | G | K | - | E | R | A | L | R | R | L | E | R | A | L | A | - | - | - | - | - | - |
| - | - | - | - | - | - | D | W | D | K | L | N | W | L | N | R | Q | Y | I | Q | - | - | Q | L | E | P | E | E | F | L | A | E | L | I | P | L | W | Q | G | A | G | Y | A | F | D | - | - | E | - | - | - | - | - | - | - | - | - | E | R | D | R | P | W | L | F | D | L | A | Q | L | L | Q | P | - | - | - | - | - | - | - | - | G | L | N | T | L | R | E | A | I | D | Q | G | A | V | F | F | I | P | - | - | - | - | - | S | V | T | F | D | S | E | A | M | A | - | Q | L | G | Q | P | Q | S | A | T | I | L | A | Y | L | L | E | H | L | P | A | E | P | A | L | T | V | A | M | G | Q | Q | L | I | Q | Q | A | A | K | A | A | G | V | K | K | G | A | T | M | R | T | L | R | A | A | L | T | G | A | V | H | G | P | D | L | M | A | A | W | Q | I | L | H | Q | R | G | W | D | E | P | R | L | A | A | A | L | K | Q | A | Q | T | T | S |
| - | - | - | - | - | - | D | P | - | K | L | K | W | - | N | G | Y | Y | L | R | - | N | - | - | P | I | E | K | L | A | E | L | A | K | P | F | F | E | K | A | G | I | K | I | I | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | E | E | Y | F | K | K | V | L | E | I | T | K | E | - | - | - | - | - | - | - | - | R | V | E | V | L | S | E | F | P | E | E | S | R | F | F | F | E | - | - | - | - | - | - | D | P | A | P | V | - | - | - | - | - | - | - | E | I | P | E | E | - | K | E | V | F | S | Q | L | - | E | E | L | Q | N | V | - | R | W | T | - | E | E | I | T | P | V | F | K | K | V | L | K | Q | H | G | V | K | P | K | E | F | Y | - | T | L | R | R | V | L | T | G | R | E | E | G | P | E | L | V | N | I | I | P | L | L | G | K | - | E | I | F | L | R | R | I | E | R | S | L | G | - | - | - | - | - | - |
| - | - | - | - | - | - | D | Y | Q | K | L | E | W | V | N | G | K | H | M | R | - | - | R | I | D | L | E | D | L | K | R | E | F | I | E | W | A | K | Y | A | G | K | E | I | P | - | - | S | - | - | - | - | - | - | - | - | - | - | V | D | E | R | Y | F | S | E | T | L | R | I | C | R | E | - | - | - | - | - | - | - | - | K | V | N | T | L | S | Q | L | Y | D | I | M | Y | P | F | M | N | D | - | - | - | - | - | D | Y | E | Y | E | K | D | Y | V | E | K | F | L | K | R | E | E | A | E | R | V | L | E | E | A | K | K | A | F | K | D | L | N | S | W | N | M | E | E | I | E | K | T | L | R | D | L | S | E | K | G | L | A | S | K | K | V | V | F | Q | L | I | R | G | A | V | T | G | K | L | V | T | P | G | L | F | E | T | I | E | V | L | G | K | - | E | R | T | L | K | R | L | E | R | T | L | Q | F | L | K | K | - | - |
| - | - | - | - | - | - | D | H | N | K | L | N | W | L | N | N | H | Y | I | K | - | - | E | A | D | D | A | R | L | A | G | L | A | K | P | F | F | A | A | L | G | I | D | - | - | - | - | A | - | - | - | - | - | - | - | - | - | G | A | I | G | P | D | L | V | S | V | M | G | L | M | K | - | - | - | - | - | - | - | - | - | D | A | S | T | V | K | E | I | A | E | N | S | A | M | F | Y | R | A | - | - | - | - | - | P | A | T | P | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | A | V | L | L | L | F | G | V | V | S | R | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | D | Y | H | K | L | D | F | F | N | S | Y | Y | I | R | - | - | E | K | K | D | E | D | L | F | N | L | L | L | P | F | F | Q | K | - | G | Y | V | S | K | P | S | T | - | - | - | - | - | - | - | - | - | L | E | E | N | Q | K | L | K | L | L | I | P | L | I | K | S | - | - | - | - | - | - | - | - | R | I | - | K | L | S | D | A | L | N | M | T | K | F | F | Y | E | - | - | - | - | - | - | - | - | D | I | W | N | E | F | E | - | - | - | - | - | - | - | - | - | - | C | I | L | E | L | K | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | R | S | E | N | D | A | - | - | - | - | - | - | - | - | - | G | L | P | I | R | I | A | A | L | G | S | - | V | S | P | P | L | F | D | S | L | K | L | I | G | - | - | - | - | - | - | S | K | V | F | E | R | I | A | Q | F | R | N | - | - |
| - | - | - | - | - | - | - | - | S | W | L | N | W | L | N | A | H | Y | L | Q | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | L | L | E | L | L | P | F | S | F | - | S | D | L | P | A | L | L | D | A | L | K | - | - | - | - | - | - | - | - | - | - | - | - | E | S | Q | T | L | K | E | L | A | L | K | I | D | E | V | L | I | A | P | Y | E | E | F | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | L | M | P | L | L | E | F | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | E | I | A | - | - | - | - | - | - | - | G | S | F | M | Q | P | L | R | L | A | L | L | G | G | - | - | G | G | I | G | L | E | A | L | F | I | L | G | - | - | - | - | - | - | - | - | K | E | S | R | I | N | F | L | - | - | - | - |
| - | - | - | - | - | - | D | M | - | A | L | G | W | V | N | Q | H | F | L | K | - | - | T | E | D | V | A | A | I | V | P | H | L | V | Y | Q | L | Q | K | L | G | L | D | V | A | - | - | A | - | - | - | - | - | - | - | - | - | G | P | A | - | - | - | P | E | D | V | V | V | A | L | R | E | - | - | - | - | - | - | - | - | R | V | Q | T | L | K | E | M | A | E | K | A | V | V | W | Y | Q | P | - | - | - | - | - | L | T | E | Y | D | E | A | A | V | A | K | H | F | - | K | A | G | A | E | V | A | L | G | K | A | R | E | L | L | A | A | L | P | - | W | T | A | E | S | V | G | V | A | L | H | D | A | A | A | A | L | E | I | G | M | G | K | V | A | Q | P | L | R | V | A | I | T | G | T | Q | V | S | P | D | I | S | H | T | V | Y | L | A | G | R | - | E | Q | A | L | - | R | I | D | V | A | I | T | K | V | A | - | - | - |
