solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1knca- ( 2 ) siekLkaaLp---eyakdikl----nLssIt-
d1vkea- ( 6 ) farlngtlntkff-nldsavyrpgkldvktkElm
d2af7a1 ( 2 ) eryrrGe--ilnr-nksytairdlapdlarfvae
d2cwqa1 ( 2 ) drthervlqaaenlgeGlpraipllAekapglLlehgr
d2gmya1 ( 2 ) ktrinyaka----speafkavma
d2ouwa1 ( 2 ) atvrlLddaeistlVavFddIratrgsfVnniWrglAn-dpal-LrtWq
d2oyoa1 ( 12 ) iSslpvpdatqVpegVrklwakaeanigfvPnvfrAqAv-ngeqFlawwn
d2q0ta1 ( 2 ) tqttpqPdpsrLrdeLvlHgkaspeWdSlVrldprfVdayLk
d3c1la- ( 5 ) isald-----glsepTkayfakcee-lglVpnvlkayAf-ddkklraftd
aa aaaa aaaaaaa
60 70 80 90 100
d1knca- ( 26 ) -----r--ssvLdqeqLWGTLLASAaAt--rnpqVladIgaeAtdhLsaa
d1vkea- ( 50 ) gLvasTvl----rçddçIryhLvrCVqegAsdeeIfeaLdiAlvvg--gs
d2af7a1 ( 40 ) fayGdvysr-gvllktrelltLaaltvlra--ddqLkshVrgAlna--g-
d2cwqa1 ( 41 ) swtyapekg-aldektrtlillgiAlatgs--eacvka-ahrAkrl--gl
d2gmya1 ( 21 ) lenyVqs--SgLehrFIhLIkLrASiin--gCafCvdmHvkeSrhd-g--
d2ouwa1 ( 55 ) vkt--vvgg-aldpltre-iylavstansç--syçahshtaaarak--g-
d2oyoa1 ( 61 ) yfnllLnkegyLtnaeRELVAVVVSgvn--rClYCavshgAaLReflg--
d2q0ta1 ( 45 ) fagvPr--rnhLddktrAFIaLaADACatqlyapGvarhirAlsfgAtre
d3c1la- ( 53 ) iyndll-gesgLskldre-IAVAVSsin--hCyyCltahGaaVRqlsg--
aaaaaaaaaaaaaa aaaaaaaaaaaaa
110 120 130 140 150
d1knca- ( 67 ) ArhAAlgAaaiMgmNNvfyrgrgfle---------------------gry
d1vkea- ( 94 ) iViphlrAvgfLeeLremengetis
d2af7a1 ( 85 ) -CskeIieviqavyagfpaainavlAAkevfte
d2cwqa1 ( 87 ) skealletlkiarqaqanavlghaapllevl
d2gmya1 ( 64 ) lseqWInlMsvWreSpvYt-------------------------------
d2ouwa1 ( 100 ) tpaqhaevlaiiglaaqtnalvtaqipvdaflv
d2oyoa1 ( 107 ) dpqkAdaVavnwrhAd-Lt-------------------------------
d2q0ta1 ( 95 ) eliEVleLvsTIgihtsnvgvpvllevleeeglrkgapplderrqklkae
d3c1la- ( 99 ) dpalGelv--nFraad-ls-------------------------------
aaa
160 170 180 190 200
d1knca- ( 96 ) ddlrpglrmniia-----------------------nPgipkanFeLWSF
d1vkea-
d2af7a1
d2cwqa1
d2gmya1 ( 83 ) ------------------------------------------eqErALLg
d2ouwa1
d2oyoa1 ( 126 ) ------------------------------------------reqaLAay
d2q0ta1 ( 145 ) FetnrgywhptwegLLeldpdlFeayvefSsvPwrtgvLspkiKEFyCAF
d3c1la- ( 117 ) ------------------------------------------prqtalef
210 220 230 240 250
d1knca- ( 123 ) AVSaingCshCl--vahehtLrtvgvdreaIfeAlkAAaiVsgVAqaLat
d1vkea-
d2af7a1
d2cwqa1
d2gmya1 ( 91 ) wVDavTkiaetgapddafetLra-hFsdeeivkItvAIgAIntwNriAvg
d2ouwa1
d2oyoa1 ( 134 ) AeklTrhpa--evtaadlepLravgLddhqi-eLvqVIg-FnltNrvSsa
d2q0ta1 ( 196 ) DASathlyvpgl--klhirnAlrygAta-eelELleiVs-vtgihgaElg
d3c1la- ( 126 ) AvklTeepa--kiveadraaLrkagFsdrdiwdIasTAa-ffnsNrvaaa
260
d1knca- ( 171 ) ieals
d1vkea-
d2af7a1
d2cwqa1
d2gmya1 ( 140 ) fr--sqhp----vea
d2ouwa1
d2oyoa1 ( 182 ) lg--FvPnpeyyqar
d2q0ta1 ( 243 ) Aplleaalkrsgaaa
d3c1la- ( 174 ) id---rPndeyhaar
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Antioxidant defense protein AhpD | AhpD | Mycobacterium tuberculosis [TaxId: 1773] | 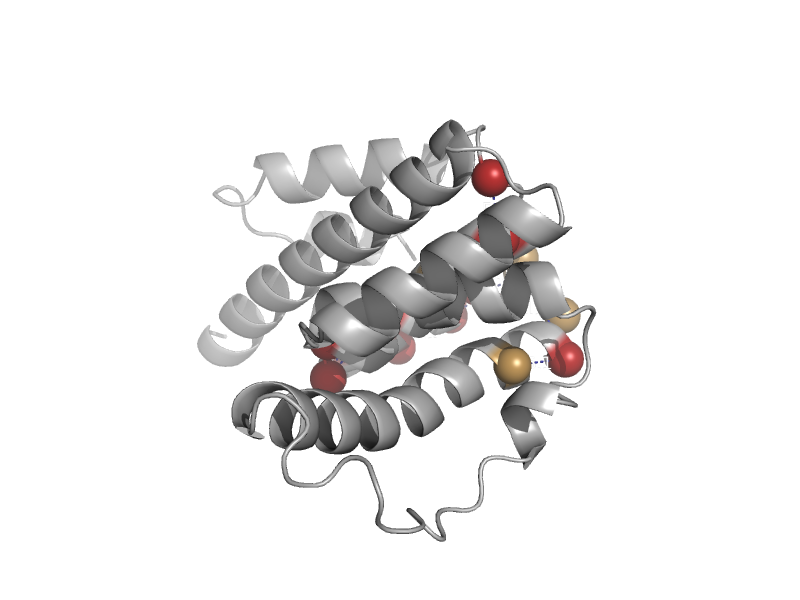 | view | |
| Hypothetical protein TM1620 | CMD-like | Thermotoga maritima [TaxId: 2336] | 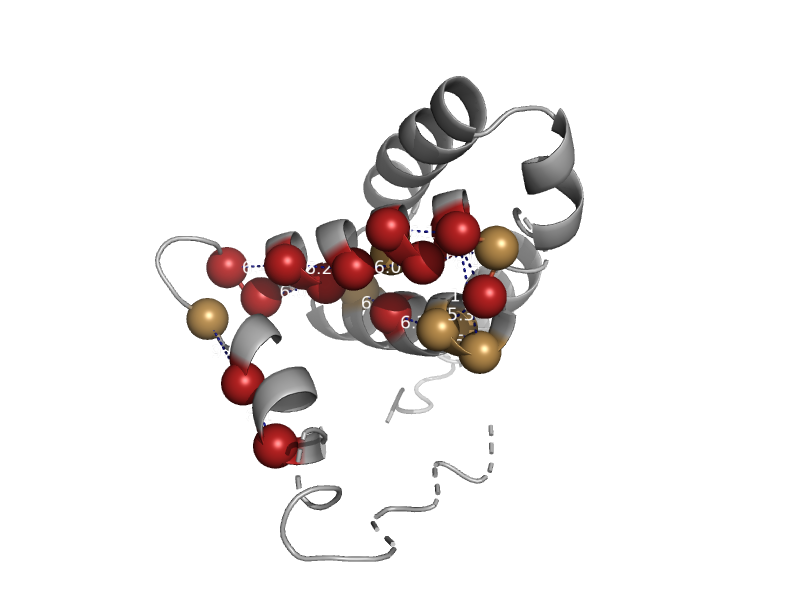 | view | |
| Gamma-carboxymuconolactone decarboxylase, CMD | CMD-like | Methanobacterium thermoautotrophicum [TaxId: 145262] | 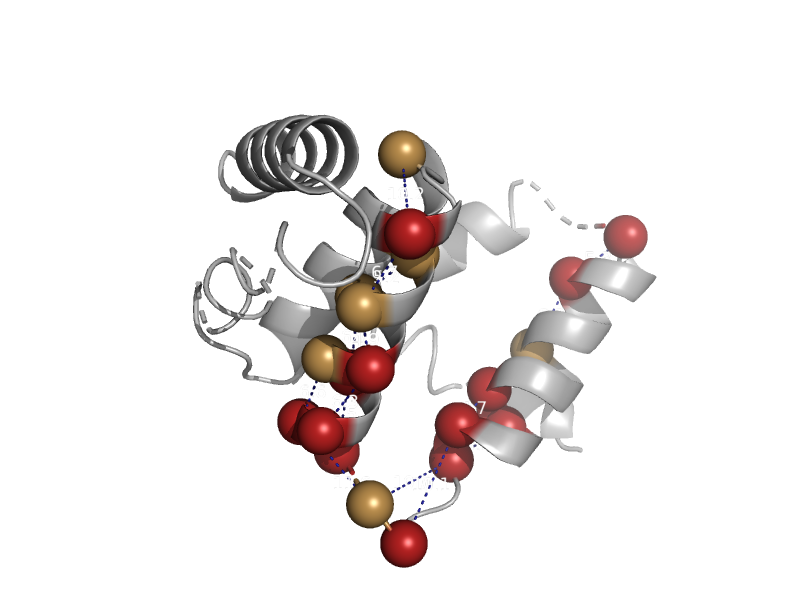 | view | |
| Hypothetical protein TTHA0727 | TTHA0727-like | Thermus thermophilus [TaxId: 274] | 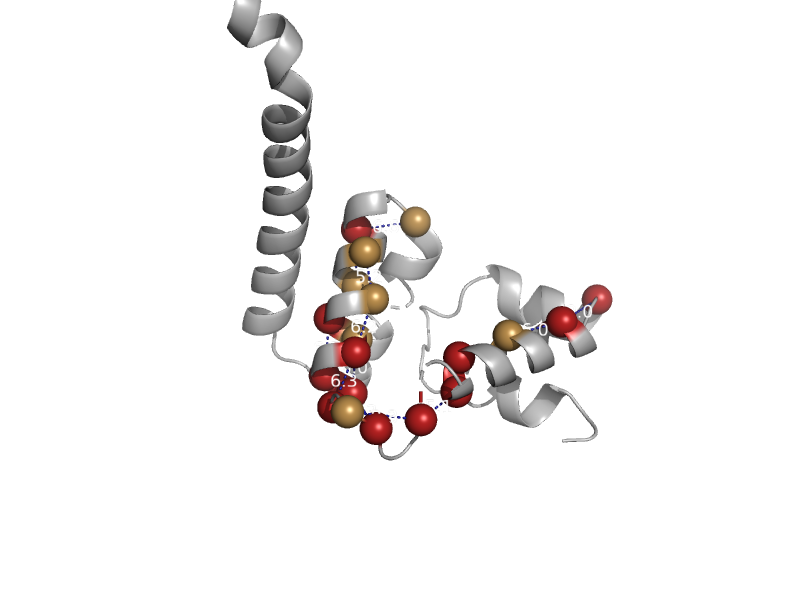 | view | |
| Hypothetical protein Atu0492 | Atu0492-like | Agrobacterium tumefaciens [TaxId: 358] |  | view | |
| Hypothetical protein Rru_A0301 | TTHA0727-like | Rhodospirillum rubrum [TaxId: 1085] | 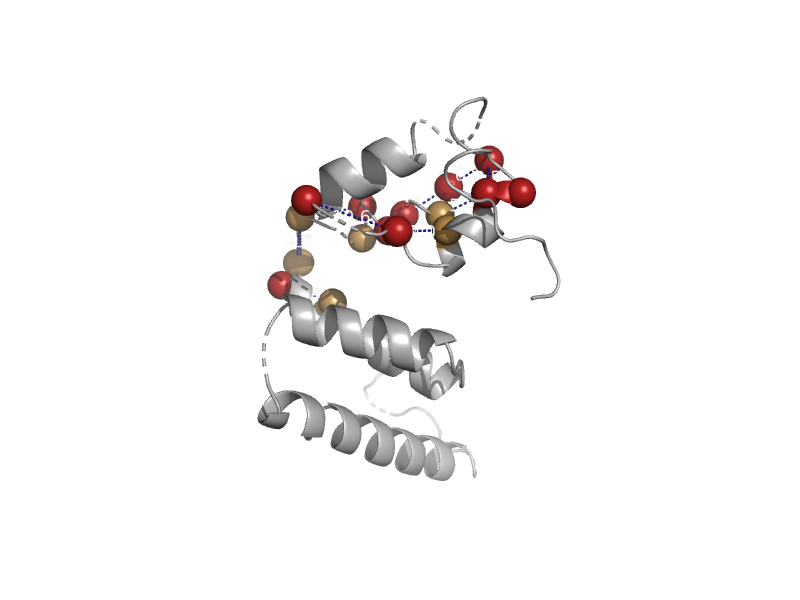 | view | |
| Uncharacterized protein Dgeo_1446 | Atu0492-like | Deinococcus geothermalis [TaxId: 68909] | 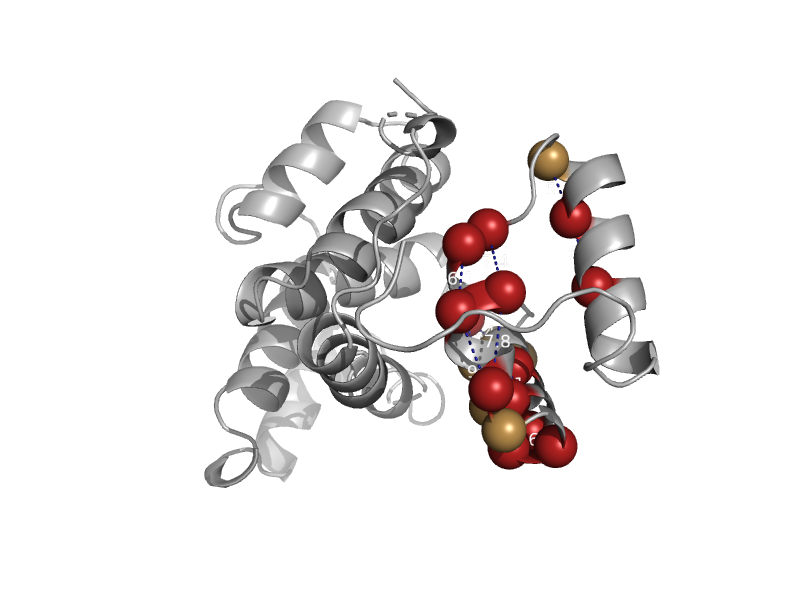 | view | |
| Hypothetical protein Bxeno_B2006 | AhpD | Burkholderia xenovorans [TaxId: 36873] | 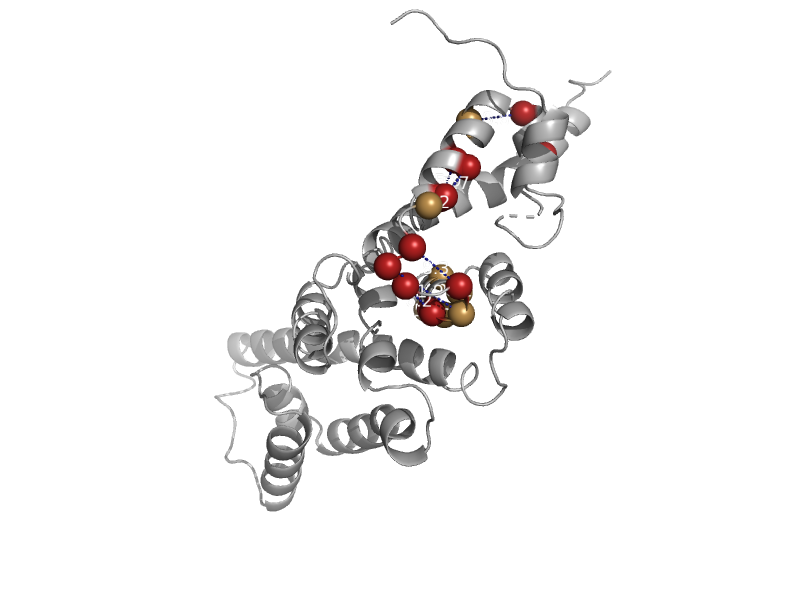 | view | |
| automated matches | Atu0492-like | Mesorhizobium loti [TaxId: 266835] | 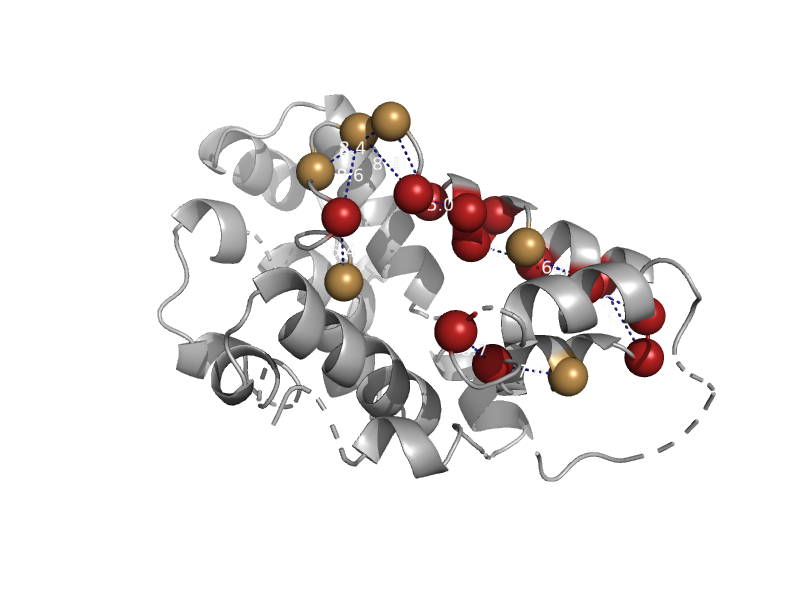 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | 230 | 240 | 250 | 260 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | I | E | K | L | K | A | A | L | P | - | - | - | E | Y | A | K | D | I | K | L | - | - | - | - | N | L | S | S | I | T | - | - | - | - | - | - | R | - | - | S | S | V | L | D | Q | E | Q | L | W | G | T | L | L | A | S | A | A | A | T | - | - | R | N | P | Q | V | L | A | D | I | G | A | E | A | T | D | H | L | S | A | A | A | R | H | A | A | L | G | A | A | A | I | M | G | M | N | N | V | F | Y | R | G | R | G | F | L | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | R | Y | D | D | L | R | P | G | L | R | M | N | I | I | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | P | G | I | P | K | A | N | F | E | L | W | S | F | A | V | S | A | I | N | G | C | S | H | C | L | - | - | V | A | H | E | H | T | L | R | T | V | G | V | D | R | E | A | I | F | E | A | L | K | A | A | A | I | V | S | G | V | A | Q | A | L | A | T | I | E | A | L | S | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | F | A | R | L | N | G | T | L | N | T | K | F | F | - | N | L | D | S | A | V | Y | R | P | G | K | L | D | V | K | T | K | E | L | M | G | L | V | A | S | T | V | L | - | - | - | - | R | C | D | D | C | I | R | Y | H | L | V | R | C | V | Q | E | G | A | S | D | E | E | I | F | E | A | L | D | I | A | L | V | V | G | - | - | G | S | I | V | I | P | H | L | R | A | V | G | F | L | E | E | L | R | E | M | E | N | G | E | T | I | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | R | Y | R | R | G | E | - | - | I | L | N | R | - | N | K | S | Y | T | A | I | R | D | L | A | P | D | L | A | R | F | V | A | E | F | A | Y | G | D | V | Y | S | R | - | G | V | L | L | K | T | R | E | L | L | T | L | A | A | L | T | V | L | R | A | - | - | D | D | Q | L | K | S | H | V | R | G | A | L | N | A | - | - | G | - | - | C | S | K | E | I | I | E | V | I | Q | A | V | Y | A | G | F | P | A | A | I | N | A | V | L | A | A | K | E | V | F | T | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | D | R | T | H | E | R | V | L | Q | A | A | E | N | L | G | E | G | L | P | R | A | I | P | L | L | A | E | K | A | P | G | L | L | L | E | H | G | R | S | W | T | Y | A | P | E | K | G | - | A | L | D | E | K | T | R | T | L | I | L | L | G | I | A | L | A | T | G | S | - | - | E | A | C | V | K | A | - | A | H | R | A | K | R | L | - | - | G | L | S | K | E | A | L | L | E | T | L | K | I | A | R | Q | A | Q | A | N | A | V | L | G | H | A | A | P | L | L | E | V | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | T | R | I | N | Y | A | K | A | - | - | - | - | S | P | E | A | F | K | A | V | M | A | L | E | N | Y | V | Q | S | - | - | S | G | L | E | H | R | F | I | H | L | I | K | L | R | A | S | I | I | N | - | - | G | C | A | F | C | V | D | M | H | V | K | E | S | R | H | D | - | G | - | - | L | S | E | Q | W | I | N | L | M | S | V | W | R | E | S | P | V | Y | T | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | Q | E | R | A | L | L | G | W | V | D | A | V | T | K | I | A | E | T | G | A | P | D | D | A | F | E | T | L | R | A | - | H | F | S | D | E | E | I | V | K | I | T | V | A | I | G | A | I | N | T | W | N | R | I | A | V | G | F | R | - | - | S | Q | H | P | - | - | - | - | V | E | A |
| - | A | T | V | R | L | L | D | D | A | E | I | S | T | L | V | A | V | F | D | D | I | R | A | T | R | G | S | F | V | N | N | I | W | R | G | L | A | N | - | D | P | A | L | - | L | R | T | W | Q | V | K | T | - | - | V | V | G | G | - | A | L | D | P | L | T | R | E | - | I | Y | L | A | V | S | T | A | N | S | C | - | - | S | Y | C | A | H | S | H | T | A | A | A | R | A | K | - | - | G | - | T | P | A | Q | H | A | E | V | L | A | I | I | G | L | A | A | Q | T | N | A | L | V | T | A | Q | I | P | V | D | A | F | L | V | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| I | S | S | L | P | V | P | D | A | T | Q | V | P | E | G | V | R | K | L | W | A | K | A | E | A | N | I | G | F | V | P | N | V | F | R | A | Q | A | V | - | N | G | E | Q | F | L | A | W | W | N | Y | F | N | L | L | L | N | K | E | G | Y | L | T | N | A | E | R | E | L | V | A | V | V | V | S | G | V | N | - | - | R | C | L | Y | C | A | V | S | H | G | A | A | L | R | E | F | L | G | - | - | D | P | Q | K | A | D | A | V | A | V | N | W | R | H | A | D | - | L | T | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | E | Q | A | L | A | A | Y | A | E | K | L | T | R | H | P | A | - | - | E | V | T | A | A | D | L | E | P | L | R | A | V | G | L | D | D | H | Q | I | - | E | L | V | Q | V | I | G | - | F | N | L | T | N | R | V | S | S | A | L | G | - | - | F | V | P | N | P | E | Y | Y | Q | A | R |
| - | - | - | - | - | - | - | - | T | Q | T | T | P | Q | P | D | P | S | R | L | R | D | E | L | V | L | H | G | K | A | S | P | E | W | D | S | L | V | R | L | D | P | R | F | V | D | A | Y | L | K | F | A | G | V | P | R | - | - | R | N | H | L | D | D | K | T | R | A | F | I | A | L | A | A | D | A | C | A | T | Q | L | Y | A | P | G | V | A | R | H | I | R | A | L | S | F | G | A | T | R | E | E | L | I | E | V | L | E | L | V | S | T | I | G | I | H | T | S | N | V | G | V | P | V | L | L | E | V | L | E | E | E | G | L | R | K | G | A | P | P | L | D | E | R | R | Q | K | L | K | A | E | F | E | T | N | R | G | Y | W | H | P | T | W | E | G | L | L | E | L | D | P | D | L | F | E | A | Y | V | E | F | S | S | V | P | W | R | T | G | V | L | S | P | K | I | K | E | F | Y | C | A | F | D | A | S | A | T | H | L | Y | V | P | G | L | - | - | K | L | H | I | R | N | A | L | R | Y | G | A | T | A | - | E | E | L | E | L | L | E | I | V | S | - | V | T | G | I | H | G | A | E | L | G | A | P | L | L | E | A | A | L | K | R | S | G | A | A | A |
| I | S | A | L | D | - | - | - | - | - | G | L | S | E | P | T | K | A | Y | F | A | K | C | E | E | - | L | G | L | V | P | N | V | L | K | A | Y | A | F | - | D | D | K | K | L | R | A | F | T | D | I | Y | N | D | L | L | - | G | E | S | G | L | S | K | L | D | R | E | - | I | A | V | A | V | S | S | I | N | - | - | H | C | Y | Y | C | L | T | A | H | G | A | A | V | R | Q | L | S | G | - | - | D | P | A | L | G | E | L | V | - | - | N | F | R | A | A | D | - | L | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | R | Q | T | A | L | E | F | A | V | K | L | T | E | E | P | A | - | - | K | I | V | E | A | D | R | A | A | L | R | K | A | G | F | S | D | R | D | I | W | D | I | A | S | T | A | A | - | F | F | N | S | N | R | V | A | A | A | I | D | - | - | - | R | P | N | D | E | Y | H | A | A | R |
| Member | Chain | UniProtKB | GO term |
|---|---|---|---|
| d1knca_ | A | P9WQB5 | GO:0004601 |
| d1knca_ | A | P9WQB5 | GO:0005515 |
| d1knca_ | A | P9WQB5 | GO:0005829 |
| d1knca_ | A | P9WQB5 | GO:0005886 |
| d1knca_ | A | P9WQB5 | GO:0006979 |
| d1knca_ | A | P9WQB5 | GO:0008785 |
| d1knca_ | A | P9WQB5 | GO:0015036 |
| d1knca_ | A | P9WQB5 | GO:0016209 |
| d1knca_ | A | P9WQB5 | GO:0032843 |
| d1knca_ | A | P9WQB5 | GO:0045454 |
| d1knca_ | A | P9WQB5 | GO:0051920 |
| d1knca_ | A | P9WQB5 | GO:0098869 |
| d1vkea_ | A | Q9X1V5 | GO:0016491 |
| d1vkea_ | A | Q9X1V5 | GO:0051920 |
| d1vkea_ | A | Q9X1V5 | GO:0098869 |
| d2af7a1 | A | O26336 | GO:0051920 |
| d2af7a1 | A | O26336 | GO:0098869 |
| d2cwqa1 | A | Q5SMF5 | GO:0016491 |
| d2cwqa1 | A | Q5SMF5 | GO:0051920 |
| d2cwqa1 | A | Q5SMF5 | GO:0098869 |
| d2gmya1 | A | Q7D1C9 | GO:0051920 |
| d2gmya1 | A | Q7D1C9 | GO:0098869 |
| d2ouwa1 | A | Q2RXN9 | GO:0051920 |
| d2ouwa1 | A | Q2RXN9 | GO:0098869 |
| d2oyoa1 | A | Q1IYE3 | GO:0004601 |
| d2oyoa1 | A | Q1IYE3 | GO:0051920 |
| d2oyoa1 | A | Q1IYE3 | GO:0098869 |
| d2q0ta1 | A | Q13LR5 | GO:0016491 |
| d2q0ta1 | A | Q13LR5 | GO:0051920 |
| d2q0ta1 | A | Q13LR5 | GO:0098869 |
