solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1h3za- ( 118 ) servnYkpgmrvltkms---
d1khca- ( 223 ) eYd-efgigdLVWGkik---
d1mhna- ( 89 ) lqqwkvgdkCSAiws---
d1oz2a1 ( 204 ) ecwswesyleeqkaitapvslfqdsqavthnkN-gfklgmkLEGidp---
d1oz2a2 ( 314 ) fswsqylrstraqaapkhlfv--sqshsppPlgfqvgmklEAvdr---
d1wjra1 ( 8 ) pidlitvgSliElqds---
d2g3ra1 (1485 ) sfvglrVvAkws---
d2hqxa1 ( 8 ) tqfqklMenMrndiasp---gyarg-fCIAkf-vd-
d2jnga1 ( 5 ) rsefasgntyAlyvrdtlqpgmrvrMlddyee
d2l89a- ( 45 ) saddrlnfGdrilvkap---
d2l8da1 ( 5 ) mpnrkYadgevvmGrwp---
d3fdra- ( 6 ) qld-lv-nMtqhYensvpetvhvgdiVAAplptn-
d4rxja- ( 958 ) lhykqiVwVlg----
d6iipa1 ( 8 ) eYkagdlVFAkmk---
bbbbb
60 70 80 90 100
d1h3za- ( 135 ) g--fpwwPSMvvteskMtsvArkskpkr-----agtfypviFFpnke---
d1khca- ( 242 ) g---swWPAmVvswaTskrqAmp------------gmRwVqWfgdgk---
d1mhna- ( 104 ) ed-gciypAtIasIdfk-----------------retCvVvYtgygn---
d1oz2a1 ( 250 ) qhpsmyfiLtVaevc-------------------gyrLrLhFdgy--sec
d1oz2a2 ( 357 ) mnpslVcvAsVtdvv-------------------dsrFlVhFdnw--ddt
d1wjra1 ( 24 ) qnpfqYWivsvienv-------------------gGrlrlryvgledtes
d2g3ra1 (1497 ) sn-gyfysGkItrdvga------------------gkykLlFd-dgy---
d2hqxa1 ( 46 ) ---gewyRArvekvesp------------------akihVfYIDygn---
d2jnga1 ( 37 ) isagdeGeFrqsn------------------ngv-ppvqvfWestgr---
d2l89a- ( 62 ) g--ypwwPAlllrr-ket------kdslntnssfnvlYkvlfFpdfn---
d2l8da1 ( 22 ) gs-vlyyevqvtsYdda-----------------shlYtvkYk-dgt---
d3fdra- ( 42 ) ---gswyRArVlgtlen------------------gnldLyfVDfg----
d4rxja- ( 970 ) --nyrwwpAeIcnpsVp----------niqglhdlgDfPVfffgshd---
d6iipa1 ( 21 ) g--yphwPArIdelpegavkppa------------nkypIfFFGthe---
bbbbbbb bbbbbb
110 120 130 140 150
d1h3za- ( 175 ) -YlWtgsd--sltpl-tseaisqfle-----kpkpktaslikAYkmAqst
d1khca- ( 276 ) -fseisad--kLva--LG-lFsqhFn-----latfnlvsYrkAMyhTLek
d1mhna- ( 133 ) -reeqnLs--dLlsp-ice
d1oz2a1 ( 279 ) hdfwvnAnspdIhp---agwfektghklqppk-gy------keee
d1oz2a2 ( 386 ) ydywCdpsspyIhp---vgwCqkqgkpltppq-dyp-----dpdn
d1wjra1 ( 55 ) ydqwlfYldyrlrp---vgwCqenkyrmdPpseiyplkmaseWkctleks
d2g3ra1 (1524 ) -ecdVlgk--dIllc-dp
d2hqxa1 ( 72 ) -revLpst--rLGtLs---pafstvlpaqa
d2jnga1 ( 65 ) -tywvhWh--mleilG--fee
d2l89a- ( 100 ) -fawvkrn--svkpl-ldseiakflg-----sskrkskelieAYeaSktp
d2l8da1 ( 50 ) -elalkes--dirlq-ssfk------------------------------
d3fdra- ( 68 ) -ngdCpl---dLraLr---sdfl-slpfqaie
d4rxja- (1008 ) --ywVhqg--rVfpyvegfaegqtsintfale------------------
d6iipa1 ( 54 ) -taflgpk--dLfp--Ykeykdkfgk-------snkrkgFneGlweIenn
bbbb b
160 170 180
d1h3za- ( 216 ) pdldslsvps
d1khca- ( 316 ) ArvrAgktfssspgesedqLkpMlewAhggFkptgigLp
d1mhna-
d1oz2a1
d1oz2a2
d1wjra1 ( 102 ) lidAakfplpmevfkdhadl
d2g3ra1
d2hqxa1
d2jnga1
d2l89a- ( 141 ) pdlkeesstdle
d2l8da1 ( 66 ) --------------------------------------q
d3fdra-
d4rxja- (1043 ) -------------------eaakrfqelkaqresealei
d6iipa1 ( 92 ) pgv
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Hypothetical protein SPBC215.07c | PWWP domain | Fission yeast (Schizosaccharomyces pombe) [TaxId: 4896] | 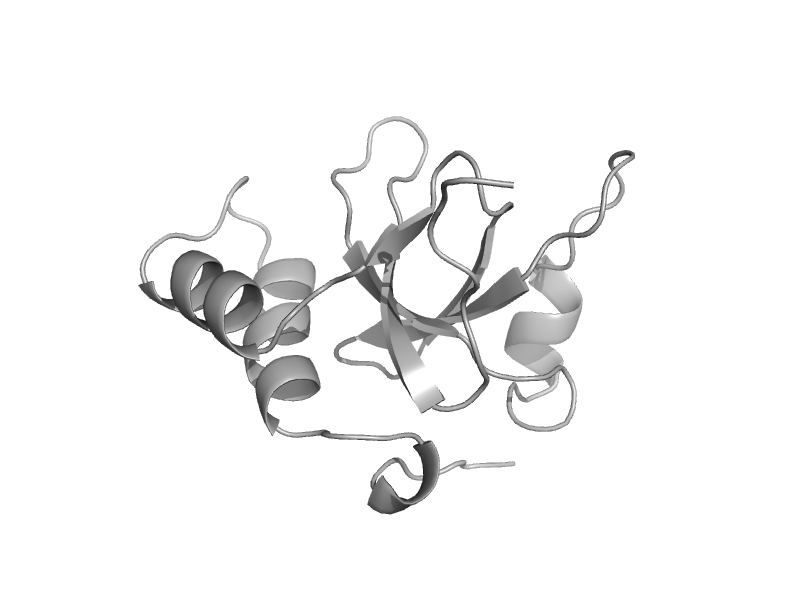 | view | |
| DNA methyltransferase DNMT3B | PWWP domain | Mouse (Mus musculus) [TaxId: 10090] | 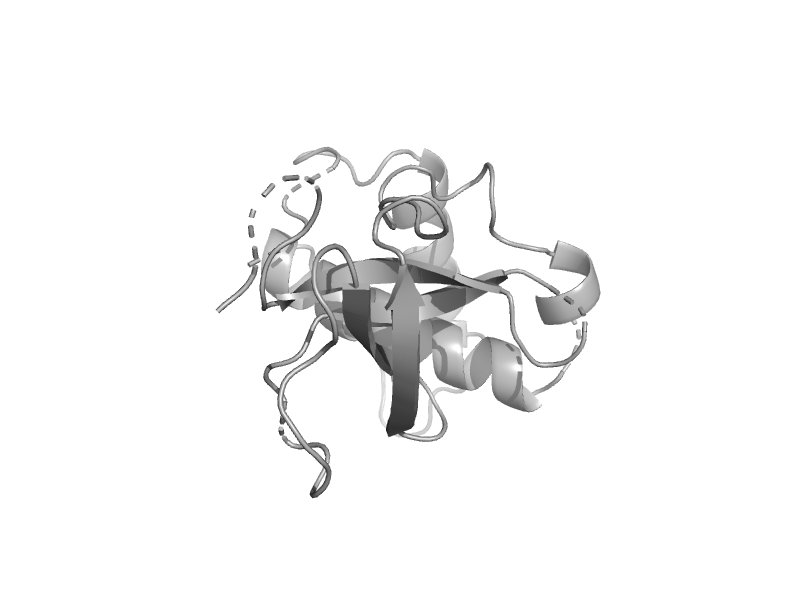 | view | |
| Survival motor neuron protein 1, smn | Tudor domain | Human (Homo sapiens) [TaxId: 9606] | 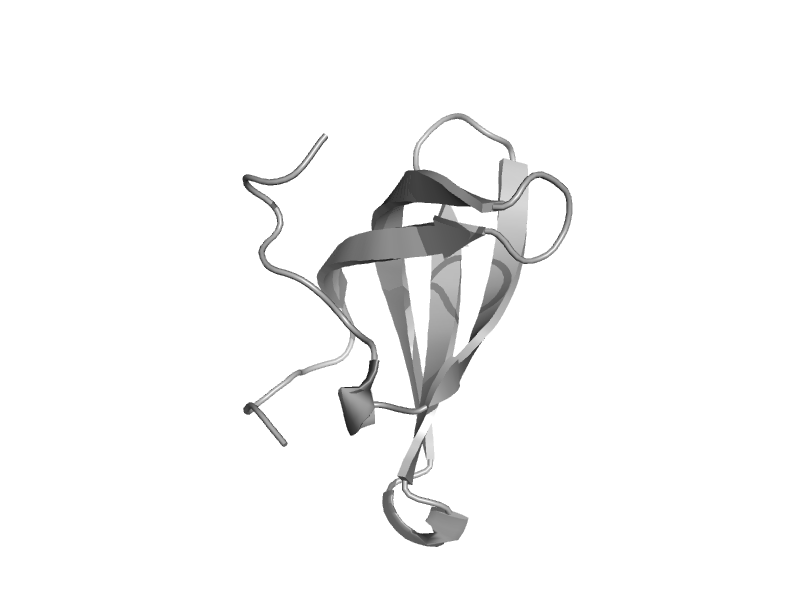 | view | |
| Lethal(3)malignant brain tumor-like protein | MBT repeat | Human (Homo sapiens) [TaxId: 9606] | 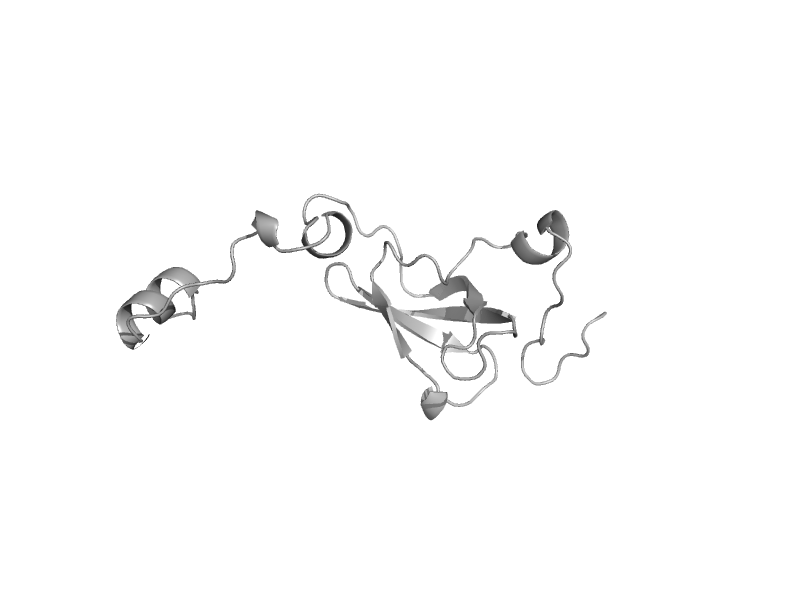 | view | |
| Lethal(3)malignant brain tumor-like protein | MBT repeat | Human (Homo sapiens) [TaxId: 9606] | 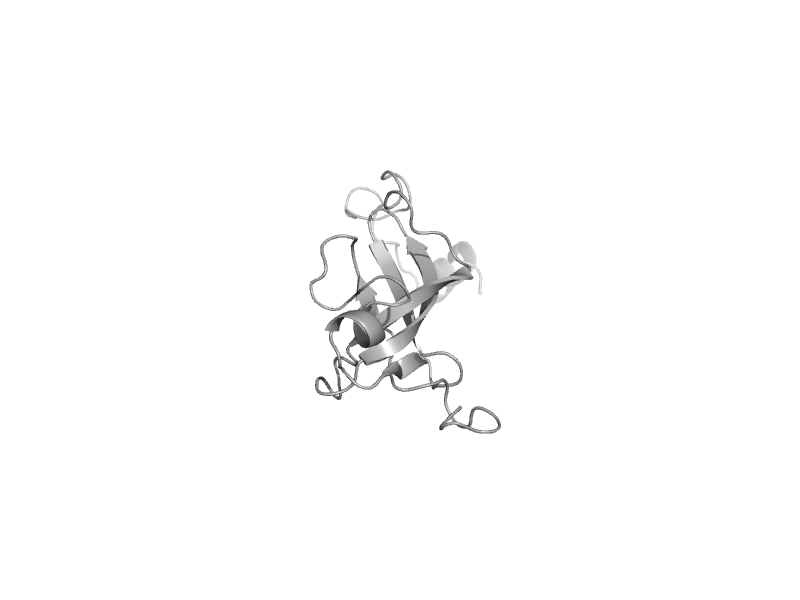 | view | |
| Scm-like with four MBT domains protein 2, SFMBT2 (KIAA1617) | MBT repeat | Human (Homo sapiens) [TaxId: 9606] | 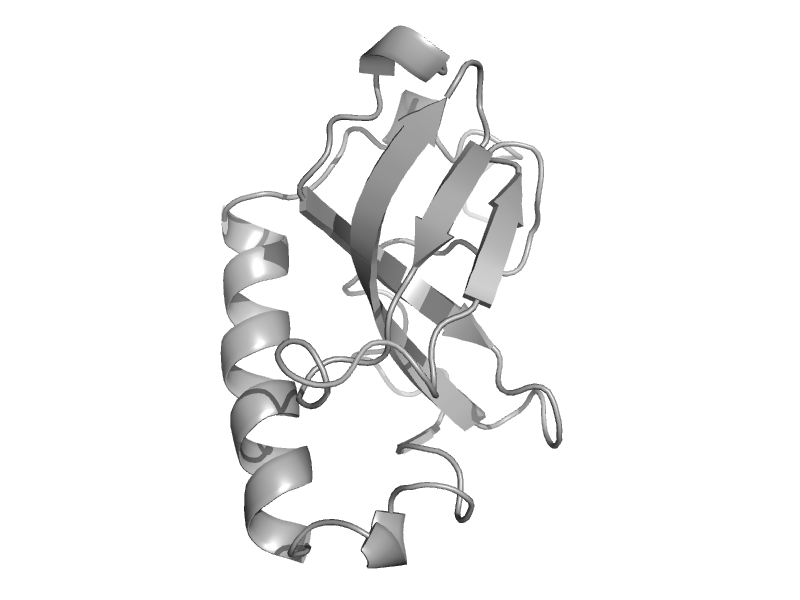 | view | |
| p53-binding protein 1, 53BP1, N-terminal domain | Tudor domain | Human (Homo sapiens) [TaxId: 9606] | 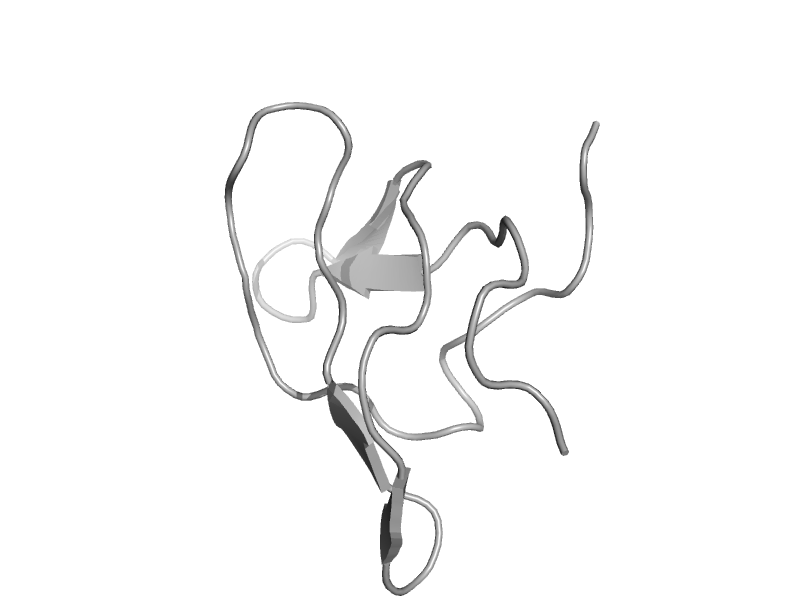 | view | |
| P100 co-activator, SND1 | Tudor domain | Human (Homo sapiens) [TaxId: 9606] | 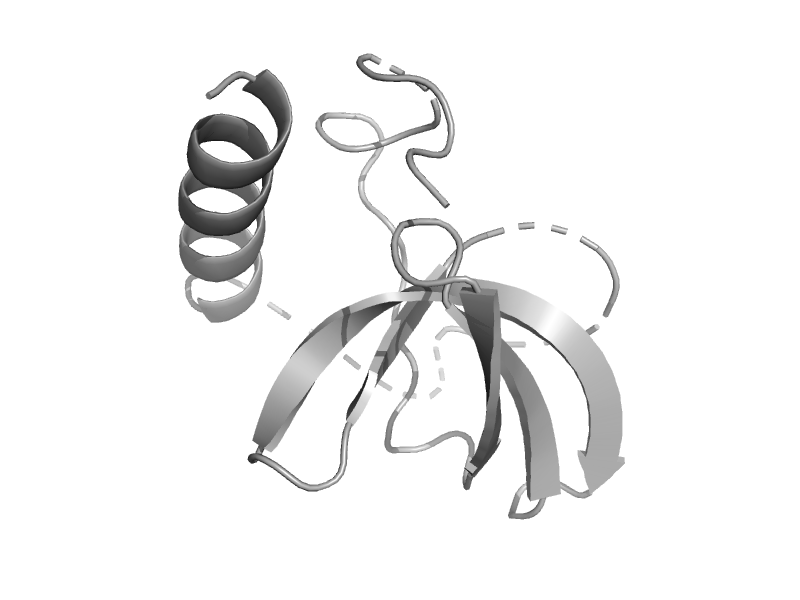 | view | |
| Cullin-7 | CPH domain | Human (Homo sapiens) [TaxId: 9606] | 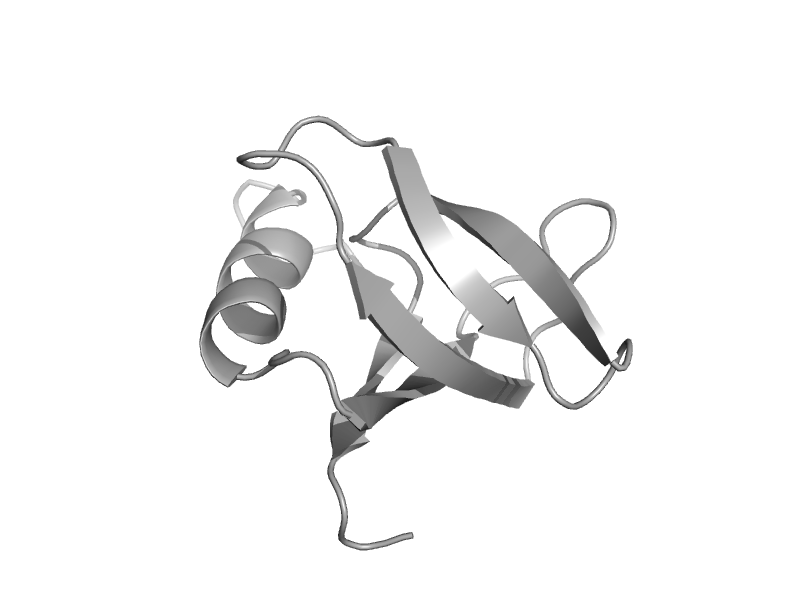 | view | |
| automated matches | automated matches | Fission yeast (Schizosaccharomyces pombe) [TaxId: 4896] | 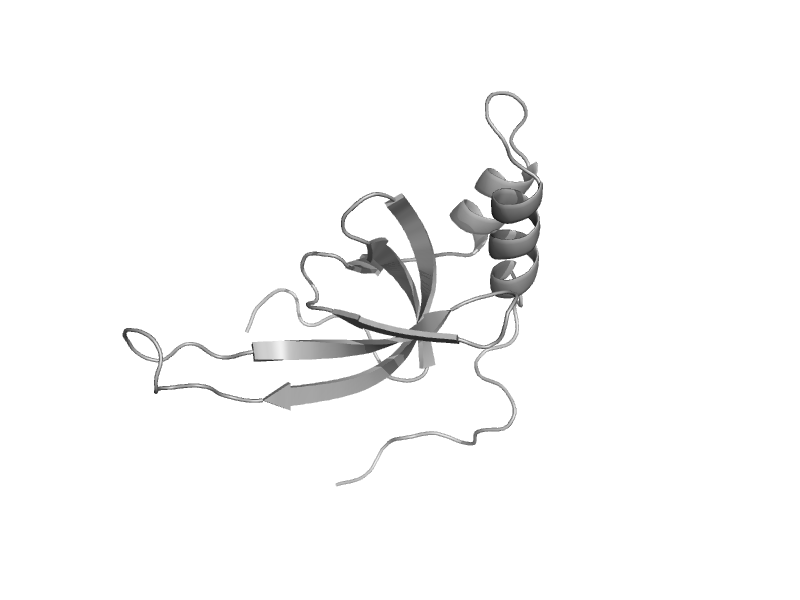 | view | |
| Lamin-b receptor | Tudor domain | Chicken (Gallus gallus) [TaxId: 9031] | 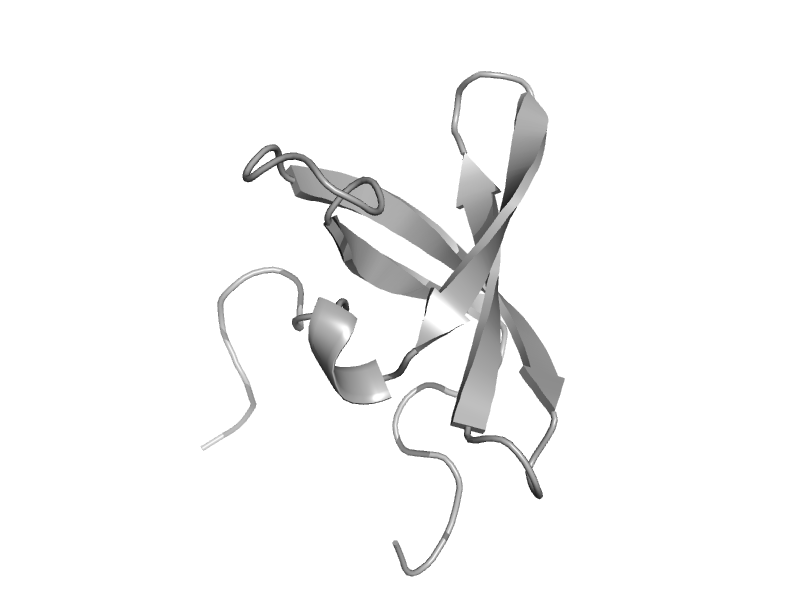 | view | |
| Tudor and KH domain-containing protein TDRKH | Tudor domain | Human (Homo sapiens) [TaxId: 9606] | 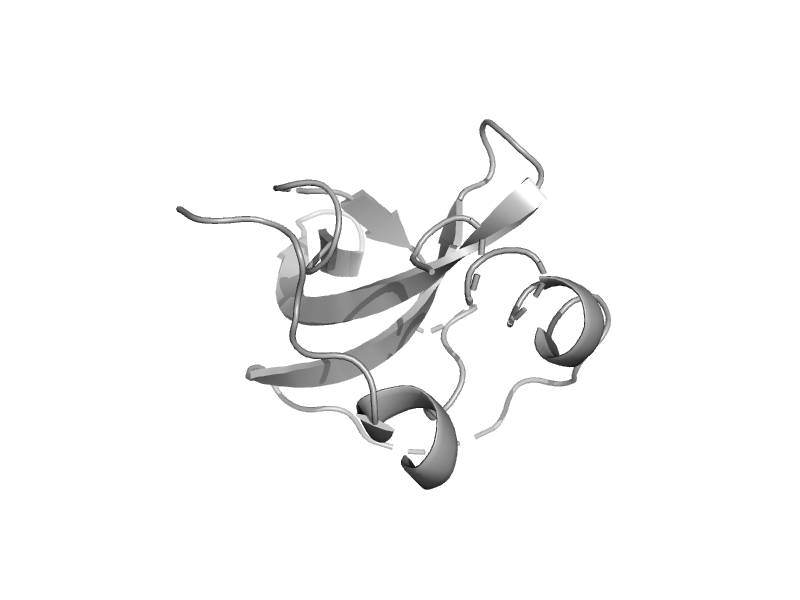 | view | |
| automated matches | PWWP domain | Human (Homo sapiens) [TaxId: 9606] |  | view | |
| automated matches | PWWP domain | Human (Homo sapiens) [TaxId: 9606] |  | view |
| Domain ID | Name |
|---|---|
| d2qqra1 | 2qqr A:897-955 |
| d2qqra2 | 2qqr A:956-1011 |
| d2g3ra2 | 2g3r A:1538-1603 |
| d3s6wa- | 3s6w A: |
| d2r58a2 | 2r58 A:282-385 |
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | E | R | V | N | Y | K | P | G | M | R | V | L | T | K | M | S | - | - | - | G | - | - | F | P | W | W | P | S | M | V | V | T | E | S | K | M | T | S | V | A | R | K | S | K | P | K | R | - | - | - | - | - | A | G | T | F | Y | P | V | I | F | F | P | N | K | E | - | - | - | - | Y | L | W | T | G | S | D | - | - | S | L | T | P | L | - | T | S | E | A | I | S | Q | F | L | E | - | - | - | - | - | K | P | K | P | K | T | A | S | L | I | K | A | Y | K | M | A | Q | S | T | P | D | L | D | S | L | S | V | P | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | Y | D | - | E | F | G | I | G | D | L | V | W | G | K | I | K | - | - | - | G | - | - | - | S | W | W | P | A | M | V | V | S | W | A | T | S | K | R | Q | A | M | P | - | - | - | - | - | - | - | - | - | - | - | - | G | M | R | W | V | Q | W | F | G | D | G | K | - | - | - | - | F | S | E | I | S | A | D | - | - | K | L | V | A | - | - | L | G | - | L | F | S | Q | H | F | N | - | - | - | - | - | L | A | T | F | N | L | V | S | Y | R | K | A | M | Y | H | T | L | E | K | A | R | V | R | A | G | K | T | F | S | S | S | P | G | E | S | E | D | Q | L | K | P | M | L | E | W | A | H | G | G | F | K | P | T | G | I | G | L | P |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | Q | Q | W | K | V | G | D | K | C | S | A | I | W | S | - | - | - | E | D | - | G | C | I | Y | P | A | T | I | A | S | I | D | F | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | E | T | C | V | V | V | Y | T | G | Y | G | N | - | - | - | - | R | E | E | Q | N | L | S | - | - | D | L | L | S | P | - | I | C | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| E | C | W | S | W | E | S | Y | L | E | E | Q | K | A | I | T | A | P | V | S | L | F | Q | D | S | Q | A | V | T | H | N | K | N | - | G | F | K | L | G | M | K | L | E | G | I | D | P | - | - | - | Q | H | P | S | M | Y | F | I | L | T | V | A | E | V | C | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | Y | R | L | R | L | H | F | D | G | Y | - | - | S | E | C | H | D | F | W | V | N | A | N | S | P | D | I | H | P | - | - | - | A | G | W | F | E | K | T | G | H | K | L | Q | P | P | K | - | G | Y | - | - | - | - | - | - | K | E | E | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | F | S | W | S | Q | Y | L | R | S | T | R | A | Q | A | A | P | K | H | L | F | V | - | - | S | Q | S | H | S | P | P | P | L | G | F | Q | V | G | M | K | L | E | A | V | D | R | - | - | - | M | N | P | S | L | V | C | V | A | S | V | T | D | V | V | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | S | R | F | L | V | H | F | D | N | W | - | - | D | D | T | Y | D | Y | W | C | D | P | S | S | P | Y | I | H | P | - | - | - | V | G | W | C | Q | K | Q | G | K | P | L | T | P | P | Q | - | D | Y | P | - | - | - | - | - | D | P | D | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | I | D | L | I | T | V | G | S | L | I | E | L | Q | D | S | - | - | - | Q | N | P | F | Q | Y | W | I | V | S | V | I | E | N | V | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | G | R | L | R | L | R | Y | V | G | L | E | D | T | E | S | Y | D | Q | W | L | F | Y | L | D | Y | R | L | R | P | - | - | - | V | G | W | C | Q | E | N | K | Y | R | M | D | P | P | S | E | I | Y | P | L | K | M | A | S | E | W | K | C | T | L | E | K | S | L | I | D | A | A | K | F | P | L | P | M | E | V | F | K | D | H | A | D | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | F | V | G | L | R | V | V | A | K | W | S | - | - | - | S | N | - | G | Y | F | Y | S | G | K | I | T | R | D | V | G | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | K | Y | K | L | L | F | D | - | D | G | Y | - | - | - | - | E | C | D | V | L | G | K | - | - | D | I | L | L | C | - | D | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | T | Q | F | Q | K | L | M | E | N | M | R | N | D | I | A | S | P | - | - | - | G | Y | A | R | G | - | F | C | I | A | K | F | - | V | D | - | - | - | - | G | E | W | Y | R | A | R | V | E | K | V | E | S | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | K | I | H | V | F | Y | I | D | Y | G | N | - | - | - | - | R | E | V | L | P | S | T | - | - | R | L | G | T | L | S | - | - | - | P | A | F | S | T | V | L | P | A | Q | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | S | E | F | A | S | G | N | T | Y | A | L | Y | V | R | D | T | L | Q | P | G | M | R | V | R | M | L | D | D | Y | E | E | I | S | A | G | D | E | G | E | F | R | Q | S | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | G | V | - | P | P | V | Q | V | F | W | E | S | T | G | R | - | - | - | - | T | Y | W | V | H | W | H | - | - | M | L | E | I | L | G | - | - | F | E | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | A | D | D | R | L | N | F | G | D | R | I | L | V | K | A | P | - | - | - | G | - | - | Y | P | W | W | P | A | L | L | L | R | R | - | K | E | T | - | - | - | - | - | - | K | D | S | L | N | T | N | S | S | F | N | V | L | Y | K | V | L | F | F | P | D | F | N | - | - | - | - | F | A | W | V | K | R | N | - | - | S | V | K | P | L | - | L | D | S | E | I | A | K | F | L | G | - | - | - | - | - | S | S | K | R | K | S | K | E | L | I | E | A | Y | E | A | S | K | T | P | P | D | L | K | E | E | S | S | T | D | L | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | P | N | R | K | Y | A | D | G | E | V | V | M | G | R | W | P | - | - | - | G | S | - | V | L | Y | Y | E | V | Q | V | T | S | Y | D | D | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | H | L | Y | T | V | K | Y | K | - | D | G | T | - | - | - | - | E | L | A | L | K | E | S | - | - | D | I | R | L | Q | - | S | S | F | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | L | D | - | L | V | - | N | M | T | Q | H | Y | E | N | S | V | P | E | T | V | H | V | G | D | I | V | A | A | P | L | P | T | N | - | - | - | - | G | S | W | Y | R | A | R | V | L | G | T | L | E | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | N | L | D | L | Y | F | V | D | F | G | - | - | - | - | - | N | G | D | C | P | L | - | - | - | D | L | R | A | L | R | - | - | - | S | D | F | L | - | S | L | P | F | Q | A | I | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | H | Y | K | Q | I | V | W | V | L | G | - | - | - | - | - | - | N | Y | R | W | W | P | A | E | I | C | N | P | S | V | P | - | - | - | - | - | - | - | - | - | - | N | I | Q | G | L | H | D | L | G | D | F | P | V | F | F | F | G | S | H | D | - | - | - | - | - | Y | W | V | H | Q | G | - | - | R | V | F | P | Y | V | E | G | F | A | E | G | Q | T | S | I | N | T | F | A | L | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | A | A | K | R | F | Q | E | L | K | A | Q | R | E | S | E | A | L | E | I |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | Y | K | A | G | D | L | V | F | A | K | M | K | - | - | - | G | - | - | Y | P | H | W | P | A | R | I | D | E | L | P | E | G | A | V | K | P | P | A | - | - | - | - | - | - | - | - | - | - | - | - | N | K | Y | P | I | F | F | F | G | T | H | E | - | - | - | - | T | A | F | L | G | P | K | - | - | D | L | F | P | - | - | Y | K | E | Y | K | D | K | F | G | K | - | - | - | - | - | - | - | S | N | K | R | K | G | F | N | E | G | L | W | E | I | E | N | N | P | G | V | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| Member | Chain | UniProtKB | GO term |
|---|---|---|---|
| d1mhna_ | A | Q16637 | GO:0003723 |
| d1mhna_ | A | Q16637 | GO:0005634 |
| d1mhna_ | A | Q16637 | GO:0005737 |
| d1mhna_ | A | Q16637 | GO:0006397 |
| d1mhna_ | A | Q16637 | GO:0003723 |
| d1mhna_ | A | Q16637 | GO:0005634 |
| d1mhna_ | A | Q16637 | GO:0005737 |
| d1mhna_ | A | Q16637 | GO:0006397 |
| d1oz2a1 | A | Q9Y468 | GO:0005634 |
| d1oz2a1 | A | Q9Y468 | GO:0006355 |
| d1oz2a1 | A | Q9Y468 | GO:0005634 |
| d1oz2a1 | A | Q9Y468 | GO:0006355 |
| d1oz2a1 | A | Q9Y468 | GO:0005634 |
| d1oz2a1 | A | Q9Y468 | GO:0006355 |
| d1oz2a1 | A | Q9Y468 | GO:0005634 |
| d1oz2a1 | A | Q9Y468 | GO:0006355 |
| d1oz2a2 | A | Q9Y468 | GO:0005634 |
| d1oz2a2 | A | Q9Y468 | GO:0006355 |
| d1oz2a2 | A | Q9Y468 | GO:0005634 |
| d1oz2a2 | A | Q9Y468 | GO:0006355 |
| d1oz2a2 | A | Q9Y468 | GO:0005634 |
| d1oz2a2 | A | Q9Y468 | GO:0006355 |
| d1oz2a2 | A | Q9Y468 | GO:0005634 |
| d1oz2a2 | A | Q9Y468 | GO:0006355 |
