solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1a0ia2 ( 2 )
d1ckma2 ( 11 ) ni
d1fvia2 ( 2 )
d1p16a2 ( 2 ) vq
d1s68a- ( 1 )
d1x9na3 ( 534 )
d1xdna- ( 52 )
d1xk5a- ( 97 )
d3jsla- ( 2 ) adlssrVneLhdlLnqysyeyyvednpsvpdseydkLlheLikieeehpe
d7k72a- ( 10 ) apevlrqwqalAeVreHqfryyvrDapiisdaefdellrrLealEq-hpe
60 70 80 90 100
d1a0ia2 ( 2 ) vnikTnPFkAv
d1ckma2 ( 13 ) tteravltlnglqikLhkVvgeSrddIvakMkdlAmddhkfprLPGpnpv
d1fvia2 ( 2 ) aItkPLlAa
d1p16a2 ( 4 ) leereiP-----vipgnkldeeetkelrl-VAelLgrr--ntgfPGsqpv
d1s68a- ( 1 ) mfkkysslen
d1x9na3 ( 534 ) lspgiplkplahpT
d1xdna- ( 52 ) qsdfSpyieidl
d1xk5a- ( 97 ) hyanqLMlse
d3jsla- ( 52 ) yktv----------------dSPtvrvgg-eaqAsfnkvnHdtpMlslgn
d7k72a- ( 61 ) lrtp----------------dSPTqlVggagfatdfepvdHlerMlsldn
bb
110 120 130 140 150
d1a0ia2 ( 13 ) sfvesaIkkaldnAg-------yLIAEIkydgvrGNICVdn----tAnSy
d1ckma2 ( 63 ) sierkdfeklkq---------nkYvVSEkTdGirFMMFFtrVfg-fkvCT
d1fvia2 ( 11 ) tLe--niedVq---f-------pClAtpkIaGirSVkq-------t---q
d1p16a2 ( 47 ) sferrHleetlq---------kdYfVcekTdGlrCLLFLindpdkgegVf
d1s68a- ( 11 ) hynskfiekLyslgltg----geWVAREkIhGtNFSLIIer-----dkvt
d1x9na3 ( 549 ) rgIseVlkrfee---------aaFTCeYkYdGqrAQIHal--egg--evk
d1xdna- ( 64 ) p-sesriqsLhksgLAa----qeWVAcEkVhGtNFGIYLInqgdh-evvr
d1xk5a- ( 107 ) wli-dvpsdlgq----------eWiVVVCPvgkrALIvAsr-----gsTs
d3jsla- ( 85 ) AfneddLrkFDqrIreqIg-nVeYMCeLkIdGLaVSLkYvd----GyFvq
d7k72a- ( 95 ) AftadeLaawagrIhaeVgdaAhYLCELkIdGvaLSLvYre----grLtr
b aaaaaaaaa bbbbbb bbbbbbbb bbb
160 170 180 190 200
d1a0ia2 ( 52 ) Wlsr----vsktIpALehlngfdvrWkrLLnddrCf------ykd--GFM
d1ckma2 ( 103 ) IIdraMtVylLpFknIpr-----------------v------Lfq--GSI
d1fvia2 ( 39 ) Mlsr----tfkpIrn--------svMnrLLt-elLp------------eg
d1p16a2 ( 89 ) LVTrendYyfIpnIhFPlsv--------netrekpt------yHh--gTL
d1s68a- ( 52 ) CAkrtgpIlpaedffgyeiI--lknyadsIkaVqdiMets----avvSyQ
d1x9na3 ( 586 ) IFsrnqedntg---kYpdIi------sr-ipkIklp------sVt--SFI
d1xdna- ( 108 ) fAkrsgi-dpnenffGyHil--ideFtaQIriLndlLkqkyglsrVGRLV
d1xk5a- ( 141 ) AYtksGycvnrfsSlLPGGN------rrnsta-----------kd--yTi
d3jsla- ( 130 ) GLtr--gdGttGedIteNLk-TIhAIplkMke-----------pl--nVE
d7k72a- ( 141 ) ASTr--gdGrtGedVtlNAr-tIadVperLtpgdyp------vPe--vLE
bb bb
210 220 230 240 250
d1a0ia2 ( 90 ) LDGeLmVk-gvdfntgSglLr-tk----wTdtkNqefhrkkdkvpFkLht
d1ckma2 ( 128 ) FDGeLCVD-iv--------------------------------------e
d1fvia2 ( 64 ) SDGeIsie-gatfqdTtsaVm-tg----ha--------------------
d1p16a2 ( 123 ) LDGeLVle-nrn-----------------------------------vse
d1s68a- ( 96 ) VFGeFAGp-------------------------------gIqk--nVdYc
d1x9na3 ( 618 ) LDTeAVAw-dkk--------------------qiqpfqvLttrkdaseiq
d1xdna- ( 156 ) LNGeLFGAkYkhplVpkSekwCtLpngkkfpIag----vqIqrepfPqYS
d1xk5a- ( 172 ) LDCiyN---evn--------------------------------------
d3jsla- ( 164 ) VrGeAyMp-rrsflrlneeke-kndeqlfanprnAAagSLrqldSklTak
d7k72a- ( 181 ) VrGeVfFr-lddfqalnaslv-e-gkapfanpRNSAAgSLRqkdpavTar
bbbbbb
260 270 280 290 300
d1a0ia2 ( 142 ) ghLhIkLyAILp--lhiVesgedCdvmtllMqehVknMlpLLqeyfp---
d1ckma2 ( 139 ) kkfAFVLfdAVVVsg-----vtVsqmdlasRffaMkrsLkeFk-------
d1fvia2 ( 92 ) -kFSYyWfDyVt--d-------dplkkYidRVedMknyitvhphileha-
d1p16a2 ( 137 ) pvlrYvIfdALAIhg-----kcIidrpLpkRlgyItenVkPFdnfkkhnp
d1s68a- ( 113 ) -dkdFYVfDIIvtte--sgdvty--vdDymMesfCntf------------
d1x9na3 ( 653 ) vqVCLYAfDLIylng-----eslvrepLsrRrqlLrenFvet--------
d1xdna- ( 202 ) peLhFFAfDIkYSvsgaeedfvl--LgydeFveFSskVp-----------
d1xk5a- ( 181 ) --qTYYVLDVMCWrG-----hpfydcqTdfRFywMhskLpeeegLge---
d3jsla- ( 212 ) rkLsVfIysVndft--------------dFnArsQseALdeLdklg----
d7k72a- ( 229 ) rrLrMiChglGhve--------------gfrpatLhqAylALraWg----
bbbbbbbbbb aaaaaaaaaaa
310 320 330 340 350
d1a0ia2 ( 187 ) --------eIeWqaAesyeVy----------dmveLqqlyeqKrae----
d1ckma2 ( 177 ) ---nvpeDpAiLryKewip--------------lehptiikdhlk-kan-
d1fvia2 ( 131 ) --------qVkIipliPveIn----------niteLlqyerdVlsk----
d1p16a2 ( 183 ) diVnspeFPFkvgfkt-lt--------------syha---ddvls-kdk-
d1s68a- ( 146 ) ---------kFkMAPLLgrgkFeeliklpndLdSvVqdYnftvdhaglvd
d1x9na3 ( 690 ) --------egeFvfAtsld--------------tkdieqIaflqS-vkd-
d1xdna- ( 239 ) ---------nLlyaralvrgtLdeClafdvenfTpLpallglg-------
d1xk5a- ( 221 ) ---ktkLNpfkFvgLknfp--------------Ct-pesLcdVLs-md--
d3jsla- ( 244 ) ----------FtTnknrarvn----------nIdgVleyiekWtsqres-
d7k72a- ( 261 ) ----------LpVsehttlat----------dlagVrerIdyWgehrhe-
bb bb aaaaaaaa
360 370 380 390 400
d1a0ia2 ( 215 ) ---------------gheGLiVKdP------mciYkRgkk----sgWWkm
d1ckma2 ( 208 ) ------------aiyhTdgliImsv------depViyg----rnfnLFkl
d1fvia2 ( 159 ) ---------------gfeGVmIRkp------dGkYkfgrStlkegIlLkM
d1p16a2 ( 215 ) ------------lfhasdGLiytCA------etpYvfg----tdqtlLkw
d1s68a- ( 187 ) ANkcvwnAeakgevftAeGYvLKpCypswLr----------ngnRVAIkC
d1x9na3 ( 718 ) ---------------sCeGLVktld------vdAtYeiak--rshnwLkL
d1xdna- ( 274 ) ----nyple----gnlAEGVvIrHvrrgdpave-------khnvsTIIkl
d1xk5a- ( 250 ) ------------FpfevdGLLFYhk------qthYspg----stplVGwL
d3jsla- ( 273 ) ------------lpydIdGIvIKVNdldqQdeMgft----qksPrwAiAy
d7k72a- ( 290 ) ------------VdheIdGVvVkVDevalqrrLgst----srAprwAIAy
bbbbbb bbbb
410 420
d1a0ia2 ( 240 ) k
d1ckma2 ( 236 ) kpg
d1fvia2 ( 188 ) kq
d1p16a2 ( 243 ) kpa
d1s68a- ( 227 ) knskfse
d1x9na3 ( 746 ) kkdyldgv
d1xdna- ( 309 ) rCssfel
d1xk5a- ( 278 ) rpymVsdvLgvavpagplttkpd
d3jsla- ( 307 ) kFp
d7k72a- ( 324 ) kyp
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| ATP-dependent DNA ligase, N-terminal domain | ATP-dependent DNA ligase catalytic domain | Bacteriophage T7 [TaxId: 10760] |  | view | |
| RNA guanylyltransferase (mRNA capping enzyme), N-terminal domain | mRNA capping enzyme | Chlorella virus PBCV-1 [TaxId: 10506] |  | view | |
| ATP-dependent DNA ligase, N-terminal domain | ATP-dependent DNA ligase catalytic domain | Chlorella virus PBCV-1 [TaxId: 10506] |  | view | |
| mRNA capping enzyme alpha subunit | mRNA capping enzyme | Yeast (Candida albicans) [TaxId: 5476] |  | view | |
| RNA ligase 2, N-terminal domain | RNA ligase | Bacteriophage T4 [TaxId: 10665] | 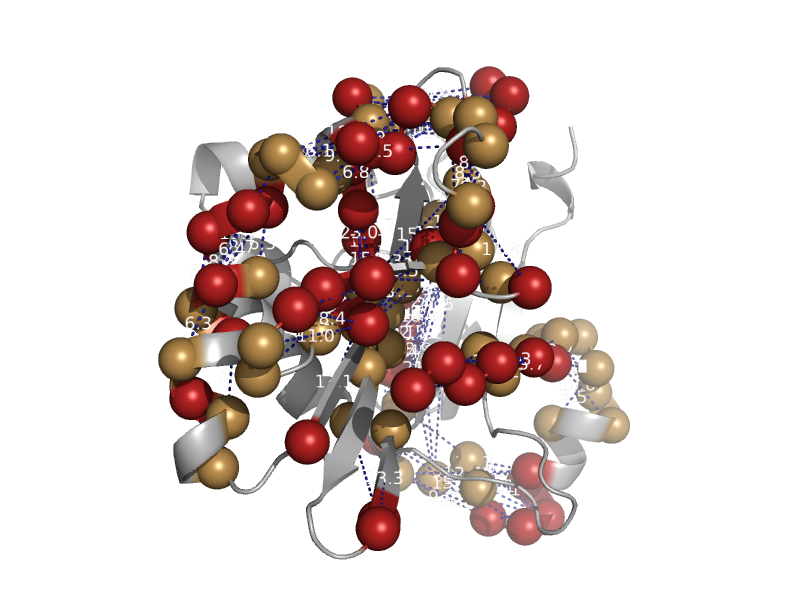 | view | |
| DNA ligase I (LIG1) | ATP-dependent DNA ligase catalytic domain | Human (Homo sapiens) [TaxId: 9606] | 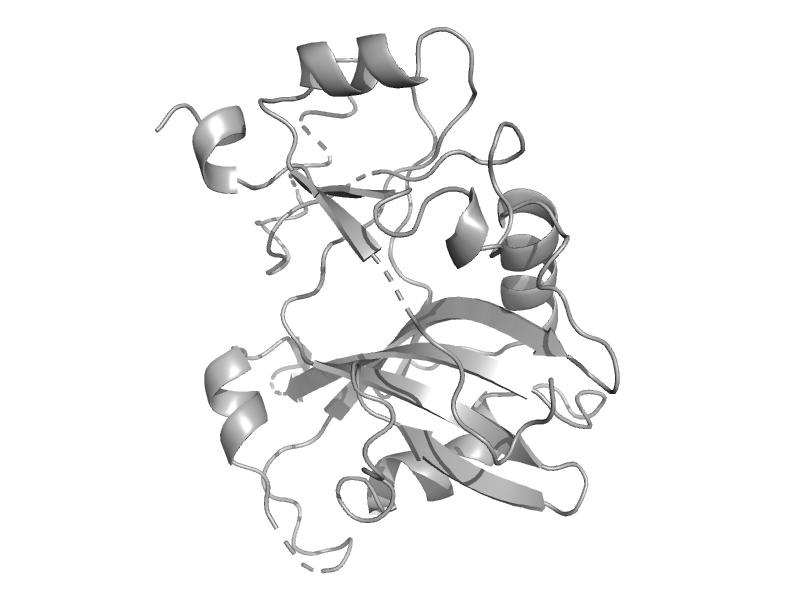 | view | |
| RNA editing ligase MP52 | RNA ligase | Trypanosome (Trypanosoma brucei) [TaxId: 5691] | 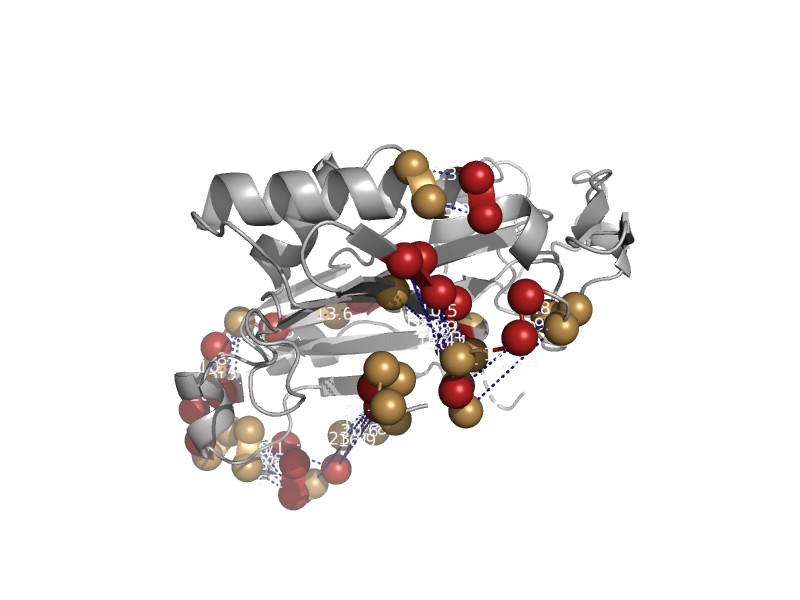 | view | |
| m3G-cap binding domain of snurportin-1 | m3G-cap binding domain of snurportin-1 | Human (Homo sapiens) [TaxId: 9606] | 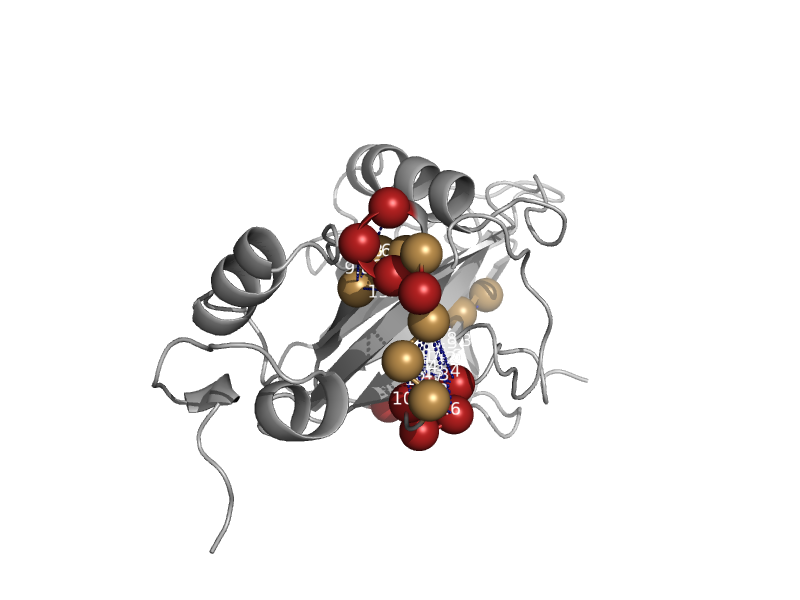 | view | |
| Adenylation domain of NAD+-dependent DNA ligase | Adenylation domain of NAD+-dependent DNA ligase | Staphylococcus aureus [TaxId: 1280] | 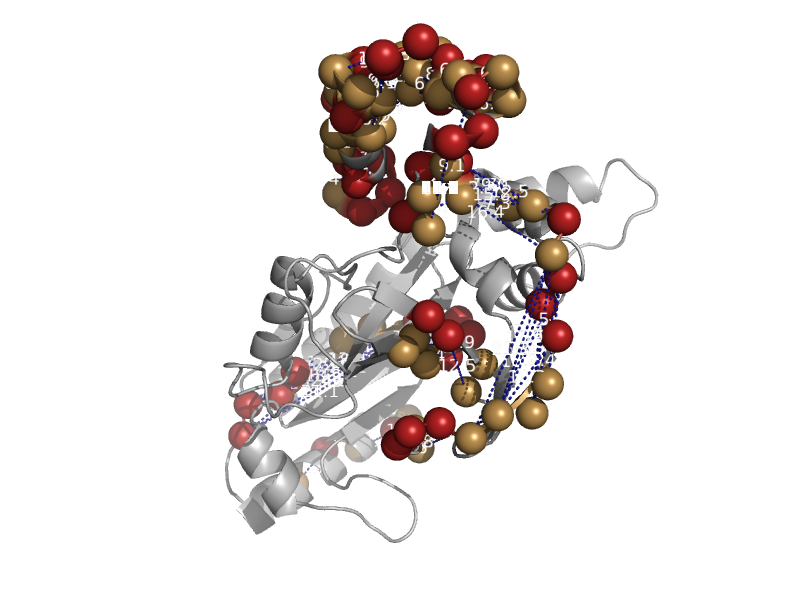 | view | |
| automated matches | automated matches | Mycobacterium tuberculosis [TaxId: 83332] | 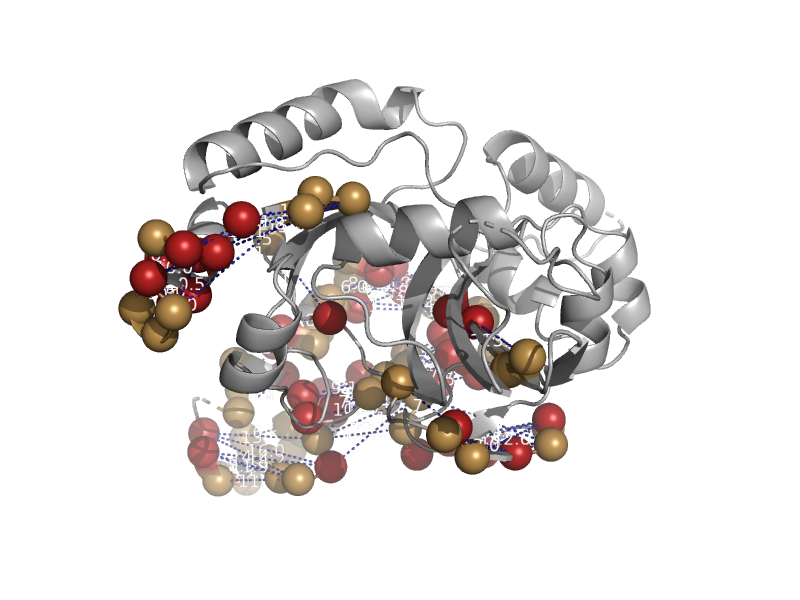 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | 230 | 240 | 250 | 260 | 270 | 280 | 290 | 300 | 310 | 320 | 330 | 340 | 350 | 360 | 370 | 380 | 390 | 400 | 410 | 420 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | V | N | I | K | T | N | P | F | K | A | V | S | F | V | E | S | A | I | K | K | A | L | D | N | A | G | - | - | - | - | - | - | - | Y | L | I | A | E | I | K | Y | D | G | V | R | G | N | I | C | V | D | N | - | - | - | - | T | A | N | S | Y | W | L | S | R | - | - | - | - | V | S | K | T | I | P | A | L | E | H | L | N | G | F | D | V | R | W | K | R | L | L | N | D | D | R | C | F | - | - | - | - | - | - | Y | K | D | - | - | G | F | M | L | D | G | E | L | M | V | K | - | G | V | D | F | N | T | G | S | G | L | L | R | - | T | K | - | - | - | - | W | T | D | T | K | N | Q | E | F | H | R | K | K | D | K | V | P | F | K | L | H | T | G | H | L | H | I | K | L | Y | A | I | L | P | - | - | L | H | I | V | E | S | G | E | D | C | D | V | M | T | L | L | M | Q | E | H | V | K | N | M | L | P | L | L | Q | E | Y | F | P | - | - | - | - | - | - | - | - | - | - | - | E | I | E | W | Q | A | A | E | S | Y | E | V | Y | - | - | - | - | - | - | - | - | - | - | D | M | V | E | L | Q | Q | L | Y | E | Q | K | R | A | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | H | E | G | L | I | V | K | D | P | - | - | - | - | - | - | M | C | I | Y | K | R | G | K | K | - | - | - | - | S | G | W | W | K | M | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | I | T | T | E | R | A | V | L | T | L | N | G | L | Q | I | K | L | H | K | V | V | G | E | S | R | D | D | I | V | A | K | M | K | D | L | A | M | D | D | H | K | F | P | R | L | P | G | P | N | P | V | S | I | E | R | K | D | F | E | K | L | K | Q | - | - | - | - | - | - | - | - | - | N | K | Y | V | V | S | E | K | T | D | G | I | R | F | M | M | F | F | T | R | V | F | G | - | F | K | V | C | T | I | I | D | R | A | M | T | V | Y | L | L | P | F | K | N | I | P | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | V | - | - | - | - | - | - | L | F | Q | - | - | G | S | I | F | D | G | E | L | C | V | D | - | I | V | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | K | K | F | A | F | V | L | F | D | A | V | V | V | S | G | - | - | - | - | - | V | T | V | S | Q | M | D | L | A | S | R | F | F | A | M | K | R | S | L | K | E | F | K | - | - | - | - | - | - | - | - | - | - | N | V | P | E | D | P | A | I | L | R | Y | K | E | W | I | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | E | H | P | T | I | I | K | D | H | L | K | - | K | A | N | - | - | - | - | - | - | - | - | - | - | - | - | - | A | I | Y | H | T | D | G | L | I | I | M | S | V | - | - | - | - | - | - | D | E | P | V | I | Y | G | - | - | - | - | R | N | F | N | L | F | K | L | K | P | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | I | T | K | P | L | L | A | A | T | L | E | - | - | N | I | E | D | V | Q | - | - | - | F | - | - | - | - | - | - | - | P | C | L | A | T | P | K | I | A | G | I | R | S | V | K | Q | - | - | - | - | - | - | - | T | - | - | - | Q | M | L | S | R | - | - | - | - | T | F | K | P | I | R | N | - | - | - | - | - | - | - | - | S | V | M | N | R | L | L | T | - | E | L | L | P | - | - | - | - | - | - | - | - | - | - | - | - | E | G | S | D | G | E | I | S | I | E | - | G | A | T | F | Q | D | T | T | S | A | V | M | - | T | G | - | - | - | - | H | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | F | S | Y | Y | W | F | D | Y | V | T | - | - | D | - | - | - | - | - | - | - | D | P | L | K | K | Y | I | D | R | V | E | D | M | K | N | Y | I | T | V | H | P | H | I | L | E | H | A | - | - | - | - | - | - | - | - | - | Q | V | K | I | I | P | L | I | P | V | E | I | N | - | - | - | - | - | - | - | - | - | - | N | I | T | E | L | L | Q | Y | E | R | D | V | L | S | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | F | E | G | V | M | I | R | K | P | - | - | - | - | - | - | D | G | K | Y | K | F | G | R | S | T | L | K | E | G | I | L | L | K | M | K | Q | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | V | Q | L | E | E | R | E | I | P | - | - | - | - | - | V | I | P | G | N | K | L | D | E | E | E | T | K | E | L | R | L | - | V | A | E | L | L | G | R | R | - | - | N | T | G | F | P | G | S | Q | P | V | S | F | E | R | R | H | L | E | E | T | L | Q | - | - | - | - | - | - | - | - | - | K | D | Y | F | V | C | E | K | T | D | G | L | R | C | L | L | F | L | I | N | D | P | D | K | G | E | G | V | F | L | V | T | R | E | N | D | Y | Y | F | I | P | N | I | H | F | P | L | S | V | - | - | - | - | - | - | - | - | N | E | T | R | E | K | P | T | - | - | - | - | - | - | Y | H | H | - | - | G | T | L | L | D | G | E | L | V | L | E | - | N | R | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | V | S | E | P | V | L | R | Y | V | I | F | D | A | L | A | I | H | G | - | - | - | - | - | K | C | I | I | D | R | P | L | P | K | R | L | G | Y | I | T | E | N | V | K | P | F | D | N | F | K | K | H | N | P | D | I | V | N | S | P | E | F | P | F | K | V | G | F | K | T | - | L | T | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | Y | H | A | - | - | - | D | D | V | L | S | - | K | D | K | - | - | - | - | - | - | - | - | - | - | - | - | - | L | F | H | A | S | D | G | L | I | Y | T | C | A | - | - | - | - | - | - | E | T | P | Y | V | F | G | - | - | - | - | T | D | Q | T | L | L | K | W | K | P | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | F | K | K | Y | S | S | L | E | N | H | Y | N | S | K | F | I | E | K | L | Y | S | L | G | L | T | G | - | - | - | - | G | E | W | V | A | R | E | K | I | H | G | T | N | F | S | L | I | I | E | R | - | - | - | - | - | D | K | V | T | C | A | K | R | T | G | P | I | L | P | A | E | D | F | F | G | Y | E | I | I | - | - | L | K | N | Y | A | D | S | I | K | A | V | Q | D | I | M | E | T | S | - | - | - | - | A | V | V | S | Y | Q | V | F | G | E | F | A | G | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | I | Q | K | - | - | N | V | D | Y | C | - | D | K | D | F | Y | V | F | D | I | I | V | T | T | E | - | - | S | G | D | V | T | Y | - | - | V | D | D | Y | M | M | E | S | F | C | N | T | F | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | F | K | M | A | P | L | L | G | R | G | K | F | E | E | L | I | K | L | P | N | D | L | D | S | V | V | Q | D | Y | N | F | T | V | D | H | A | G | L | V | D | A | N | K | C | V | W | N | A | E | A | K | G | E | V | F | T | A | E | G | Y | V | L | K | P | C | Y | P | S | W | L | R | - | - | - | - | - | - | - | - | - | - | N | G | N | R | V | A | I | K | C | K | N | S | K | F | S | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | S | P | G | I | P | L | K | P | L | A | H | P | T | R | G | I | S | E | V | L | K | R | F | E | E | - | - | - | - | - | - | - | - | - | A | A | F | T | C | E | Y | K | Y | D | G | Q | R | A | Q | I | H | A | L | - | - | E | G | G | - | - | E | V | K | I | F | S | R | N | Q | E | D | N | T | G | - | - | - | K | Y | P | D | I | I | - | - | - | - | - | - | S | R | - | I | P | K | I | K | L | P | - | - | - | - | - | - | S | V | T | - | - | S | F | I | L | D | T | E | A | V | A | W | - | D | K | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | I | Q | P | F | Q | V | L | T | T | R | K | D | A | S | E | I | Q | V | Q | V | C | L | Y | A | F | D | L | I | Y | L | N | G | - | - | - | - | - | E | S | L | V | R | E | P | L | S | R | R | R | Q | L | L | R | E | N | F | V | E | T | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | G | E | F | V | F | A | T | S | L | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | T | K | D | I | E | Q | I | A | F | L | Q | S | - | V | K | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | C | E | G | L | V | K | T | L | D | - | - | - | - | - | - | V | D | A | T | Y | E | I | A | K | - | - | R | S | H | N | W | L | K | L | K | K | D | Y | L | D | G | V | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | S | D | F | S | P | Y | I | E | I | D | L | P | - | S | E | S | R | I | Q | S | L | H | K | S | G | L | A | A | - | - | - | - | Q | E | W | V | A | C | E | K | V | H | G | T | N | F | G | I | Y | L | I | N | Q | G | D | H | - | E | V | V | R | F | A | K | R | S | G | I | - | D | P | N | E | N | F | F | G | Y | H | I | L | - | - | I | D | E | F | T | A | Q | I | R | I | L | N | D | L | L | K | Q | K | Y | G | L | S | R | V | G | R | L | V | L | N | G | E | L | F | G | A | K | Y | K | H | P | L | V | P | K | S | E | K | W | C | T | L | P | N | G | K | K | F | P | I | A | G | - | - | - | - | V | Q | I | Q | R | E | P | F | P | Q | Y | S | P | E | L | H | F | F | A | F | D | I | K | Y | S | V | S | G | A | E | E | D | F | V | L | - | - | L | G | Y | D | E | F | V | E | F | S | S | K | V | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | L | L | Y | A | R | A | L | V | R | G | T | L | D | E | C | L | A | F | D | V | E | N | F | T | P | L | P | A | L | L | G | L | G | - | - | - | - | - | - | - | - | - | - | - | N | Y | P | L | E | - | - | - | - | G | N | L | A | E | G | V | V | I | R | H | V | R | R | G | D | P | A | V | E | - | - | - | - | - | - | - | K | H | N | V | S | T | I | I | K | L | R | C | S | S | F | E | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | H | Y | A | N | Q | L | M | L | S | E | W | L | I | - | D | V | P | S | D | L | G | Q | - | - | - | - | - | - | - | - | - | - | E | W | I | V | V | V | C | P | V | G | K | R | A | L | I | V | A | S | R | - | - | - | - | - | G | S | T | S | A | Y | T | K | S | G | Y | C | V | N | R | F | S | S | L | L | P | G | G | N | - | - | - | - | - | - | R | R | N | S | T | A | - | - | - | - | - | - | - | - | - | - | - | K | D | - | - | Y | T | I | L | D | C | I | Y | N | - | - | - | E | V | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | T | Y | Y | V | L | D | V | M | C | W | R | G | - | - | - | - | - | H | P | F | Y | D | C | Q | T | D | F | R | F | Y | W | M | H | S | K | L | P | E | E | E | G | L | G | E | - | - | - | - | - | - | K | T | K | L | N | P | F | K | F | V | G | L | K | N | F | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | C | T | - | P | E | S | L | C | D | V | L | S | - | M | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | F | P | F | E | V | D | G | L | L | F | Y | H | K | - | - | - | - | - | - | Q | T | H | Y | S | P | G | - | - | - | - | S | T | P | L | V | G | W | L | R | P | Y | M | V | S | D | V | L | G | V | A | V | P | A | G | P | L | T | T | K | P | D |
| A | D | L | S | S | R | V | N | E | L | H | D | L | L | N | Q | Y | S | Y | E | Y | Y | V | E | D | N | P | S | V | P | D | S | E | Y | D | K | L | L | H | E | L | I | K | I | E | E | E | H | P | E | Y | K | T | V | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | S | P | T | V | R | V | G | G | - | E | A | Q | A | S | F | N | K | V | N | H | D | T | P | M | L | S | L | G | N | A | F | N | E | D | D | L | R | K | F | D | Q | R | I | R | E | Q | I | G | - | N | V | E | Y | M | C | E | L | K | I | D | G | L | A | V | S | L | K | Y | V | D | - | - | - | - | G | Y | F | V | Q | G | L | T | R | - | - | G | D | G | T | T | G | E | D | I | T | E | N | L | K | - | T | I | H | A | I | P | L | K | M | K | E | - | - | - | - | - | - | - | - | - | - | - | P | L | - | - | N | V | E | V | R | G | E | A | Y | M | P | - | R | R | S | F | L | R | L | N | E | E | K | E | - | K | N | D | E | Q | L | F | A | N | P | R | N | A | A | A | G | S | L | R | Q | L | D | S | K | L | T | A | K | R | K | L | S | V | F | I | Y | S | V | N | D | F | T | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | F | N | A | R | S | Q | S | E | A | L | D | E | L | D | K | L | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | F | T | T | N | K | N | R | A | R | V | N | - | - | - | - | - | - | - | - | - | - | N | I | D | G | V | L | E | Y | I | E | K | W | T | S | Q | R | E | S | - | - | - | - | - | - | - | - | - | - | - | - | - | L | P | Y | D | I | D | G | I | V | I | K | V | N | D | L | D | Q | Q | D | E | M | G | F | T | - | - | - | - | Q | K | S | P | R | W | A | I | A | Y | K | F | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| A | P | E | V | L | R | Q | W | Q | A | L | A | E | V | R | E | H | Q | F | R | Y | Y | V | R | D | A | P | I | I | S | D | A | E | F | D | E | L | L | R | R | L | E | A | L | E | Q | - | H | P | E | L | R | T | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | S | P | T | Q | L | V | G | G | A | G | F | A | T | D | F | E | P | V | D | H | L | E | R | M | L | S | L | D | N | A | F | T | A | D | E | L | A | A | W | A | G | R | I | H | A | E | V | G | D | A | A | H | Y | L | C | E | L | K | I | D | G | V | A | L | S | L | V | Y | R | E | - | - | - | - | G | R | L | T | R | A | S | T | R | - | - | G | D | G | R | T | G | E | D | V | T | L | N | A | R | - | T | I | A | D | V | P | E | R | L | T | P | G | D | Y | P | - | - | - | - | - | - | V | P | E | - | - | V | L | E | V | R | G | E | V | F | F | R | - | L | D | D | F | Q | A | L | N | A | S | L | V | - | E | - | G | K | A | P | F | A | N | P | R | N | S | A | A | G | S | L | R | Q | K | D | P | A | V | T | A | R | R | R | L | R | M | I | C | H | G | L | G | H | V | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | F | R | P | A | T | L | H | Q | A | Y | L | A | L | R | A | W | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | P | V | S | E | H | T | T | L | A | T | - | - | - | - | - | - | - | - | - | - | D | L | A | G | V | R | E | R | I | D | Y | W | G | E | H | R | H | E | - | - | - | - | - | - | - | - | - | - | - | - | - | V | D | H | E | I | D | G | V | V | V | K | V | D | E | V | A | L | Q | R | R | L | G | S | T | - | - | - | - | S | R | A | P | R | W | A | I | A | Y | K | Y | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
