solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1cg2a2 ( 214 )
d1fnoa3 ( 208 )
d1lfwa2 ( 187 ) qGiFtLeFsFknddtkgdyvLdkFkaGiatnvTPqvTrAtIsGpdLeaVk
d1r3na2 ( 248 )
d1vgya2 ( 181 )
d1ysja2 ( 178 )
d1z2la2 ( 213 )
d2q43a2 ( 195 )
60 70 80 90 100
d1cg2a2 ( 214 ) sGiAyVqVnItGkashagaape-lGvnAlvEA
d1fnoa3 ( 208 ) fnAAsvnikIvGnnvhpgtak--gvvnAlsla
d1lfwa2 ( 237 ) laYesfLadkeLdGsFeindesAdIvLi-GqgahAsa--PqvGknSATFL
d1r3na2 ( 248 ) AynwqkVtVhGvgahagttpwrlrkdAllMS
d1vgya2 ( 181 ) gsLsGnLtV-gkqghiay--phlAinPvhtF
d1ysja2 ( 178 ) svDrFeivIkgkn----------sidPiaAa
d1z2la2 ( 213 ) vGqrrYtVtLnGesnhagttpmgyrrdTvyAf
d2q43a2 ( 195 ) aGagvFeAv-Itgkt---------idPvvAA
bbbbbbbb aaaaa
110 120 130 140 150
d1cg2a2 ( 245 ) s-------------dLvlrTmnidd------k-----------anlFnwt
d1fnoa3 ( 239 ) a-------------rIhaevpadea------pettegy-----eGfyhla
d1lfwa2 ( 284 ) AlFLdqyaFagrDknFLhFLAevEhedfyGkkLgIfhhddlmgdLaSSPS
d1r3na2 ( 279 ) s-------------kMivaAseIAq------rhn----------GlFtcg
d1vgya2 ( 210 ) a-------------pAlleLtq-evWdeGne---------yfppTsFqis
d1ysja2 ( 207 ) g-------------qIisglq----------------------nAvvsit
d1z2la2 ( 245 ) s-------------rIchqSvekak------rmg---d-----pLvLtFg
d2q43a2 ( 225 ) s-------------sIvlsLqq-lvs--ret--------dpldskvVtVs
a aaaaaa bbbb
160 170 180 190 200
d1cg2a2 ( 267 ) iaagv---sniipasAtLnAdVrYan--dfdaamtL--erAqqkkl----
d1fnoa3 ( 265 ) skgt--------vdrAe-hyiIrDfdrkqFearkrk--eiakkVgkglp-
d1lfwa2 ( 334 ) mFdYehag------kAsLlNNVrYPqgTdpdtMikqVldkfsg-------
d1r3na2 ( 300 ) iidakpysvniIpgeVsFtLdFrhpsddvlatMlkeAaaeFdrlIkindg
d1vgya2 ( 237 ) ninggtgatnvIpgeLnVkFnFrFStesteagLkqrVhaILdkh--gV--
d1ysja2 ( 233 ) rvqag-tswnvipd-qAeeGtVrTfqkearqavpehr-rvAegiAagy--
d1z2la2 ( 268 ) kveprpntvnvVpGkTtFtIdCrhtdaavLrdFTqqLendMraiCdem--
d2q43a2 ( 251 ) kvnp---------dsitIgGtLrAf--tgftqLqqrVkevItkqAavh--
bb bbbbbbbb aaaaaa aaaaa
210 220
d1cg2a2 ( 312 ) ---pAdVvivtgrpA
d1fnoa3 ( 308 ) -dcyIelviedsyy
d1lfwa2 ( 371 ) ---iLdVtyn-------gfeep
d1r3na2 ( 350 ) gaLsyesetlqvsp
d1vgya2 ( 283 ) ---qydLqwscsgq
d1ysja2 ( 280 ) -gAqAefkwfpylp
d1z2la2 ( 316 ) -dIgidIdlwmdeep
d2q43a2 ( 296 ) ---rCnAsVn-ltpngrepmpp
bbbbbbb
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Carboxypeptidase G2 | Bacterial exopeptidase dimerisation domain | Pseudomonas sp., strain rs-16 [TaxId: 306] | 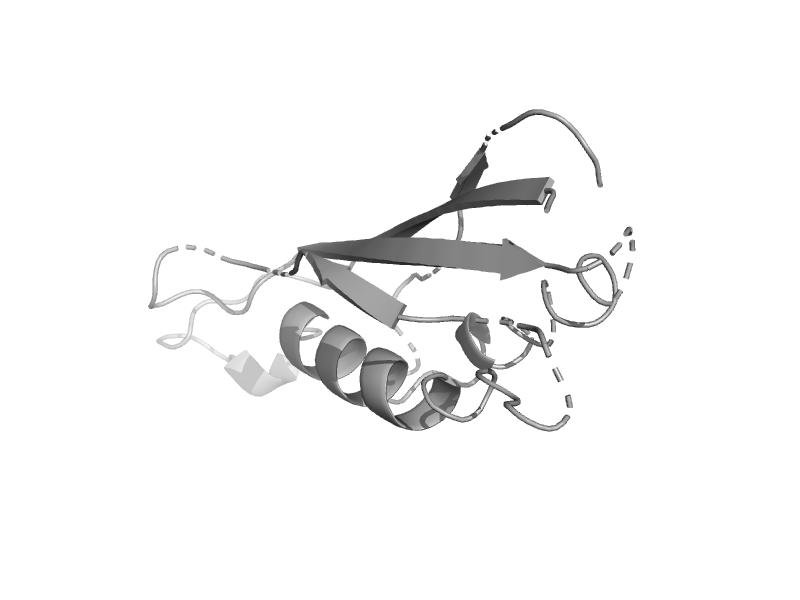 | view | |
| Peptidase T (tripeptidase) | Bacterial exopeptidase dimerisation domain | Salmonella typhimurium [TaxId: 90371] |  | view | |
| Aminopeptidase PepV | Bacterial exopeptidase dimerisation domain | Lactobacillus delbrueckii [TaxId: 1584] | 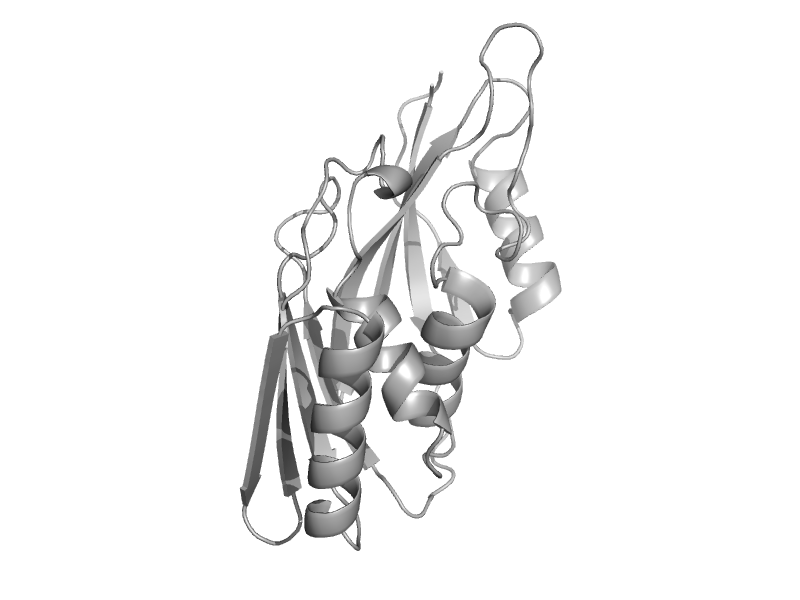 | view | |
| Peptidase-like beta-alanine synthase | Bacterial exopeptidase dimerisation domain | Yeast (Saccharomyces kluyveri) [TaxId: 4934] | 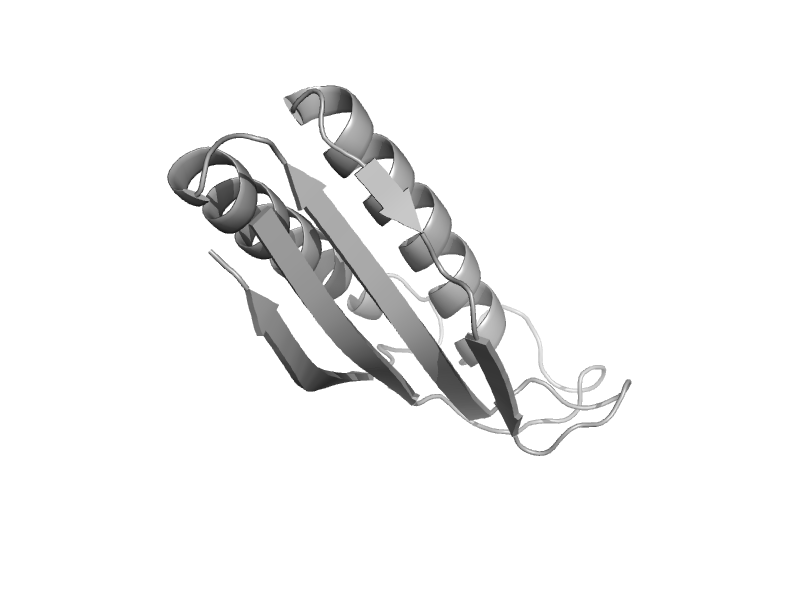 | view | |
| Succinyl-diaminopimelate desuccinylase | Bacterial exopeptidase dimerisation domain | Neisseria meningitidis [TaxId: 487] |  | view | |
| Protein YxeP | Bacterial exopeptidase dimerisation domain | Bacillus subtilis [TaxId: 1423] |  | view | |
| Allantoate amidohydrolase AllC | Bacterial exopeptidase dimerisation domain | Escherichia coli [TaxId: 562] |  | view | |
| IAA-amino acid hydrolase | Bacterial exopeptidase dimerisation domain | Thale cress (Arabidopsis thaliana) [TaxId: 3702] |  | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | G | I | A | Y | V | Q | V | N | I | T | G | K | A | S | H | A | G | A | A | P | E | - | L | G | V | N | A | L | V | E | A | S | - | - | - | - | - | - | - | - | - | - | - | - | - | D | L | V | L | R | T | M | N | I | D | D | - | - | - | - | - | - | K | - | - | - | - | - | - | - | - | - | - | - | A | N | L | F | N | W | T | I | A | A | G | V | - | - | - | S | N | I | I | P | A | S | A | T | L | N | A | D | V | R | Y | A | N | - | - | D | F | D | A | A | M | T | L | - | - | E | R | A | Q | Q | K | K | L | - | - | - | - | - | - | - | P | A | D | V | V | I | V | T | G | R | P | A | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | F | N | A | A | S | V | N | I | K | I | V | G | N | N | V | H | P | G | T | A | K | - | - | G | V | V | N | A | L | S | L | A | A | - | - | - | - | - | - | - | - | - | - | - | - | - | R | I | H | A | E | V | P | A | D | E | A | - | - | - | - | - | - | P | E | T | T | E | G | Y | - | - | - | - | - | E | G | F | Y | H | L | A | S | K | G | T | - | - | - | - | - | - | - | - | V | D | R | A | E | - | H | Y | I | I | R | D | F | D | R | K | Q | F | E | A | R | K | R | K | - | - | E | I | A | K | K | V | G | K | G | L | P | - | - | D | C | Y | I | E | L | V | I | E | D | S | Y | Y | - | - | - | - | - | - | - | - |
| Q | G | I | F | T | L | E | F | S | F | K | N | D | D | T | K | G | D | Y | V | L | D | K | F | K | A | G | I | A | T | N | V | T | P | Q | V | T | R | A | T | I | S | G | P | D | L | E | A | V | K | L | A | Y | E | S | F | L | A | D | K | E | L | D | G | S | F | E | I | N | D | E | S | A | D | I | V | L | I | - | G | Q | G | A | H | A | S | A | - | - | P | Q | V | G | K | N | S | A | T | F | L | A | L | F | L | D | Q | Y | A | F | A | G | R | D | K | N | F | L | H | F | L | A | E | V | E | H | E | D | F | Y | G | K | K | L | G | I | F | H | H | D | D | L | M | G | D | L | A | S | S | P | S | M | F | D | Y | E | H | A | G | - | - | - | - | - | - | K | A | S | L | L | N | N | V | R | Y | P | Q | G | T | D | P | D | T | M | I | K | Q | V | L | D | K | F | S | G | - | - | - | - | - | - | - | - | - | - | I | L | D | V | T | Y | N | - | - | - | - | - | - | - | G | F | E | E | P |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | Y | N | W | Q | K | V | T | V | H | G | V | G | A | H | A | G | T | T | P | W | R | L | R | K | D | A | L | L | M | S | S | - | - | - | - | - | - | - | - | - | - | - | - | - | K | M | I | V | A | A | S | E | I | A | Q | - | - | - | - | - | - | R | H | N | - | - | - | - | - | - | - | - | - | - | G | L | F | T | C | G | I | I | D | A | K | P | Y | S | V | N | I | I | P | G | E | V | S | F | T | L | D | F | R | H | P | S | D | D | V | L | A | T | M | L | K | E | A | A | A | E | F | D | R | L | I | K | I | N | D | G | G | A | L | S | Y | E | S | E | T | L | Q | V | S | P | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | S | L | S | G | N | L | T | V | - | G | K | Q | G | H | I | A | Y | - | - | P | H | L | A | I | N | P | V | H | T | F | A | - | - | - | - | - | - | - | - | - | - | - | - | - | P | A | L | L | E | L | T | Q | - | E | V | W | D | E | G | N | E | - | - | - | - | - | - | - | - | - | Y | F | P | P | T | S | F | Q | I | S | N | I | N | G | G | T | G | A | T | N | V | I | P | G | E | L | N | V | K | F | N | F | R | F | S | T | E | S | T | E | A | G | L | K | Q | R | V | H | A | I | L | D | K | H | - | - | G | V | - | - | - | - | - | Q | Y | D | L | Q | W | S | C | S | G | Q | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | V | D | R | F | E | I | V | I | K | G | K | N | - | - | - | - | - | - | - | - | - | - | S | I | D | P | I | A | A | A | G | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | I | I | S | G | L | Q | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | A | V | V | S | I | T | R | V | Q | A | G | - | T | S | W | N | V | I | P | D | - | Q | A | E | E | G | T | V | R | T | F | Q | K | E | A | R | Q | A | V | P | E | H | R | - | R | V | A | E | G | I | A | A | G | Y | - | - | - | G | A | Q | A | E | F | K | W | F | P | Y | L | P | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | V | G | Q | R | R | Y | T | V | T | L | N | G | E | S | N | H | A | G | T | T | P | M | G | Y | R | R | D | T | V | Y | A | F | S | - | - | - | - | - | - | - | - | - | - | - | - | - | R | I | C | H | Q | S | V | E | K | A | K | - | - | - | - | - | - | R | M | G | - | - | - | D | - | - | - | - | - | P | L | V | L | T | F | G | K | V | E | P | R | P | N | T | V | N | V | V | P | G | K | T | T | F | T | I | D | C | R | H | T | D | A | A | V | L | R | D | F | T | Q | Q | L | E | N | D | M | R | A | I | C | D | E | M | - | - | - | D | I | G | I | D | I | D | L | W | M | D | E | E | P | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | G | A | G | V | F | E | A | V | - | I | T | G | K | T | - | - | - | - | - | - | - | - | - | I | D | P | V | V | A | A | S | - | - | - | - | - | - | - | - | - | - | - | - | - | S | I | V | L | S | L | Q | Q | - | L | V | S | - | - | R | E | T | - | - | - | - | - | - | - | - | D | P | L | D | S | K | V | V | T | V | S | K | V | N | P | - | - | - | - | - | - | - | - | - | D | S | I | T | I | G | G | T | L | R | A | F | - | - | T | G | F | T | Q | L | Q | Q | R | V | K | E | V | I | T | K | Q | A | A | V | H | - | - | - | - | - | R | C | N | A | S | V | N | - | L | T | P | N | G | R | E | P | M | P | P |
