solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1t95a3 ( 162 ) ilplkfeeeiaIkIp-pehtgrAisaLynf-----gg-Vtreewqr--dg
d1vi7a2 ( 138 ) p-lteytLqCe-yhqltgIealLgqC-----dGkiinsdyqa---f
d2bm1a4 ( 404 ) vpepvidvaIePktkadqekLsqALarlaeedptFrvsth----petg
d2cvea2 ( 125 ) vervglaFlVp-faEvgrVyalLear-----alkA-eetytp--eG
d2dy1a4 ( 378 ) lpdpnvpvALhpkgrtdearLgeALrkLleedpsLklerq----eetg
d2dy1a5 ( 570 ) vl-lepiyrLkVlAp-qervgdVlsdLqar-----rGrilgmeqe--gal
d3b82a3 ( 482 ) kfsvspvvqVaVevknandlpkLveGlkrLsksdpcVltyms----e-sg
d3b82a5 ( 726 ) epvflVeIqCp-eqavggIysVLnkk-----rGqvvseeqrpgtpl
d6id0c3 ( 582 ) fnt-tsvikIaVePvnpselpkMldGlrkVnksypsLtTkve----e-sg
d6id0c5 ( 829 ) EpyyfVeVqAp-adcvsaVytVLarr-----rGhvtqdapipgspl
bbbbbbb aaaaaaaaaa bbbb
60 70 80 90 100
d1t95a3 ( 204 ) SWiCvripsg--ygdldllgkVAkgeAltk
d1vi7a2 ( 174 ) VlLrVALpaakvaeFsakLadfsrgsLqLlaie
d2bm1a4 ( 448 ) qtiIsGmgelhLeiivdrLkref-kVdAnvgk
d2cvea2 ( 162 ) VrFaLlLpkperegFlraLldaTrgqVale
d2dy1a4 ( 422 ) elLLwGhgelhLatAkerLqdy--gVeVefsvpkv
d2dy1a5 ( 611 ) svVhAeVPlaeVleYykaLpglTggaGaytlefshyaeVp----------
d3b82a3 ( 527 ) ehiVAGtgelhLeiClqdLehdhAgvpLkisppv
d3b82a5 ( 766 ) ftVkAyLPvnesfgFtgeLrqaTggqAfpqmvfdhwstlgsdpldp----
d6id0c3 ( 626 ) ehVilGtgelyLdcVmhdlrkmySeidIkvadpv
d6id0c5 ( 869 ) ytIkAfIPaidsfgFetdLrthTqgqAfSlsvfhhwqivpGdpldksivi
bbbbb aaaaaaaa bbb
110 120 130
d1t95a3
d1vi7a2
d2bm1a4
d2cvea2
d2dy1a4
d2dy1a5 ( 651 ) --------phlaqrivqeraqe----------------g
d3b82a3
d3b82a5 ( 812 ) --------tskaGeivlaarkrhgmkeevpgwqeyydkl
d6id0c3
d6id0c5 ( 919 ) rplepQpaphlarefmiktrrrkgl
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Hypothetical protein AF0491, C-terminal domain | Hypothetical protein AF0491, C-terminal domain | Archaeoglobus fulgidus [TaxId: 2234] |  | view | |
| Hypothetical protein YigZ, C-terminal domain | YigZ C-terminal domain-like | Escherichia coli [TaxId: 562] | 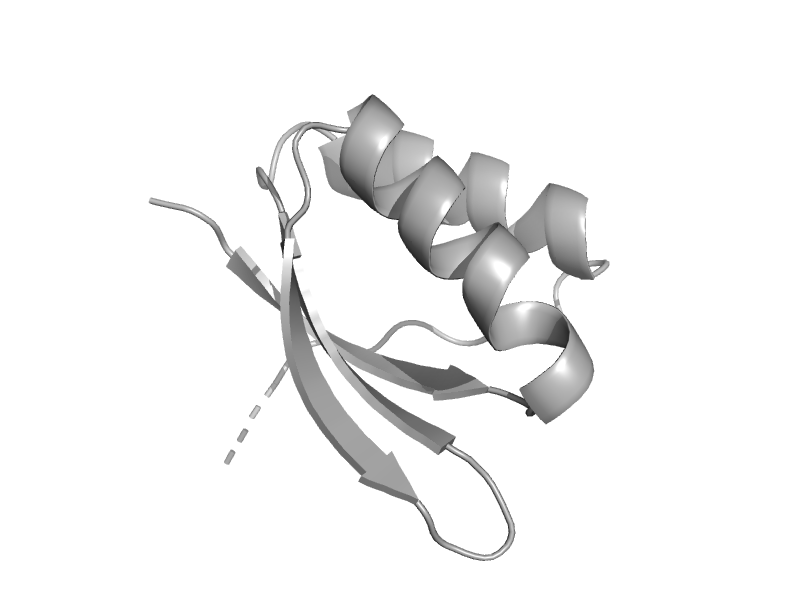 | view | |
| Elongation factor G (EF-G) | EF-G/eEF-2 domains III and V | Thermus thermophilus [TaxId: 274] | 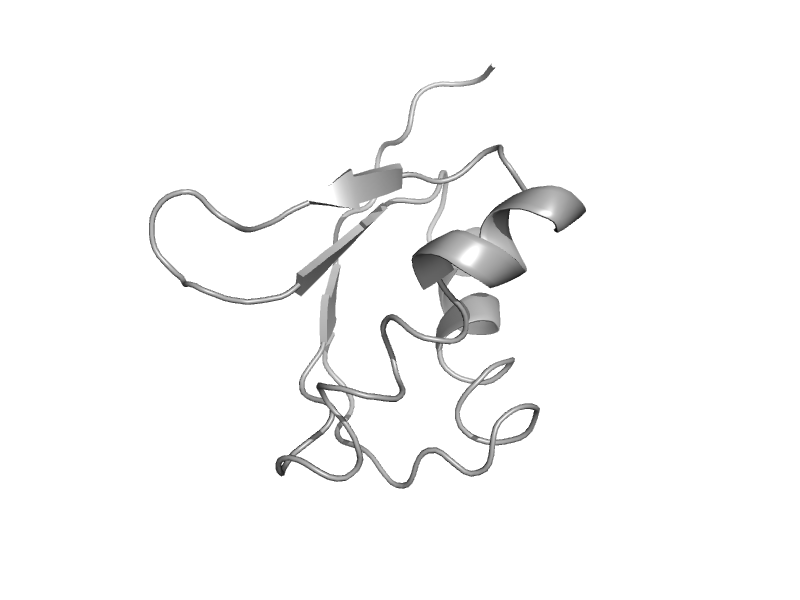 | view | |
| Hypothetical protein TTHA1053, C-terminal domain | YigZ C-terminal domain-like | Thermus thermophilus [TaxId: 274] |  | view | |
| Elongation factor G (EF-G) | EF-G/eEF-2 domains III and V | Thermus thermophilus, EF-G-2 [TaxId: 274] | 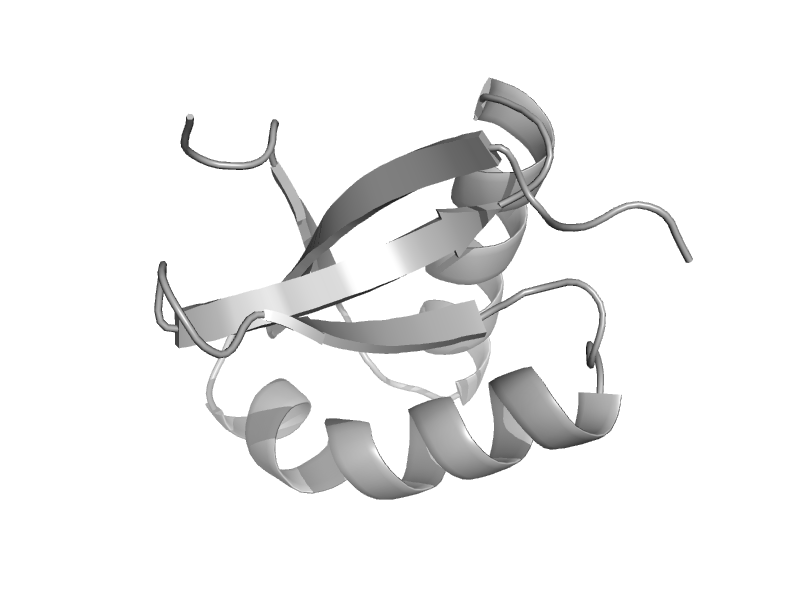 | view | |
| Elongation factor G (EF-G) | EF-G/eEF-2 domains III and V | Thermus thermophilus, EF-G-2 [TaxId: 274] | 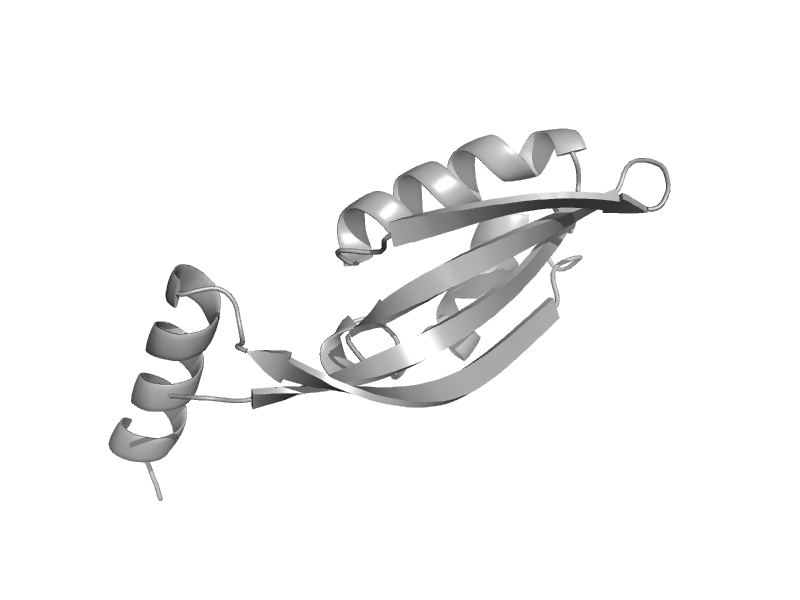 | view | |
| Elongation factor 2 (eEF-2), N-terminal domain | EF-G/eEF-2 domains III and V | Baker's yeast (Saccharomyces cerevisiae) [TaxId: 4932] | 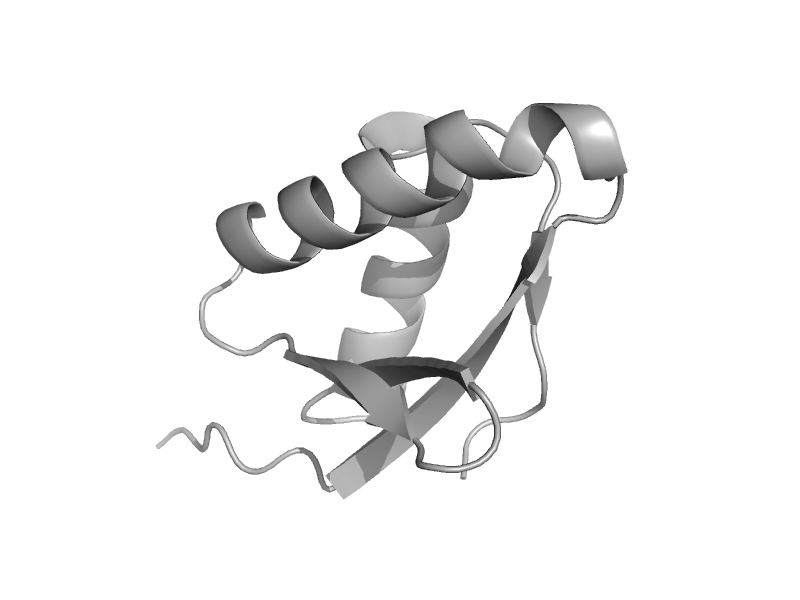 | view | |
| Elongation factor 2 (eEF-2), C-terminal domain | EF-G/eEF-2 domains III and V | Baker's yeast (Saccharomyces cerevisiae) [TaxId: 4932] | 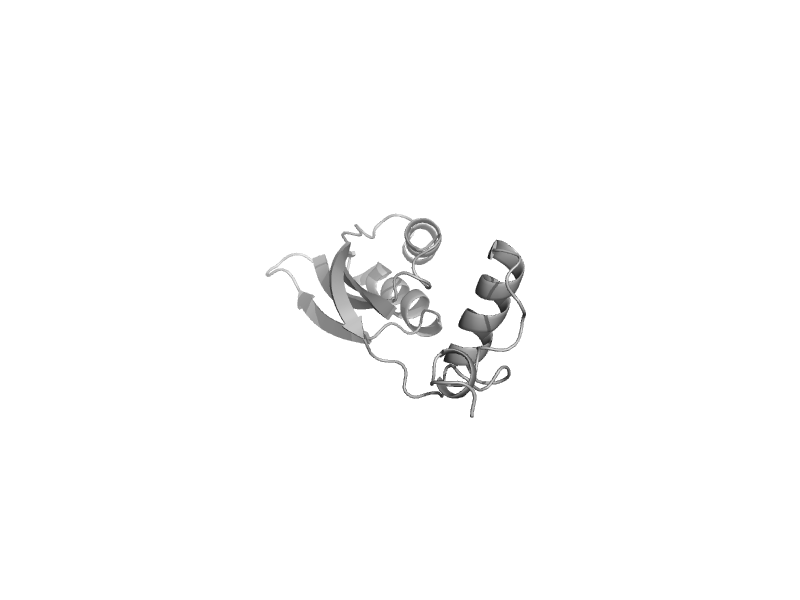 | view | |
| automated matches | automated matches | Human (Homo sapiens) [TaxId: 9606] | 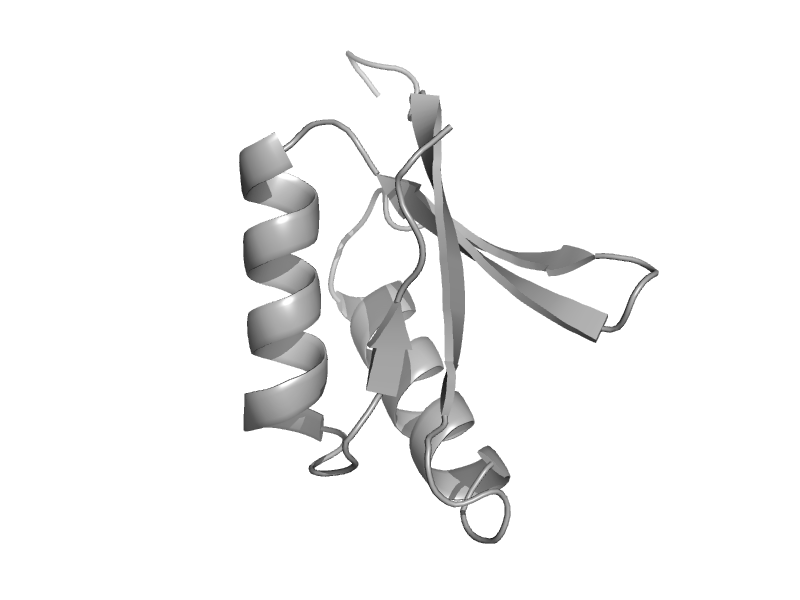 | view | |
| automated matches | automated matches | Human (Homo sapiens) [TaxId: 9606] | 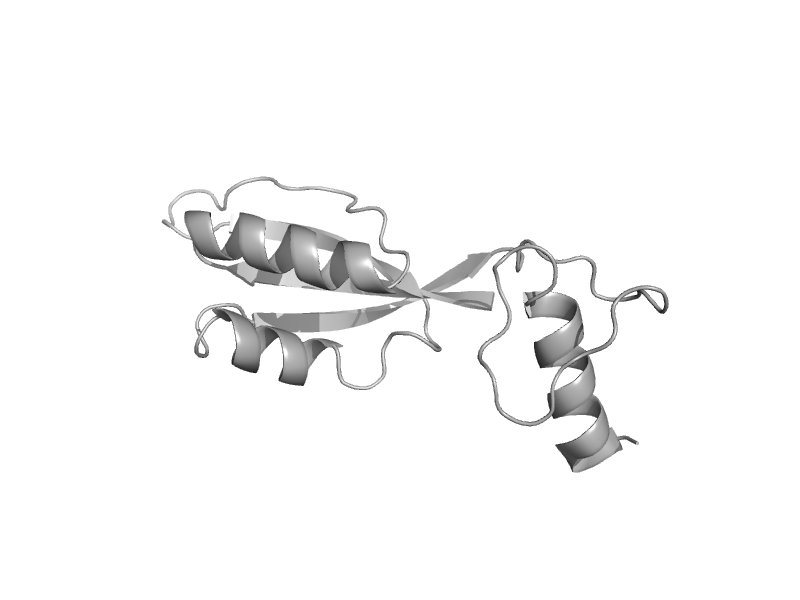 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| I | L | P | L | K | F | E | E | E | I | A | I | K | I | P | - | P | E | H | T | G | R | A | I | S | A | L | Y | N | F | - | - | - | - | - | G | G | - | V | T | R | E | E | W | Q | R | - | - | D | G | S | W | I | C | V | R | I | P | S | G | - | - | Y | G | D | L | D | L | L | G | K | V | A | K | G | E | A | L | T | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | P | - | L | T | E | Y | T | L | Q | C | E | - | Y | H | Q | L | T | G | I | E | A | L | L | G | Q | C | - | - | - | - | - | D | G | K | I | I | N | S | D | Y | Q | A | - | - | - | F | V | L | L | R | V | A | L | P | A | A | K | V | A | E | F | S | A | K | L | A | D | F | S | R | G | S | L | Q | L | L | A | I | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | V | P | E | P | V | I | D | V | A | I | E | P | K | T | K | A | D | Q | E | K | L | S | Q | A | L | A | R | L | A | E | E | D | P | T | F | R | V | S | T | H | - | - | - | - | P | E | T | G | Q | T | I | I | S | G | M | G | E | L | H | L | E | I | I | V | D | R | L | K | R | E | F | - | K | V | D | A | N | V | G | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | V | E | R | V | G | L | A | F | L | V | P | - | F | A | E | V | G | R | V | Y | A | L | L | E | A | R | - | - | - | - | - | A | L | K | A | - | E | E | T | Y | T | P | - | - | E | G | V | R | F | A | L | L | L | P | K | P | E | R | E | G | F | L | R | A | L | L | D | A | T | R | G | Q | V | A | L | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | L | P | D | P | N | V | P | V | A | L | H | P | K | G | R | T | D | E | A | R | L | G | E | A | L | R | K | L | L | E | E | D | P | S | L | K | L | E | R | Q | - | - | - | - | E | E | T | G | E | L | L | L | W | G | H | G | E | L | H | L | A | T | A | K | E | R | L | Q | D | Y | - | - | G | V | E | V | E | F | S | V | P | K | V | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| V | L | - | L | E | P | I | Y | R | L | K | V | L | A | P | - | Q | E | R | V | G | D | V | L | S | D | L | Q | A | R | - | - | - | - | - | R | G | R | I | L | G | M | E | Q | E | - | - | G | A | L | S | V | V | H | A | E | V | P | L | A | E | V | L | E | Y | Y | K | A | L | P | G | L | T | G | G | A | G | A | Y | T | L | E | F | S | H | Y | A | E | V | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | H | L | A | Q | R | I | V | Q | E | R | A | Q | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G |
| K | F | S | V | S | P | V | V | Q | V | A | V | E | V | K | N | A | N | D | L | P | K | L | V | E | G | L | K | R | L | S | K | S | D | P | C | V | L | T | Y | M | S | - | - | - | - | E | - | S | G | E | H | I | V | A | G | T | G | E | L | H | L | E | I | C | L | Q | D | L | E | H | D | H | A | G | V | P | L | K | I | S | P | P | V | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | E | P | V | F | L | V | E | I | Q | C | P | - | E | Q | A | V | G | G | I | Y | S | V | L | N | K | K | - | - | - | - | - | R | G | Q | V | V | S | E | E | Q | R | P | G | T | P | L | F | T | V | K | A | Y | L | P | V | N | E | S | F | G | F | T | G | E | L | R | Q | A | T | G | G | Q | A | F | P | Q | M | V | F | D | H | W | S | T | L | G | S | D | P | L | D | P | - | - | - | - | - | - | - | - | - | - | - | - | T | S | K | A | G | E | I | V | L | A | A | R | K | R | H | G | M | K | E | E | V | P | G | W | Q | E | Y | Y | D | K | L |
| F | N | T | - | T | S | V | I | K | I | A | V | E | P | V | N | P | S | E | L | P | K | M | L | D | G | L | R | K | V | N | K | S | Y | P | S | L | T | T | K | V | E | - | - | - | - | E | - | S | G | E | H | V | I | L | G | T | G | E | L | Y | L | D | C | V | M | H | D | L | R | K | M | Y | S | E | I | D | I | K | V | A | D | P | V | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | E | P | Y | Y | F | V | E | V | Q | A | P | - | A | D | C | V | S | A | V | Y | T | V | L | A | R | R | - | - | - | - | - | R | G | H | V | T | Q | D | A | P | I | P | G | S | P | L | Y | T | I | K | A | F | I | P | A | I | D | S | F | G | F | E | T | D | L | R | T | H | T | Q | G | Q | A | F | S | L | S | V | F | H | H | W | Q | I | V | P | G | D | P | L | D | K | S | I | V | I | R | P | L | E | P | Q | P | A | P | H | L | A | R | E | F | M | I | K | T | R | R | R | K | G | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
