solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1knxa2 ( 133 ) atvaqihGVLLeVf-----gVGVLLtgrsgigkseCAldLInk---nHlF
d1ko7a2 ( 130 ) arttslhGVLVdVy-----gVGVLItGdsgIgKsetAleLIkr---ghrL
d2olra1 ( 228 ) iAsMhCSANVgg------dVAVFFGlsgTgKttLSTdp------krrL
d3tqfa- ( 15 ) kqtwhAnFLvId-----k-GVLItgeanigkseLSlaLIdr---ghqL
d4gmua2 ( 260 ) wLaEhMLILGItnpegkkKyLAAAFPsaCgKtnLAMMnptLpgWkVeC
bbb bbbb bbbbb aaaaa bb
60 70 80 90 100
d1knxa2 ( 175 ) VGDdaIeIyrlg-nrLfGrAqevakKfMeir-------------------
d1ko7a2 ( 172 ) VADdnVeIrEiskdeLiGrApkliehlLeir-------------------
d2olra1 ( 266 ) IGDdeHGWdd-----dGVfNfeg-gCyAkTikLs--aepeIynAirrdAl
d3tqfa- ( 55 ) VÇddvIdLkqen-nqLiGsÇpsvangyIlIt-------------------
d4gmua2 ( 308 ) VGDdiAwMkFdaqGnLrAiNpen-gFfGvApgtSvktNpnAikTIqknTi
bb bbbbb bbbb bbb
110 120 130 140 150
d1knxa2 ( 205 ) -----------------------------------------------glg
d1ko7a2 ( 203 ) -----------------------------------------------glg
d2olra1 ( 310 ) lenVtvrd--gtidfddgs------------------------ktenTrV
d3tqfa- ( 85 ) -----------------------------------------------gig
d4gmua2 ( 357 ) ftnVAeTsdggvyWegideplapgvtItswknkewrpqdeepCAhpnSrF
b
160 170 180 190 200
d1knxa2 ( 208 ) iInVerfyg---lqITkqrTeIqLmVnLlsl----------------tfe
d1ko7a2 ( 206 ) iinVmtlfg---agSiltekrLrLNIhLen--------------------
d2olra1 ( 335 ) sYpiyHIdnIvkp--vskaghAtkVIFLTaDAfGVLPPVSrLtadQtqYH
d3tqfa- ( 88 ) iIdVpklfg---ldavvnqheVhLSIsLvkpekpll-----------ddp
d4gmua2 ( 407 ) cTpAsqCpiiDpawespeGVpIeGIIFGGrrpagV-PLVyEAlsWqHGVF
bbb bb bbbbbb
210 220 230 240 250
d1knxa2 ( 246 ) lgtelkkqrllgvdLsfyeipisp--------------------------
d1ko7a2 ( 246 ) ----eetlrIldTeitkktipvrp--------------------------
d2olra1 ( 383 ) FLSGFtAlaGtrgiteptptfsaCFGaaflslhptqyaevLvrmq---aa
d3tqfa- ( 125 ) lnplyrteiIlginVpkIlfpihp--------------------------
d4gmua2 ( 456 ) VGAaMRsegi---------mhDPFAmrpffgynfGkYLahWLsmahrpaA
bbb bb
260 270 280 290 300
d1knxa2 ( 270 ) --------------------------------------------------
d1ko7a2 ( 266 ) --------------------------------------------------
d2olra1 ( 433 ) g-AqAYLVNT-GwnGtgkrIs---idTraIIdAILngsLdnatftLpmFn
d3tqfa- ( 149 ) --------------------------------------------------
d4gmua2 ( 507 ) klPkIFHVNwfrkdkngkfLWpgfgeNSRVLeWMFgriegedsAkltpI-
310 320 330 340 350
d1knxa2 ( 270 ) -----------------------------grktSeiIesaVidfklkhsg
d1ko7a2 ( 266 ) -----------------------------grnvAviIevaAmnyrlnimg
d2olra1 ( 480 ) LaIPt----eLpgVdtiLdPrntyaspeqWqekAetLAklFidnFdkyt-
d3tqfa- ( 149 ) -----------------------------grnlpllIetlVrnhrlke--
d4gmua2 ( 556 ) GyvPkedaLnlkglgdVnveeLfgiskefWekEVeeIdkyLedqVnadl-
aaaaaaaaaaaaa
360
d1knxa2 ( 291 ) ynsaldfienqkailkrkk
d1ko7a2 ( 287 ) intaeefndrln
d2olra1 ( 526 ) dtpagaalvaaGPkl
d3tqfa- ( 169 ) --gydsshhfheh
d4gmua2 ( 605 ) p-yeIereLraLkqrIsqm
aaaaaaaa
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| HPr kinase HprK C-terminal domain | HPr kinase HprK C-terminal domain | Mycoplasma pneumoniae [TaxId: 2104] |  | view | |
| HPr kinase HprK C-terminal domain | HPr kinase HprK C-terminal domain | Staphylococcus xylosus [TaxId: 1288] |  | view | |
| Phosphoenolpyruvate (PEP) carboxykinase (ATP-oxaloacetate carboxy-lyase) | PEP carboxykinase C-terminal domain | Escherichia coli [TaxId: 562] |  | view | |
| automated matches | automated matches | Coxiella burnetii [TaxId: 777] |  | view | |
| Cytosolic phosphoenolpyruvate carboxykinase (GTP-hydrolyzing) | PEP carboxykinase C-terminal domain | Norway rat (Rattus norvegicus) [TaxId: 10116] | 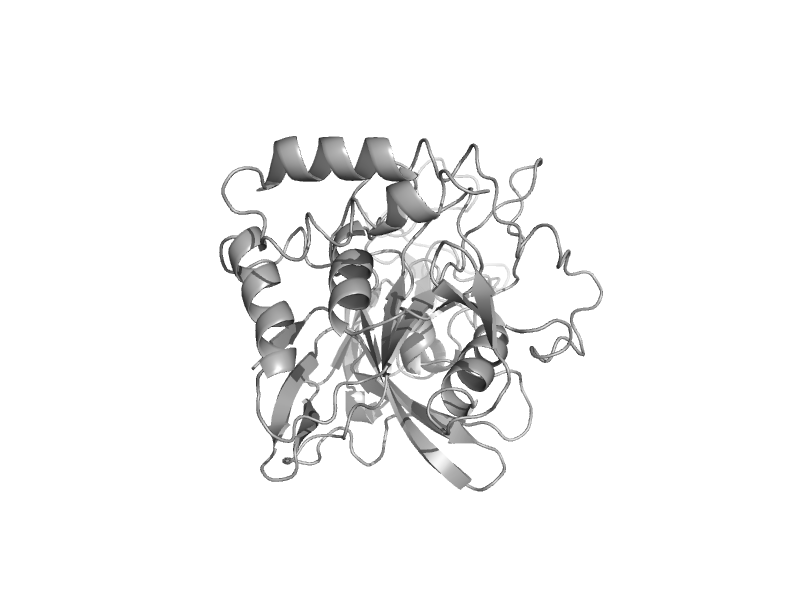 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | 230 | 240 | 250 | 260 | 270 | 280 | 290 | 300 | 310 | 320 | 330 | 340 | 350 | 360 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | T | V | A | Q | I | H | G | V | L | L | E | V | F | - | - | - | - | - | G | V | G | V | L | L | T | G | R | S | G | I | G | K | S | E | C | A | L | D | L | I | N | K | - | - | - | N | H | L | F | V | G | D | D | A | I | E | I | Y | R | L | G | - | N | R | L | F | G | R | A | Q | E | V | A | K | K | F | M | E | I | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | L | G | I | I | N | V | E | R | F | Y | G | - | - | - | L | Q | I | T | K | Q | R | T | E | I | Q | L | M | V | N | L | L | S | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | T | F | E | L | G | T | E | L | K | K | Q | R | L | L | G | V | D | L | S | F | Y | E | I | P | I | S | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | R | K | T | S | E | I | I | E | S | A | V | I | D | F | K | L | K | H | S | G | Y | N | S | A | L | D | F | I | E | N | Q | K | A | I | L | K | R | K | K |
| A | R | T | T | S | L | H | G | V | L | V | D | V | Y | - | - | - | - | - | G | V | G | V | L | I | T | G | D | S | G | I | G | K | S | E | T | A | L | E | L | I | K | R | - | - | - | G | H | R | L | V | A | D | D | N | V | E | I | R | E | I | S | K | D | E | L | I | G | R | A | P | K | L | I | E | H | L | L | E | I | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | L | G | I | I | N | V | M | T | L | F | G | - | - | - | A | G | S | I | L | T | E | K | R | L | R | L | N | I | H | L | E | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | E | T | L | R | I | L | D | T | E | I | T | K | K | T | I | P | V | R | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | R | N | V | A | V | I | I | E | V | A | A | M | N | Y | R | L | N | I | M | G | I | N | T | A | E | E | F | N | D | R | L | N | - | - | - | - | - | - | - |
| - | - | I | A | S | M | H | C | S | A | N | V | G | G | - | - | - | - | - | - | D | V | A | V | F | F | G | L | S | G | T | G | K | T | T | L | S | T | D | P | - | - | - | - | - | - | K | R | R | L | I | G | D | D | E | H | G | W | D | D | - | - | - | - | - | D | G | V | F | N | F | E | G | - | G | C | Y | A | K | T | I | K | L | S | - | - | A | E | P | E | I | Y | N | A | I | R | R | D | A | L | L | E | N | V | T | V | R | D | - | - | G | T | I | D | F | D | D | G | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | T | E | N | T | R | V | S | Y | P | I | Y | H | I | D | N | I | V | K | P | - | - | V | S | K | A | G | H | A | T | K | V | I | F | L | T | A | D | A | F | G | V | L | P | P | V | S | R | L | T | A | D | Q | T | Q | Y | H | F | L | S | G | F | T | A | L | A | G | T | R | G | I | T | E | P | T | P | T | F | S | A | C | F | G | A | A | F | L | S | L | H | P | T | Q | Y | A | E | V | L | V | R | M | Q | - | - | - | A | A | G | - | A | Q | A | Y | L | V | N | T | - | G | W | N | G | T | G | K | R | I | S | - | - | - | I | D | T | R | A | I | I | D | A | I | L | N | G | S | L | D | N | A | T | F | T | L | P | M | F | N | L | A | I | P | T | - | - | - | - | E | L | P | G | V | D | T | I | L | D | P | R | N | T | Y | A | S | P | E | Q | W | Q | E | K | A | E | T | L | A | K | L | F | I | D | N | F | D | K | Y | T | - | D | T | P | A | G | A | A | L | V | A | A | G | P | K | L | - | - | - | - |
| - | - | K | Q | T | W | H | A | N | F | L | V | I | D | - | - | - | - | - | K | - | G | V | L | I | T | G | E | A | N | I | G | K | S | E | L | S | L | A | L | I | D | R | - | - | - | G | H | Q | L | V | C | D | D | V | I | D | L | K | Q | E | N | - | N | Q | L | I | G | S | C | P | S | V | A | N | G | Y | I | L | I | T | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | I | G | I | I | D | V | P | K | L | F | G | - | - | - | L | D | A | V | V | N | Q | H | E | V | H | L | S | I | S | L | V | K | P | E | K | P | L | L | - | - | - | - | - | - | - | - | - | - | - | D | D | P | L | N | P | L | Y | R | T | E | I | I | L | G | I | N | V | P | K | I | L | F | P | I | H | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | R | N | L | P | L | L | I | E | T | L | V | R | N | H | R | L | K | E | - | - | - | - | G | Y | D | S | S | H | H | F | H | E | H | - | - | - | - | - | - |
| - | - | W | L | A | E | H | M | L | I | L | G | I | T | N | P | E | G | K | K | K | Y | L | A | A | A | F | P | S | A | C | G | K | T | N | L | A | M | M | N | P | T | L | P | G | W | K | V | E | C | V | G | D | D | I | A | W | M | K | F | D | A | Q | G | N | L | R | A | I | N | P | E | N | - | G | F | F | G | V | A | P | G | T | S | V | K | T | N | P | N | A | I | K | T | I | Q | K | N | T | I | F | T | N | V | A | E | T | S | D | G | G | V | Y | W | E | G | I | D | E | P | L | A | P | G | V | T | I | T | S | W | K | N | K | E | W | R | P | Q | D | E | E | P | C | A | H | P | N | S | R | F | C | T | P | A | S | Q | C | P | I | I | D | P | A | W | E | S | P | E | G | V | P | I | E | G | I | I | F | G | G | R | R | P | A | G | V | - | P | L | V | Y | E | A | L | S | W | Q | H | G | V | F | V | G | A | A | M | R | S | E | G | I | - | - | - | - | - | - | - | - | - | M | H | D | P | F | A | M | R | P | F | F | G | Y | N | F | G | K | Y | L | A | H | W | L | S | M | A | H | R | P | A | A | K | L | P | K | I | F | H | V | N | W | F | R | K | D | K | N | G | K | F | L | W | P | G | F | G | E | N | S | R | V | L | E | W | M | F | G | R | I | E | G | E | D | S | A | K | L | T | P | I | - | G | Y | V | P | K | E | D | A | L | N | L | K | G | L | G | D | V | N | V | E | E | L | F | G | I | S | K | E | F | W | E | K | E | V | E | E | I | D | K | Y | L | E | D | Q | V | N | A | D | L | - | P | - | Y | E | I | E | R | E | L | R | A | L | K | Q | R | I | S | Q | M |
