solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1fgha1 ( 529 ) v---dvsptsqrlqlle-------pfdkwdgkdLedLqILIkVkgkCtTd
d1l5ja2 ( 161 ) alaekltVTVFkVtgeTnTd
d1v7la- ( 1 ) mittgkVWkfgddisTd
d2b3ya1 ( 631 ) dklffwnskstyiksppffenltldlqpp--ksivdAyVLLnLgdsvtTd
d2gp4a1 ( 419 )
d2hi6a1 ( 2 )
60 70 80 90 100
d1fgha1 ( 569 ) hISaa-----------------------gpwlkfrghldnISnNLLi---
d1l5ja2 ( 181 ) dLSPa-pdawsrp-----------------------diplHAlaMLkn--
d1v7la- ( 18 ) eITPg-rynlt-k-----------------------dpkeLakiAFie--
d2b3ya1 ( 679 ) hISpagnIarnsPAAryLtnrgltpreFnsYgsRrgNdaVMarGTFan--
d2gp4a1 ( 419 ) glklLkg
d2hi6a1 ( 2 ) kfaCrAitrg
110 120 130 140 150
d1fgha1 ( 593 ) ----gAiNsenrk-ansVrnavtqefgpVPdTAryYkqhg---irWVVIG
d1l5ja2 ( 205 ) ---are----------gIePdqpgv-vGPikqIealqqkg---fpLAY-V
d1v7la- ( 41 ) ---vrp------------------------dFarnVrp-----gDVVV-A
d2b3ya1 ( 727 ) ---irLlNrFLnkqapqTihlpsgeildVfdAAerYqqag---lpLIVLA
d2gp4a1 ( 426 ) nLgrAvIkvsaVqpqhrvVeA-pAVviddqnkLdalfksgaLdrdCVVvV
d2hi6a1 ( 12 ) rAegeAlvtkeyisFlggidketGivkedceiKgesvag------rilvF
aaa bbb
160 170 180 190 200
d1fgha1 ( 635 ) denY---GegssrehSAlEPRfL------------gGrAIITksFariHe
d1l5ja2 ( 237 ) GdvV---GtgssrkSATnSVLwfMGddiphvpnkrggglCLGgkIapiFf
d1v7la- ( 58 ) gknF---GigssreSAAlALkal------------gIaGVIAesFgriFy
d2b3ya1 ( 771 ) gkeY---GagssrdWAAkGPflL------------gIkAVLAesYeriHr
d2gp4a1 ( 475 ) KGQGpkAn-gpeLhkLtplLgsLQdk---------gfkVALTDgrsg---
d2hi6a1 ( 56 ) pggk---GstvgSyvllnlrkn-----------gvAPkAiinkktetiia
aaaaaaaa bbbb aaaa
210 220 230 240 250
d1fgha1 ( 670 ) tnLKkqGLLPLtFadpadynk--IhpvDkLtIqgLkdFapgkpLtCiIkh
d1l5ja2 ( 284 ) ntMEdaGALPIeVdVs------nLnmgdvIDVyPy-------kgeVrnh-
d1v7la- ( 93 ) rnAiniGIpLLlgkTe------gLkdgdlVtVnWe-------tGeVrkg-
d2b3ya1 ( 806 ) snLVgmgVIPLeYlpgenadalgLtGqerYtIiIpenLkpqmkVqVkL--
d2gp4a1 ( 515 ) asgkVPaaihLtpEAidgGlIakVqdgdlIrVdAl-------tgeLsLlv
d2hi6a1 ( 92 ) vGAamAeiplvevrdekFfea--vktgdrvvvnAd-------eGyvelie
aaaaa bbb bbbbb bbbb
260 270 280 290 300
d1fgha1 ( 718 ) pn---gtqetIlLnHtF-netqIewfrAGSalnrmkelqqk
d1l5ja2 ( 320 ) -e---tgellatFelk--tdvLidevragGripLiigrglttkarealgl
d1v7la- ( 129 ) -d------eilmFepL--edflLeIVrEgGileyirrrgdlc--------
d2b3ya1 ( 854 ) dt---g--ktfqAvMrFdtdveltyflngGilnymirkmak
d2gp4a1 ( 558 ) sdtelatrtat----eIdlrhsryggrelfgvlrs-----nlsspetgar
d2hi6a1
aaaaa
d1fgha1
d1l5ja2 ( 364 ) phsdvfrqa
d1v7la- ( 162 ) --------i
d2b3ya1
d2gp4a1 ( 600 ) stsaidely
d2hi6a1
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Aconitase A, C-terminal domain | LeuD-like | Cow (Bos taurus) [TaxId: 9913] | 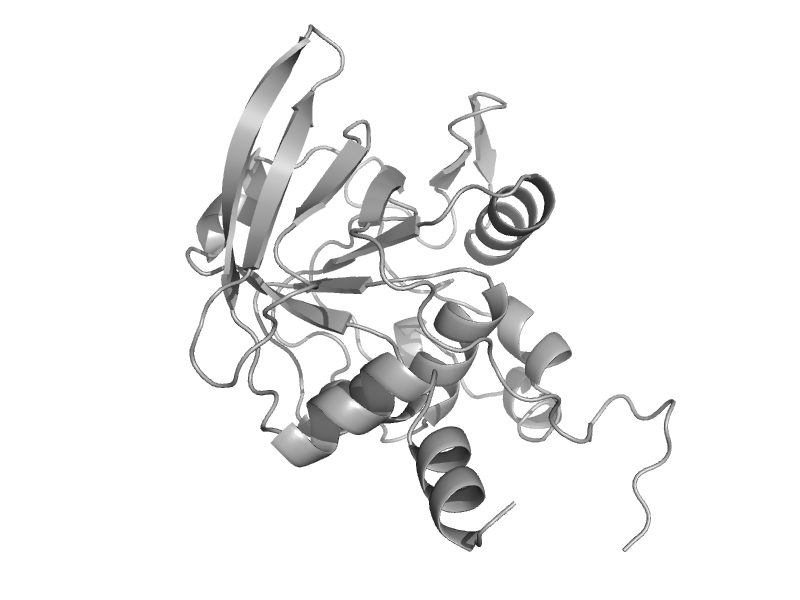 | view | |
| Aconitase B, second N-terminal domain | LeuD-like | Escherichia coli [TaxId: 562] | 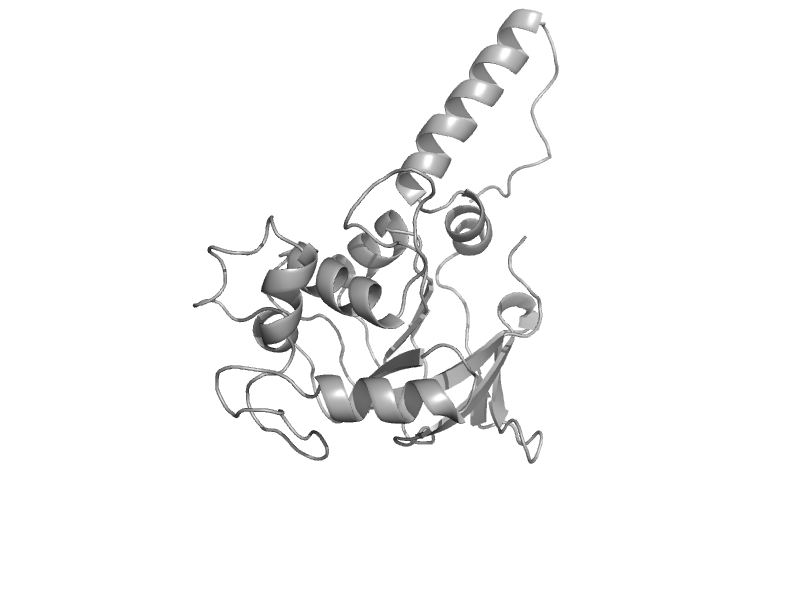 | view | |
| Isopropylmalate isomerase LeuD | LeuD-like | Pyrococcus horikoshii [TaxId: 53953] | 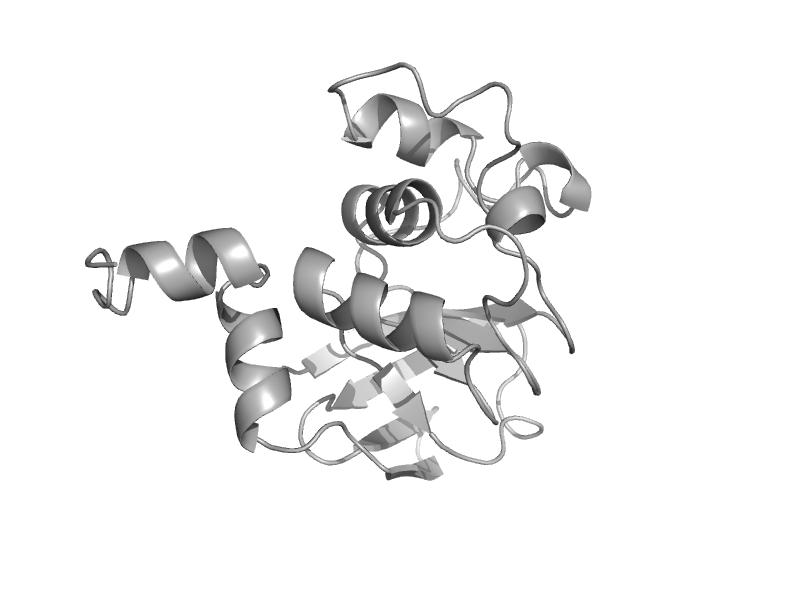 | view | |
| ron-responsive element binding protein 1, C-terminal domain | LeuD-like | Human (Homo sapiens) [TaxId: 9606] | 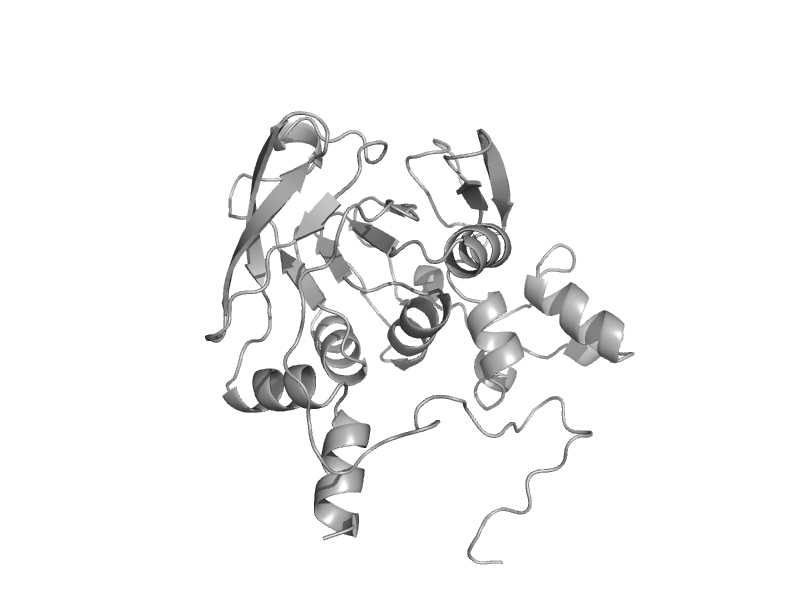 | view | |
| 6-phosphogluconate dehydratase EDD | IlvD/EDD C-terminal domain-like | Shewanella oneidensis [TaxId: 70863] | 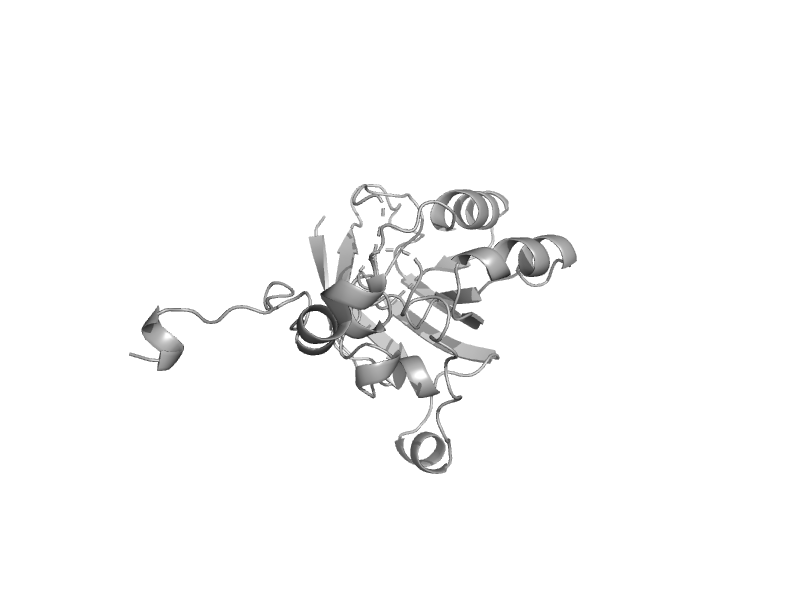 | view | |
| Hypothetical protein AF0055 | AF0055-like | Archaeoglobus fulgidus [TaxId: 2234] | 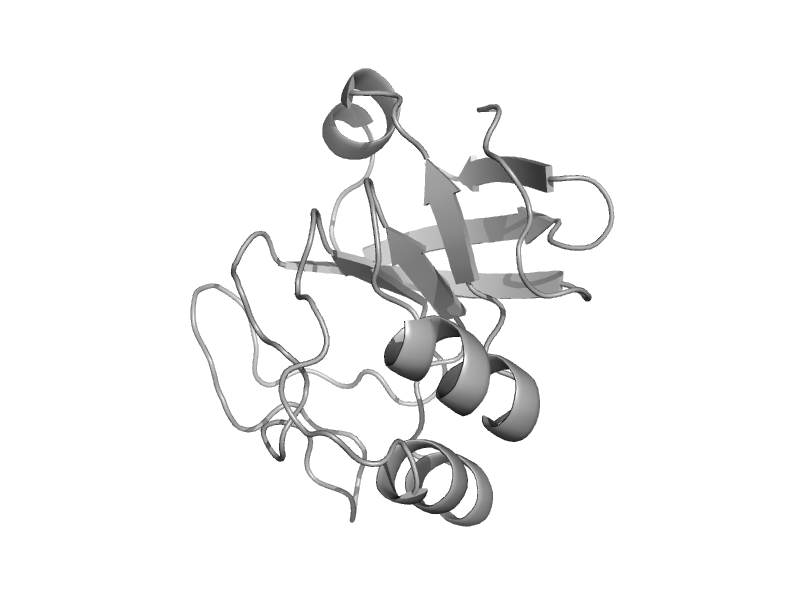 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | 230 | 240 | 250 | 260 | 270 | 280 | 290 | 300 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| V | - | - | - | D | V | S | P | T | S | Q | R | L | Q | L | L | E | - | - | - | - | - | - | - | P | F | D | K | W | D | G | K | D | L | E | D | L | Q | I | L | I | K | V | K | G | K | C | T | T | D | H | I | S | A | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | P | W | L | K | F | R | G | H | L | D | N | I | S | N | N | L | L | I | - | - | - | - | - | - | - | G | A | I | N | S | E | N | R | K | - | A | N | S | V | R | N | A | V | T | Q | E | F | G | P | V | P | D | T | A | R | Y | Y | K | Q | H | G | - | - | - | I | R | W | V | V | I | G | D | E | N | Y | - | - | - | G | E | G | S | S | R | E | H | S | A | L | E | P | R | F | L | - | - | - | - | - | - | - | - | - | - | - | - | G | G | R | A | I | I | T | K | S | F | A | R | I | H | E | T | N | L | K | K | Q | G | L | L | P | L | T | F | A | D | P | A | D | Y | N | K | - | - | I | H | P | V | D | K | L | T | I | Q | G | L | K | D | F | A | P | G | K | P | L | T | C | I | I | K | H | P | N | - | - | - | G | T | Q | E | T | I | L | L | N | H | T | F | - | N | E | T | Q | I | E | W | F | R | A | G | S | A | L | N | R | M | K | E | L | Q | Q | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | L | A | E | K | L | T | V | T | V | F | K | V | T | G | E | T | N | T | D | D | L | S | P | A | - | P | D | A | W | S | R | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | I | P | L | H | A | L | A | M | L | K | N | - | - | - | - | - | A | R | E | - | - | - | - | - | - | - | - | - | - | G | I | E | P | D | Q | P | G | V | - | V | G | P | I | K | Q | I | E | A | L | Q | Q | K | G | - | - | - | F | P | L | A | Y | - | V | G | D | V | V | - | - | - | G | T | G | S | S | R | K | S | A | T | N | S | V | L | W | F | M | G | D | D | I | P | H | V | P | N | K | R | G | G | G | L | C | L | G | G | K | I | A | P | I | F | F | N | T | M | E | D | A | G | A | L | P | I | E | V | D | V | S | - | - | - | - | - | - | N | L | N | M | G | D | V | I | D | V | Y | P | Y | - | - | - | - | - | - | - | K | G | E | V | R | N | H | - | - | E | - | - | - | T | G | E | L | L | A | T | F | E | L | K | - | - | T | D | V | L | I | D | E | V | R | A | G | G | R | I | P | L | I | I | G | R | G | L | T | T | K | A | R | E | A | L | G | L | P | H | S | D | V | F | R | Q | A |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | I | T | T | G | K | V | W | K | F | G | D | D | I | S | T | D | E | I | T | P | G | - | R | Y | N | L | T | - | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | P | K | E | L | A | K | I | A | F | I | E | - | - | - | - | - | V | R | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | F | A | R | N | V | R | P | - | - | - | - | - | G | D | V | V | V | - | A | G | K | N | F | - | - | - | G | I | G | S | S | R | E | S | A | A | L | A | L | K | A | L | - | - | - | - | - | - | - | - | - | - | - | - | G | I | A | G | V | I | A | E | S | F | G | R | I | F | Y | R | N | A | I | N | I | G | I | P | L | L | L | G | K | T | E | - | - | - | - | - | - | G | L | K | D | G | D | L | V | T | V | N | W | E | - | - | - | - | - | - | - | T | G | E | V | R | K | G | - | - | D | - | - | - | - | - | - | E | I | L | M | F | E | P | L | - | - | E | D | F | L | L | E | I | V | R | E | G | G | I | L | E | Y | I | R | R | R | G | D | L | C | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | I |
| D | K | L | F | F | W | N | S | K | S | T | Y | I | K | S | P | P | F | F | E | N | L | T | L | D | L | Q | P | P | - | - | K | S | I | V | D | A | Y | V | L | L | N | L | G | D | S | V | T | T | D | H | I | S | P | A | G | N | I | A | R | N | S | P | A | A | R | Y | L | T | N | R | G | L | T | P | R | E | F | N | S | Y | G | S | R | R | G | N | D | A | V | M | A | R | G | T | F | A | N | - | - | - | - | - | I | R | L | L | N | R | F | L | N | K | Q | A | P | Q | T | I | H | L | P | S | G | E | I | L | D | V | F | D | A | A | E | R | Y | Q | Q | A | G | - | - | - | L | P | L | I | V | L | A | G | K | E | Y | - | - | - | G | A | G | S | S | R | D | W | A | A | K | G | P | F | L | L | - | - | - | - | - | - | - | - | - | - | - | - | G | I | K | A | V | L | A | E | S | Y | E | R | I | H | R | S | N | L | V | G | M | G | V | I | P | L | E | Y | L | P | G | E | N | A | D | A | L | G | L | T | G | Q | E | R | Y | T | I | I | I | P | E | N | L | K | P | Q | M | K | V | Q | V | K | L | - | - | D | T | - | - | - | G | - | - | K | T | F | Q | A | V | M | R | F | D | T | D | V | E | L | T | Y | F | L | N | G | G | I | L | N | Y | M | I | R | K | M | A | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | L | K | L | L | K | G | N | L | G | R | A | V | I | K | V | S | A | V | Q | P | Q | H | R | V | V | E | A | - | P | A | V | V | I | D | D | Q | N | K | L | D | A | L | F | K | S | G | A | L | D | R | D | C | V | V | V | V | K | G | Q | G | P | K | A | N | - | G | P | E | L | H | K | L | T | P | L | L | G | S | L | Q | D | K | - | - | - | - | - | - | - | - | - | G | F | K | V | A | L | T | D | G | R | S | G | - | - | - | A | S | G | K | V | P | A | A | I | H | L | T | P | E | A | I | D | G | G | L | I | A | K | V | Q | D | G | D | L | I | R | V | D | A | L | - | - | - | - | - | - | - | T | G | E | L | S | L | L | V | S | D | T | E | L | A | T | R | T | A | T | - | - | - | - | E | I | D | L | R | H | S | R | Y | G | G | R | E | L | F | G | V | L | R | S | - | - | - | - | - | N | L | S | S | P | E | T | G | A | R | S | T | S | A | I | D | E | L | Y |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | F | A | C | R | A | I | T | R | G | R | A | E | G | E | A | L | V | T | K | E | Y | I | S | F | L | G | G | I | D | K | E | T | G | I | V | K | E | D | C | E | I | K | G | E | S | V | A | G | - | - | - | - | - | - | R | I | L | V | F | P | G | G | K | - | - | - | G | S | T | V | G | S | Y | V | L | L | N | L | R | K | N | - | - | - | - | - | - | - | - | - | - | - | G | V | A | P | K | A | I | I | N | K | K | T | E | T | I | I | A | V | G | A | A | M | A | E | I | P | L | V | E | V | R | D | E | K | F | F | E | A | - | - | V | K | T | G | D | R | V | V | V | N | A | D | - | - | - | - | - | - | - | E | G | Y | V | E | L | I | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
