solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1ccwb- ( 1 ) meLknkkWt---d
d1reqa1 ( 2 ) stlprfdsvdlgn----------------------apvp--------ada
d1reqb1 ( 20 ) tlslagdfpateqweevekvlnrgrp--------peq
d1uc4a- ( 1 ) mrskr
d1xrsa- ( 1 ) meskLnLdfnlVe
a
60 70 80 90 100
d1ccwb- ( 11 ) eeFhkqreeVlqqw--ptGkeVd---lqeAvdyLk------kIp------
d1reqa1 ( 22 ) arrfeelaaagtgaweTaeqIpvgtlfnedvydMd------WldtyAGi-
d1reqb1 ( 53 ) LtfaeClkr---ltvhtvdgidivpmyrpdapk----------lgypgv-
d1uc4a- ( 6 ) fealakrpvnqDgfvkewieeGfIAMeSpndpkpsIkivngaVteLDgkp
d1xrsa- ( 14 ) kArakAkaIAidtq--efIekhTTvTVERAVCrLL------gIdgvdtd-
aaaaaaaa
110 120 130 140 150
d1ccwb- ( 44 ) --aeknFAekLvlAkkkgiTMAQP--raGvAlld-----eHielLryLqd
d1reqa1 ( 68 ) ---ppFVhGPyatMYafrpWtIrqya--GfsTAk-----eSnafYrrnla
d1reqb1 ( 91 ) ---apFtrgttvrngdmdaWdvRaLh--edpd-------kfTraIlegle
d1uc4a- ( 56 ) vsdFdlIDhfIAr------------yGInlnrAeeVmamdsvkLAnmLcd
d1xrsa- ( 55 ) --evPLPniVVdhIkenngLnlGA--AMyIANAvlntgktpqeIAqaIsa
aa aaaaaaaaa
160 170 180 190 200
d1ccwb- ( 85 ) -eGgAdFLPStIDayTrqnrYdeCengikeSekag---------------
d1reqa1 ( 108 ) --agQkGLSVaFDlPTHrGydSdn---------------------prvag
d1reqb1 ( 131 ) --rgVtSLLLRVDp------------------------------------
d1uc4a- ( 94 ) pnvkrs------eIvplTtAMTPAKIveVVshMnvveMmmAMqKMRARrt
d1xrsa- ( 101 ) geldLtklpmkdlfeVktkAlsmAketvekIknnrsiresrfeeygdksg
210 220 230 240 250
d1ccwb- ( 119 ) --------------------------------------------------
d1reqa1 ( 135 ) dVGmaGVA--IDSIyDMreLFagIpLd-----------------------
d1reqb1 ( 143 ) ----dAIa-----pehLdeVLsdVlle-----------------------
d1uc4a- ( 138 ) PSQQAHVTNvkDNPVQIAADAAeGAwrgFdEQETtVav------------
d1xrsa- ( 151 ) pLLYViVa--tGnIyeDitqAvaAAkqgADVIAviRTtgQSlldyVpyga
aaaa
260 270 280 290 300
d1ccwb- ( 119 ) ------rslLnGFPGVNfGVkgCrkVLe--aVnlPLQARHGTpdSrLLAE
d1reqa1 ( 160 ) ----qmsVsMtMNGAVLPILALYVVTAeeqg-------------------
d1reqb1 ( 161 ) ----mTkVEVfSrydqgaAAeaLVsvYersd-------------------
d1uc4a- ( 176 ) ------------arYaPFNAIALLVGSQVGrpgVLTQCSleeatElkLGm
d1xrsa- ( 199 ) ttegfgGTyA-TqeNFrlMReaLdkVgaevgkYIrLCNyCsGlCMPEIAA
aaaaaaaaaaaaa
310 320 330 340 350
d1ccwb- ( 161 ) IIHAGGWTSNEGGGISYNVPy-aknvtIekSLlDWQYCDRLVGfYEeqgV
d1reqa1 ( 187 ) Vp-eqLaGtIQNDILKEFMVrnTyIYppqpSmrIISeIFayTsanMp-kW
d1reqb1 ( 188 ) kpakdLaLNLGlDpIGfAalqg--------tPdltvLGwvrrLakFs-pS
d1uc4a- ( 214 ) lgHTCYAEtIsVyGTepVFtdg--ddtPwsKgflassYas------rgLK
d1xrsa- ( 248 ) MGAierLDVMlNDALygILfr---dINMqRTMIDQnFSRIINGfA---gV
bbb aaaa aaaaaaaaaaaa
360 370 380 390 400
d1ccwb- ( 210 ) hINREPFgPLtgtLVP--PSMSNAVGITEALlAAeQ----gV-------k
d1reqa1 ( 236 ) nSIsISGyhMqeagAt--aDIEMAYTLADGVdYIraGesvglnVdqFApr
d1reqb1 ( 232 ) rAVTIdAniYHnaGAg--dvAELAWALATGAeYVraLveqgftateAFdt
d1uc4a- ( 256 ) MRFtsGSGsEvqmgyAe-gkSMLyLEARCIYITkAA------------gV
d1xrsa- ( 292 ) iINTgEdnylttadAfeeahTvLASQFINEQFAllA----gLp----eeq
b bb aa aaaaaaaaaaaaaaaaa
410 420 430 440 450
d1ccwb- ( 247 ) nITVGYgECG-nmiQDI-AALrCLeeQTneyLkaygyn---dVfVTTVFH
d1reqa1 ( 284 ) LsFfWGIgm--nffMEV-AKLRAARmlWAkLVhqfgPknpkSmSLrThSq
d1reqb1 ( 280 ) INFRVTAth--dqflTI-ARLRALReaWArIGevfgV-dedkrGArQNAi
d1uc4a- ( 293 ) qGLQNGSvscIgvPSAVpsGirAVLAENLICSSL---------dLECASS
d1xrsa- ( 334 ) MGLGhAfEMdpelkngf-lyELSqAQMAREIFp----------kApLKYm
bbbbbb aa aaaaaaaaaaaaaaa bbbb
460 470 480 490 500
d1ccwb- ( 292 ) QWmggFPq-deskAfgvIvtATtIAalAgATKVIVkTPheaigip-tkea
d1reqa1 ( 331 ) TSGwsLTaqdvynNvvrTciEAmAATqGHTqsLhTNSLDeaialp-tdfS
d1reqb1 ( 326 ) TSwreLtredPyvNilrgsiATfSASvGgAeSITTLPFTqalglpeddfP
d1uc4a- ( 334 ) NDqtFT--hsdmrrtaRllmQFlPgTD--FISSGYSAVPnyDNmfagSNE
d1xrsa- ( 373 ) PPtkfMtg-nifkGhiQDalFNmVTimTnQrIHlLGmLTealhtp-fmsD
aaaaaaaaaaaaaaa bbb aa
510 520 530 540 550
d1ccwb- ( 340 ) NAaGIkaTkmalnmle----gqrmpmskeLetemavIkaETkcILdkMfe
d1reqa1 ( 380 ) ariARntQlfLqqesgttrviDpwsgsayVEeLTwdLarkAwghI-qvev
d1reqb1 ( 376 ) lriaRntgivlAeevniGrvnDpaGgsyyVEsLTrsLadaAwefq-eVek
d1uc4a- ( 380 ) daedFddYNviQrdlkvdGgLrPvr-----eedviaiRnkAArALqaVFa
d1xrsa- ( 421 ) RalSIenAqyifnnmesiseeiqFkedgliqkrAgfVLekAnelLeeIeq
aaaaaaaaaaaaa aaaaaaaaaaaaaaaaa aaaa
560 570 580 590 600
d1ccwb- ( 386 ) lGk-gdLAiGTvkAFetGVMDIPfG-----pSk-----yNagkMmPVRDn
d1reqa1 ( 431 ) --------gGMakaiekgiPkmrIe-----eaaartqarids-gqpliGv
d1reqb1 ( 426 ) --------lgGMsAVmtehvtkvld-----acnaerakrlanrkqpitav
d1uc4a- ( 425 ) gMgLppItdeeveaATyAhGSkdMperniveDikfAqeIin---------
d1xrsa- ( 471 ) --------lgLfdTLekGiFg--g---------------vkrpkdggkgl
aaaaaa
610 620 630 640 650
d1ccwb- ( 425 ) l-GCVRYleFgnVpFteeiknyNre-rLqeRakfEgr-------------
d1reqa1 ( 468 ) nkyrleepplvlkvdnstVlaeQka-kLvkLrardpkVkaaldkItwAAg
d1reqb1 ( 464 ) sefpmi---gArsie
d1uc4a- ( 466 ) -----------knrngleVvkALaqggftdVAqdMlnIqkakltgdYlht
d1xrsa- ( 496 ) n-gVvskdenyyNPFvelMlnk
aaa
660 670 680 690
d1ccwb- ( 460 ) ----dvsf---qMVidDifAvgkgrligrpe
d1reqa1 ( 521 ) npdddpdr---nLLkLCIdAGramATVgeMSdaLevFgryt
d1reqb1
d1uc4a- ( 505 ) SAIivgdgqvlSavndvNdyaGpatGyrLqgerweeIknipgaldpn
d1xrsa-
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Glutamate mutase, large subunit | Glutamate mutase, large subunit | Clostridium cochlearium [TaxId: 1494] | 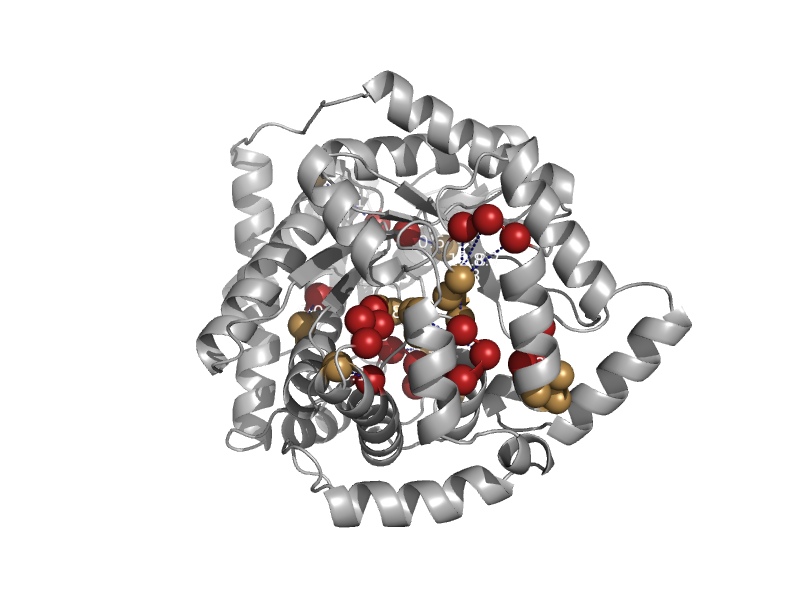 | view | |
| Methylmalonyl-CoA mutase alpha subunit, domain 1 | Methylmalonyl-CoA mutase, N-terminal (CoA-binding) domain | Propionibacterium freudenreichii, subsp. shermanii [TaxId: 1744] | 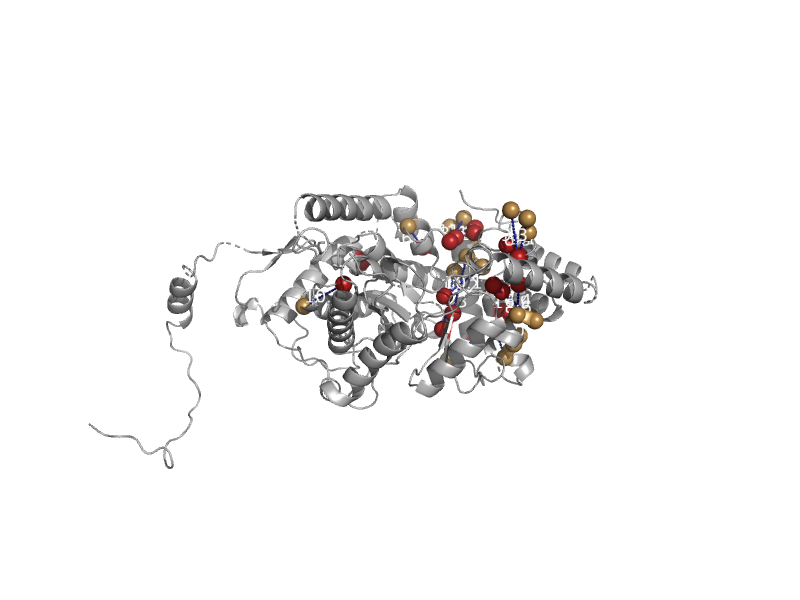 | view | |
| Methylmalonyl-CoA mutase beta subunit, domain 1 | Methylmalonyl-CoA mutase, N-terminal (CoA-binding) domain | Propionibacterium freudenreichii, subsp. shermanii [TaxId: 1744] | 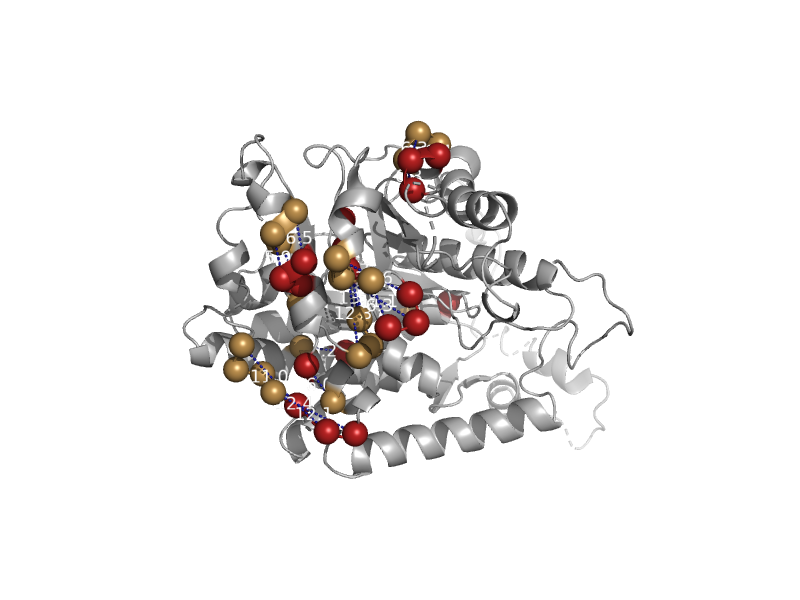 | view | |
| Diol dehydratase, alpha subunit | Diol dehydratase, alpha subunit | Klebsiella oxytoca [TaxId: 571] | 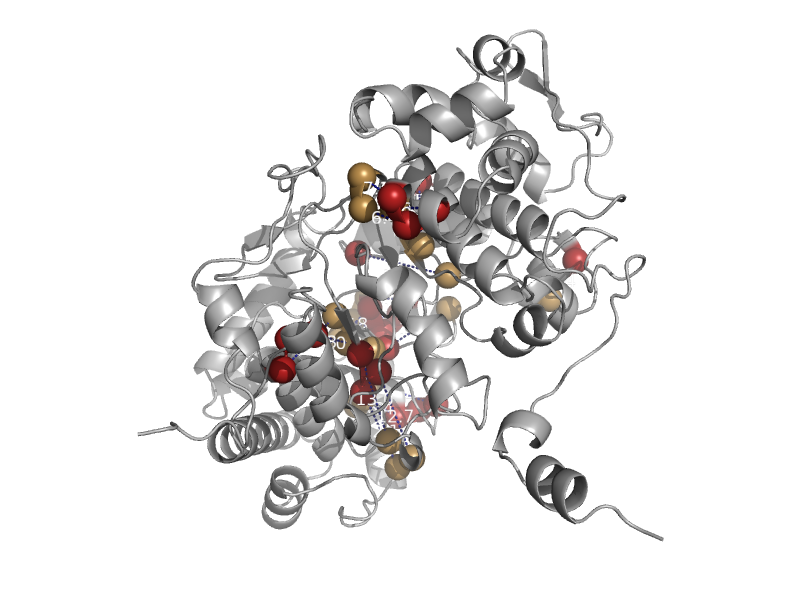 | view | |
| D-lysine 5,6-aminomutase alpha subunit, KamD | D-lysine 5,6-aminomutase alpha subunit, KamD | Clostridium sticklandii [TaxId: 1511] | 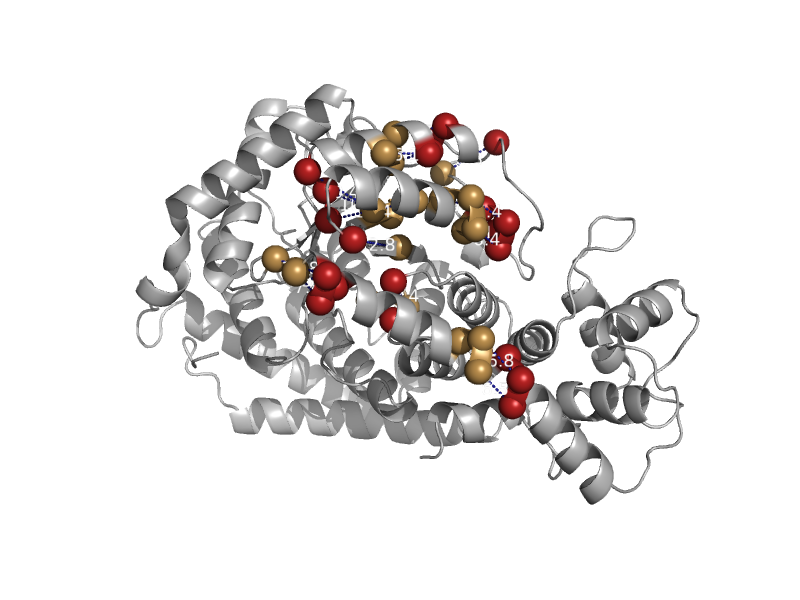 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | 230 | 240 | 250 | 260 | 270 | 280 | 290 | 300 | 310 | 320 | 330 | 340 | 350 | 360 | 370 | 380 | 390 | 400 | 410 | 420 | 430 | 440 | 450 | 460 | 470 | 480 | 490 | 500 | 510 | 520 | 530 | 540 | 550 | 560 | 570 | 580 | 590 | 600 | 610 | 620 | 630 | 640 | 650 | 660 | 670 | 680 | 690 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | E | L | K | N | K | K | W | T | - | - | - | D | E | E | F | H | K | Q | R | E | E | V | L | Q | Q | W | - | - | P | T | G | K | E | V | D | - | - | - | L | Q | E | A | V | D | Y | L | K | - | - | - | - | - | - | K | I | P | - | - | - | - | - | - | - | - | A | E | K | N | F | A | E | K | L | V | L | A | K | K | K | G | I | T | M | A | Q | P | - | - | R | A | G | V | A | L | L | D | - | - | - | - | - | E | H | I | E | L | L | R | Y | L | Q | D | - | E | G | G | A | D | F | L | P | S | T | I | D | A | Y | T | R | Q | N | R | Y | D | E | C | E | N | G | I | K | E | S | E | K | A | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | S | L | L | N | G | F | P | G | V | N | F | G | V | K | G | C | R | K | V | L | E | - | - | A | V | N | L | P | L | Q | A | R | H | G | T | P | D | S | R | L | L | A | E | I | I | H | A | G | G | W | T | S | N | E | G | G | G | I | S | Y | N | V | P | Y | - | A | K | N | V | T | I | E | K | S | L | L | D | W | Q | Y | C | D | R | L | V | G | F | Y | E | E | Q | G | V | H | I | N | R | E | P | F | G | P | L | T | G | T | L | V | P | - | - | P | S | M | S | N | A | V | G | I | T | E | A | L | L | A | A | E | Q | - | - | - | - | G | V | - | - | - | - | - | - | - | K | N | I | T | V | G | Y | G | E | C | G | - | N | M | I | Q | D | I | - | A | A | L | R | C | L | E | E | Q | T | N | E | Y | L | K | A | Y | G | Y | N | - | - | - | D | V | F | V | T | T | V | F | H | Q | W | M | G | G | F | P | Q | - | D | E | S | K | A | F | G | V | I | V | T | A | T | T | I | A | A | L | A | G | A | T | K | V | I | V | K | T | P | H | E | A | I | G | I | P | - | T | K | E | A | N | A | A | G | I | K | A | T | K | M | A | L | N | M | L | E | - | - | - | - | G | Q | R | M | P | M | S | K | E | L | E | T | E | M | A | V | I | K | A | E | T | K | C | I | L | D | K | M | F | E | L | G | K | - | G | D | L | A | I | G | T | V | K | A | F | E | T | G | V | M | D | I | P | F | G | - | - | - | - | - | P | S | K | - | - | - | - | - | Y | N | A | G | K | M | M | P | V | R | D | N | L | - | G | C | V | R | Y | L | E | F | G | N | V | P | F | T | E | E | I | K | N | Y | N | R | E | - | R | L | Q | E | R | A | K | F | E | G | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | V | S | F | - | - | - | Q | M | V | I | D | D | I | F | A | V | G | K | G | R | L | I | G | R | P | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| S | T | L | P | R | F | D | S | V | D | L | G | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | P | V | P | - | - | - | - | - | - | - | - | A | D | A | A | R | R | F | E | E | L | A | A | A | G | T | G | A | W | E | T | A | E | Q | I | P | V | G | T | L | F | N | E | D | V | Y | D | M | D | - | - | - | - | - | - | W | L | D | T | Y | A | G | I | - | - | - | - | P | P | F | V | H | G | P | Y | A | T | M | Y | A | F | R | P | W | T | I | R | Q | Y | A | - | - | G | F | S | T | A | K | - | - | - | - | - | E | S | N | A | F | Y | R | R | N | L | A | - | - | A | G | Q | K | G | L | S | V | A | F | D | L | P | T | H | R | G | Y | D | S | D | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | R | V | A | G | D | V | G | M | A | G | V | A | - | - | I | D | S | I | Y | D | M | R | E | L | F | A | G | I | P | L | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | M | S | V | S | M | T | M | N | G | A | V | L | P | I | L | A | L | Y | V | V | T | A | E | E | Q | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | V | P | - | E | Q | L | A | G | T | I | Q | N | D | I | L | K | E | F | M | V | R | N | T | Y | I | Y | P | P | Q | P | S | M | R | I | I | S | E | I | F | A | Y | T | S | A | N | M | P | - | K | W | N | S | I | S | I | S | G | Y | H | M | Q | E | A | G | A | T | - | - | A | D | I | E | M | A | Y | T | L | A | D | G | V | D | Y | I | R | A | G | E | S | V | G | L | N | V | D | Q | F | A | P | R | L | S | F | F | W | G | I | G | M | - | - | N | F | F | M | E | V | - | A | K | L | R | A | A | R | M | L | W | A | K | L | V | H | Q | F | G | P | K | N | P | K | S | M | S | L | R | T | H | S | Q | T | S | G | W | S | L | T | A | Q | D | V | Y | N | N | V | V | R | T | C | I | E | A | M | A | A | T | Q | G | H | T | Q | S | L | H | T | N | S | L | D | E | A | I | A | L | P | - | T | D | F | S | A | R | I | A | R | N | T | Q | L | F | L | Q | Q | E | S | G | T | T | R | V | I | D | P | W | S | G | S | A | Y | V | E | E | L | T | W | D | L | A | R | K | A | W | G | H | I | - | Q | V | E | V | - | - | - | - | - | - | - | - | G | G | M | A | K | A | I | E | K | G | I | P | K | M | R | I | E | - | - | - | - | - | E | A | A | A | R | T | Q | A | R | I | D | S | - | G | Q | P | L | I | G | V | N | K | Y | R | L | E | E | P | P | L | V | L | K | V | D | N | S | T | V | L | A | E | Q | K | A | - | K | L | V | K | L | R | A | R | D | P | K | V | K | A | A | L | D | K | I | T | W | A | A | G | N | P | D | D | D | P | D | R | - | - | - | N | L | L | K | L | C | I | D | A | G | R | A | M | A | T | V | G | E | M | S | D | A | L | E | V | F | G | R | Y | T | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | T | L | S | L | A | G | D | F | P | A | T | E | Q | W | E | E | V | E | K | V | L | N | R | G | R | P | - | - | - | - | - | - | - | - | P | E | Q | L | T | F | A | E | C | L | K | R | - | - | - | L | T | V | H | T | V | D | G | I | D | I | V | P | M | Y | R | P | D | A | P | K | - | - | - | - | - | - | - | - | - | - | L | G | Y | P | G | V | - | - | - | - | A | P | F | T | R | G | T | T | V | R | N | G | D | M | D | A | W | D | V | R | A | L | H | - | - | E | D | P | D | - | - | - | - | - | - | - | K | F | T | R | A | I | L | E | G | L | E | - | - | R | G | V | T | S | L | L | L | R | V | D | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | A | I | A | - | - | - | - | - | P | E | H | L | D | E | V | L | S | D | V | L | L | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | T | K | V | E | V | F | S | R | Y | D | Q | G | A | A | A | E | A | L | V | S | V | Y | E | R | S | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | P | A | K | D | L | A | L | N | L | G | L | D | P | I | G | F | A | A | L | Q | G | - | - | - | - | - | - | - | - | T | P | D | L | T | V | L | G | W | V | R | R | L | A | K | F | S | - | P | S | R | A | V | T | I | D | A | N | I | Y | H | N | A | G | A | G | - | - | D | V | A | E | L | A | W | A | L | A | T | G | A | E | Y | V | R | A | L | V | E | Q | G | F | T | A | T | E | A | F | D | T | I | N | F | R | V | T | A | T | H | - | - | D | Q | F | L | T | I | - | A | R | L | R | A | L | R | E | A | W | A | R | I | G | E | V | F | G | V | - | D | E | D | K | R | G | A | R | Q | N | A | I | T | S | W | R | E | L | T | R | E | D | P | Y | V | N | I | L | R | G | S | I | A | T | F | S | A | S | V | G | G | A | E | S | I | T | T | L | P | F | T | Q | A | L | G | L | P | E | D | D | F | P | L | R | I | A | R | N | T | G | I | V | L | A | E | E | V | N | I | G | R | V | N | D | P | A | G | G | S | Y | Y | V | E | S | L | T | R | S | L | A | D | A | A | W | E | F | Q | - | E | V | E | K | - | - | - | - | - | - | - | - | L | G | G | M | S | A | V | M | T | E | H | V | T | K | V | L | D | - | - | - | - | - | A | C | N | A | E | R | A | K | R | L | A | N | R | K | Q | P | I | T | A | V | S | E | F | P | M | I | - | - | - | G | A | R | S | I | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | R | S | K | R | F | E | A | L | A | K | R | P | V | N | Q | D | G | F | V | K | E | W | I | E | E | G | F | I | A | M | E | S | P | N | D | P | K | P | S | I | K | I | V | N | G | A | V | T | E | L | D | G | K | P | V | S | D | F | D | L | I | D | H | F | I | A | R | - | - | - | - | - | - | - | - | - | - | - | - | Y | G | I | N | L | N | R | A | E | E | V | M | A | M | D | S | V | K | L | A | N | M | L | C | D | P | N | V | K | R | S | - | - | - | - | - | - | E | I | V | P | L | T | T | A | M | T | P | A | K | I | V | E | V | V | S | H | M | N | V | V | E | M | M | M | A | M | Q | K | M | R | A | R | R | T | P | S | Q | Q | A | H | V | T | N | V | K | D | N | P | V | Q | I | A | A | D | A | A | E | G | A | W | R | G | F | D | E | Q | E | T | T | V | A | V | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | R | Y | A | P | F | N | A | I | A | L | L | V | G | S | Q | V | G | R | P | G | V | L | T | Q | C | S | L | E | E | A | T | E | L | K | L | G | M | L | G | H | T | C | Y | A | E | T | I | S | V | Y | G | T | E | P | V | F | T | D | G | - | - | D | D | T | P | W | S | K | G | F | L | A | S | S | Y | A | S | - | - | - | - | - | - | R | G | L | K | M | R | F | T | S | G | S | G | S | E | V | Q | M | G | Y | A | E | - | G | K | S | M | L | Y | L | E | A | R | C | I | Y | I | T | K | A | A | - | - | - | - | - | - | - | - | - | - | - | - | G | V | Q | G | L | Q | N | G | S | V | S | C | I | G | V | P | S | A | V | P | S | G | I | R | A | V | L | A | E | N | L | I | C | S | S | L | - | - | - | - | - | - | - | - | - | D | L | E | C | A | S | S | N | D | Q | T | F | T | - | - | H | S | D | M | R | R | T | A | R | L | L | M | Q | F | L | P | G | T | D | - | - | F | I | S | S | G | Y | S | A | V | P | N | Y | D | N | M | F | A | G | S | N | E | D | A | E | D | F | D | D | Y | N | V | I | Q | R | D | L | K | V | D | G | G | L | R | P | V | R | - | - | - | - | - | E | E | D | V | I | A | I | R | N | K | A | A | R | A | L | Q | A | V | F | A | G | M | G | L | P | P | I | T | D | E | E | V | E | A | A | T | Y | A | H | G | S | K | D | M | P | E | R | N | I | V | E | D | I | K | F | A | Q | E | I | I | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | N | R | N | G | L | E | V | V | K | A | L | A | Q | G | G | F | T | D | V | A | Q | D | M | L | N | I | Q | K | A | K | L | T | G | D | Y | L | H | T | S | A | I | I | V | G | D | G | Q | V | L | S | A | V | N | D | V | N | D | Y | A | G | P | A | T | G | Y | R | L | Q | G | E | R | W | E | E | I | K | N | I | P | G | A | L | D | P | N |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | E | S | K | L | N | L | D | F | N | L | V | E | K | A | R | A | K | A | K | A | I | A | I | D | T | Q | - | - | E | F | I | E | K | H | T | T | V | T | V | E | R | A | V | C | R | L | L | - | - | - | - | - | - | G | I | D | G | V | D | T | D | - | - | - | E | V | P | L | P | N | I | V | V | D | H | I | K | E | N | N | G | L | N | L | G | A | - | - | A | M | Y | I | A | N | A | V | L | N | T | G | K | T | P | Q | E | I | A | Q | A | I | S | A | G | E | L | D | L | T | K | L | P | M | K | D | L | F | E | V | K | T | K | A | L | S | M | A | K | E | T | V | E | K | I | K | N | N | R | S | I | R | E | S | R | F | E | E | Y | G | D | K | S | G | P | L | L | Y | V | I | V | A | - | - | T | G | N | I | Y | E | D | I | T | Q | A | V | A | A | A | K | Q | G | A | D | V | I | A | V | I | R | T | T | G | Q | S | L | L | D | Y | V | P | Y | G | A | T | T | E | G | F | G | G | T | Y | A | - | T | Q | E | N | F | R | L | M | R | E | A | L | D | K | V | G | A | E | V | G | K | Y | I | R | L | C | N | Y | C | S | G | L | C | M | P | E | I | A | A | M | G | A | I | E | R | L | D | V | M | L | N | D | A | L | Y | G | I | L | F | R | - | - | - | D | I | N | M | Q | R | T | M | I | D | Q | N | F | S | R | I | I | N | G | F | A | - | - | - | G | V | I | I | N | T | G | E | D | N | Y | L | T | T | A | D | A | F | E | E | A | H | T | V | L | A | S | Q | F | I | N | E | Q | F | A | L | L | A | - | - | - | - | G | L | P | - | - | - | - | E | E | Q | M | G | L | G | H | A | F | E | M | D | P | E | L | K | N | G | F | - | L | Y | E | L | S | Q | A | Q | M | A | R | E | I | F | P | - | - | - | - | - | - | - | - | - | - | K | A | P | L | K | Y | M | P | P | T | K | F | M | T | G | - | N | I | F | K | G | H | I | Q | D | A | L | F | N | M | V | T | I | M | T | N | Q | R | I | H | L | L | G | M | L | T | E | A | L | H | T | P | - | F | M | S | D | R | A | L | S | I | E | N | A | Q | Y | I | F | N | N | M | E | S | I | S | E | E | I | Q | F | K | E | D | G | L | I | Q | K | R | A | G | F | V | L | E | K | A | N | E | L | L | E | E | I | E | Q | - | - | - | - | - | - | - | - | L | G | L | F | D | T | L | E | K | G | I | F | G | - | - | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | V | K | R | P | K | D | G | G | K | G | L | N | - | G | V | V | S | K | D | E | N | Y | Y | N | P | F | V | E | L | M | L | N | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
