solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1euwa1 ( 2 ) mkkidvkildp---rvgkefplptyatsgs---aGldlrAClndavel
d1f7da- ( 1 ) miieg----------dGIldkrs-e-d---aGydLLAak--eihL
d1pkha- ( 1 ) miLsdkdIidyvtskrIiIkpfnkdfVgpcsYdVtLGdeFiiyd
d1sixa1 ( 1 ) m-sttlaivrldp---g----LplPsrahdgd---aGvdLySAe--dveL
d2bsya1 ( 4 ) cphiryafqnd-------kLllqqasv------grlTLvNkt--tilL
d2bsya2 ( 121 ) gpinhpqy
d2qxxa- ( 1 ) mlLsdrdLraEissgrLgIdpfddtlVqpsSIdVrLdclFrvfn
d3lqwa- ( 2 ) devllvkklv--------dAivPtrg-sca---aGIdLySnt--nFiI
d3t64a- ( 1 ) MhLkIVClsdeVremYknHkthh---egd---sgLdLfIvk--devL
d3tq5a- ( 25 ) kqpisltratpgs---aGldLcSts--htvL
d4b0ha- ( 2 ) tmqikikyldetQt-----rIski---eqg---dwidLrAAe--dvtI
d4gv8a- ( 3 ) ntlqVrlls--------nArmPern-htd---aGydIfSAe--tvvL
d4xjca- ( 1 ) miLsGktIsekltekeLeItplteeQiqpasVDLrLGphFVtid
d5y5qa- ( 3 ) ssasvvfmrfap---pgeetAlpPrratpgs---vAydLFPse--emdI
d7n6sa1 ( 1 ) mtiievkikklnf---g------lpeyatehs---aGmdLvAanq-sitI
bbbb bbb
60 70 80 90 100
d1euwa1 ( 44 ) apg--------------dttlvptGlAihiadp-----------------
d1f7da- ( 29 ) lpg--------------evkvIpTgVkLmL-pk-----------------
d1pkha- ( 45 ) d---evydlskeln--ykrikIk--nSILVCplnynLteekinyfkekyn
d1sixa1 ( 38 ) apg--------------rralVrTGVaVaV-pf-----------------
d2bsya1 ( 37 ) rpm--------------ktttvdLgLyArP-pe-----------------
d2bsya2 ( 129 ) p------------gdvGldVsLpk--dlaLfph-----------------
d2qxxa- ( 45 ) ntrythidpakqqdelTslvqpvdgepfvLhpg-----------------
d3lqwa- ( 38 ) qph--------------erflVsTGVsVqI-ph-----------------
d3t64a- ( 40 ) kpk--------------sttfVkLgIkAiAlqyksnyyykn---------
d3tq5a- ( 52 ) tpem-------------gpqaLsTgiyGpL-pp-----------------
d4b0ha- ( 37 ) kkd--------------efklVpLGVAMeLpe------------------
d4gv8a- ( 38 ) ePq--------------ekavIkTdVAVsI-p------------------
d4xjca- ( 45 ) dskeavisferpIr--yrewttsd-etivLpph-----------------
d5y5qa- ( 44 ) epm--------------glakIsTgyGIdkfpd-----------------
d7n6sa1 ( 42 ) kvg--------------siqlIpTGIaIaL-pe-----------------
bbbb bbb
110 120 130 140 150
d1euwa1 ( 63 ) ---------------------slAAmMlprsglghkhGivlGnlvGlids
d1f7da- ( 47 ) ---------------------gywGlIigkssigsk-gLdVl--ggvide
d1pkha- ( 88 ) VdyVveggVlGtTnEyIeLpndIsAqYqgrsslgr-vfLtShqtagwidA
d1sixa1 ( 56 ) ---------------------gmvGlVhprsglatrvgLsIvnspgtida
d2bsya1 ( 55 ) ---------------------gHGLmLwGsts----rpVtsh--vgiidp
d2bsya2 ( 148 ) ------qtvsvtltvpppsiphhrpTIfgrsgLam-qgIlVk--pcrWrr
d2qxxa- ( 78 ) ------efVlGSTlElFtLpdnLaGrLegksslgr-lgLlThstagfidp
d3lqwa- ( 56 ) ---------------------qCyGrIAprsslalkygIdVg--agvide
d3t64a- ( 81 ) -------------------ivntsFlLfprssIsk-tpLrLansiglida
d3tq5a- ( 71 ) ---------------------ntfGlIlgrssitm--gLqVy--pgvidn
d4b0ha- ( 55 ) ---------------------gyeAhVvprssTyknfGViQtnsmgvide
d4gv8a- ( 56 ) ---------------------gyvGlLtsrsgVsst-hLvIe--tgkida
d4xjca- ( 75 ) ------tfLlATTmEtVkLpnhLtAfVegrssvgr-lgLfIqn-agwvdp
d5y5qa- ( 63 ) ---------------------gCyGqIvsrsgMtwknnTsVp--tgtinv
d7n6sa1 ( 60 ) ---------------------sfeAqIrprsglavkhgItVanspgtida
bbbbbb aaaa bbb bb
160 170 180 190 200
d1euwa1 ( 92 ) dyq---gqlmisvwnrgq--------------------dsftiqpgeriA
d1f7da- ( 73 ) gyr---geIgViMiNvsr--------------------ksitLmerqkIA
d1pkha- ( 137 ) gf---kgkItLeIvAfd---------------------kpviLyknqrIG
d1sixa1 ( 85 ) gyr---geIkVaLiNldpa-------------------apIvVhrgdrIA
d2bsya1 ( 78 ) gyt---geLrLiLqNqrr--------------------ynstLrpseLkI
d2bsya2 ( 189 ) g------GVdVsLtNfsd--------------------qtVfLnkyrrFC
d2qxxa- ( 121 ) gf---sGhitLeLsNvan--------------------lpitLwpgmkIG
d3lqwa- ( 83 ) dyr---geIkViLfNhsn--------------------eifnGk-gdrIA
d3t64a- ( 111 ) gyr---GeIiAaLdNtsd--------------------qeyhIkkndklV
d3tq5a- ( 97 ) dyt---geIkImAkAvn---------------------nivtVsg--nIA
d4b0ha- ( 84 ) sykgdndfWfFpAyAlr----------------------dTeIkkgdrIC
d4gv8a- ( 83 ) gyh---gnLgInIkNdaiagipgVfdigeidsdairqygTYqInegdkLA
d4xjca- ( 117 ) gf---nGqitLeLfNanr--------------------lpieLpigrrIC
d5y5qa- ( 90 ) dyr---geLkViLrNhsae-------------------ksVpIrkgtSIA
d7n6sa1 ( 89 ) dyr---geIkVlLiNlgn---------------------dfiIe-gmrIA
bbbbbb bbb bbb
210 220 230 240 250
d1euwa1 ( 119 ) qMifvpvv---------qa-efnlvedf
d1f7da- ( 100 ) qLiilpck-----------------------h--------evleqgkv--
d1pkha- ( 163 ) qLiFskllspad--vgyserk-----------------------t
d1sixa1 ( 113 ) qLlvqrve---------lv-elvevssfdeag--------lastsrgd--
d2bsya1 ( 105 ) hLaafrya---------tpq------------------------------
d2bsya2 ( 213 ) qLvylh-----khhLtsfysphSdagvLgprsLfrwas----ctfeevps
d2qxxa- ( 148 ) qLcMlrltspsehpygs---sragskyqgqrgPtpsrsyqnfirs
d3lqwa- ( 110 ) qLiieris---------yc-riseve--------------lntt
d3t64a- ( 138 ) qLvSftge---------pLsfelveeLdetsrGeggfgs
d3tq5a- ( 123 ) qLillpli---------tdn------------------------------
d4b0ha- ( 112 ) qFrimkkm---------p---------------------avelveveh--
d4gv8a- ( 136 ) qLvivpiw---------tpl------qvefs
d4xjca- ( 144 ) qLvFaevtgev-apyq--------gkylfqkgAtmseiykdaf
d5y5qa- ( 118 ) qLiflryc---------dveeeqivyinettg--------ertiidss--
d7n6sa1 ( 116 ) qMiia-ye---------rv-lwaetsl
bbbbbb
d1euwa1
d1f7da- ( 117 ) -vm
d1pkha-
d1sixa1 ( 143 ) -gg
d2bsya1 ( 116 ) --m
d2bsya2 ( 254 ) lam
d2qxxa-
d3lqwa-
d3t64a-
d3tq5a- ( 136 ) --v
d4b0ha- ( 130 ) -lg
d4gv8a-
d4xjca-
d5y5qa- ( 149 ) -sk
d7n6sa1
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Deoxyuridine 5'-triphosphate nucleotidohydrolase (dUTPase) | dUTPase-like | Escherichia coli [TaxId: 562] | 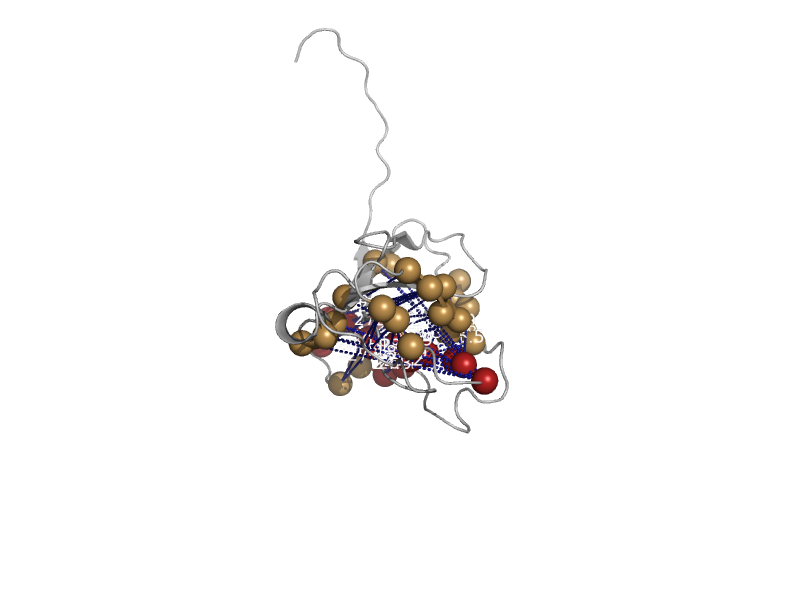 | view | |
| Deoxyuridine 5'-triphosphate nucleotidohydrolase (dUTPase) | dUTPase-like | Feline immunodeficiency virus [TaxId: 11673] | 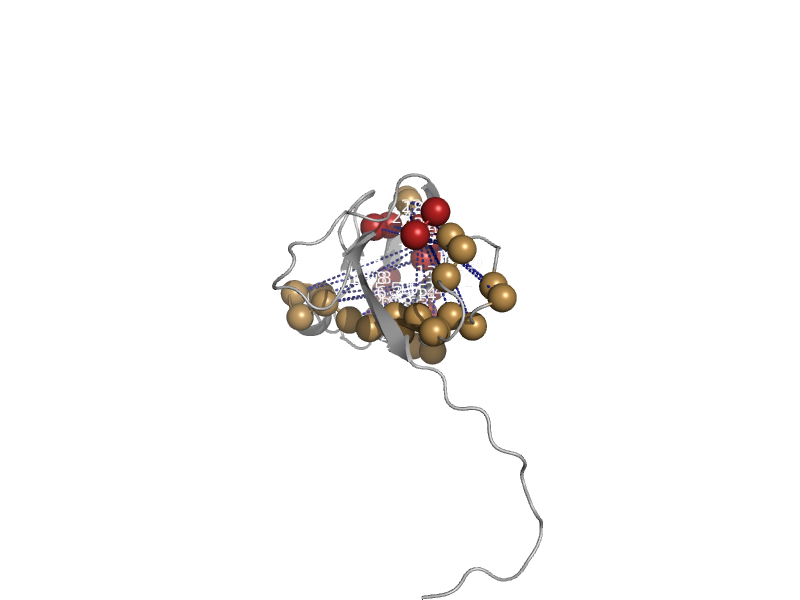 | view | |
| Bifunctional dCTP deaminase/dUTPase | dUTPase-like | Methanococcus jannaschii [TaxId: 2190] | 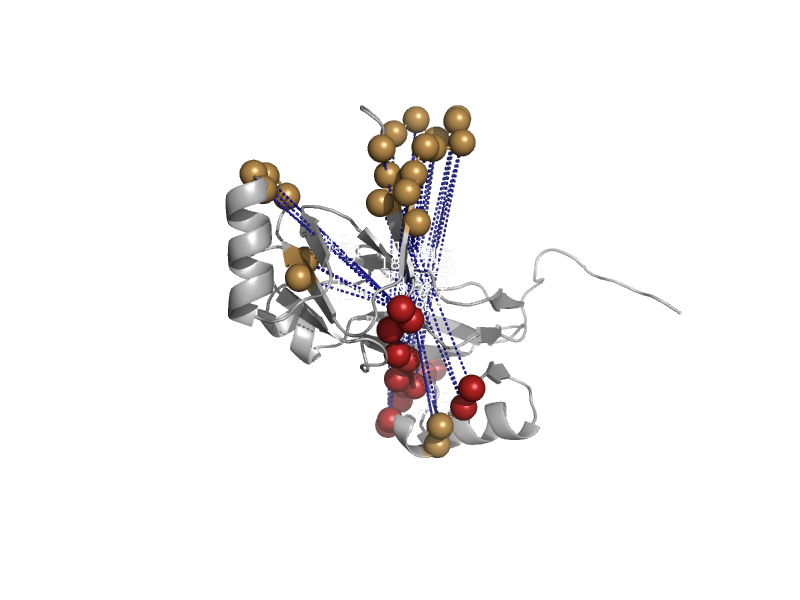 | view | |
| Deoxyuridine 5'-triphosphate nucleotidohydrolase (dUTPase) | dUTPase-like | Mycobacterium tuberculosis, rv2697c [TaxId: 1773] | 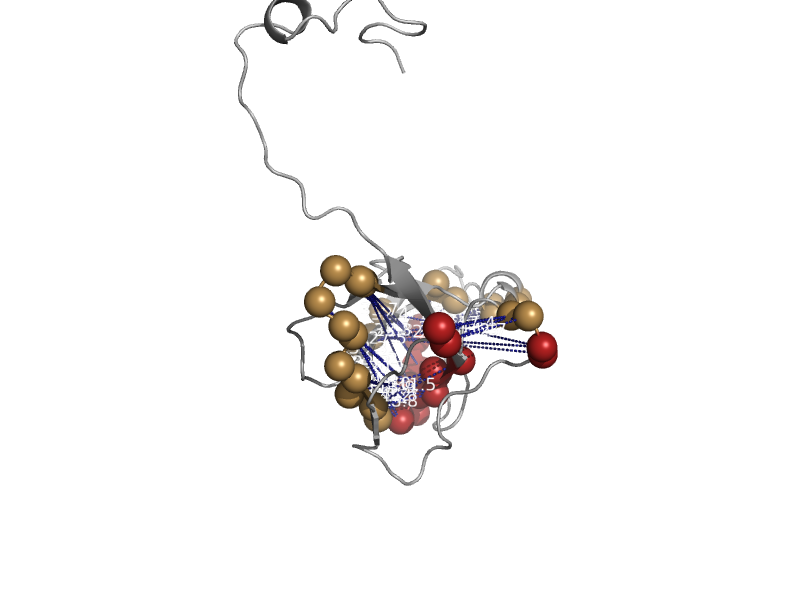 | view | |
| Monomeric viral dUTPase | dUTPase-like | Epstein-Barr virus [TaxId: 10376] | 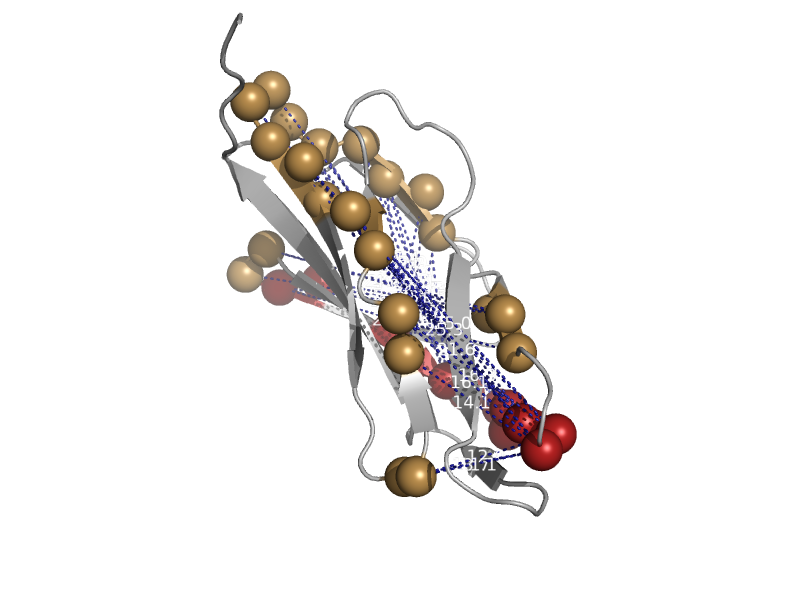 | view | |
| Monomeric viral dUTPase | dUTPase-like | Epstein-Barr virus [TaxId: 10376] | 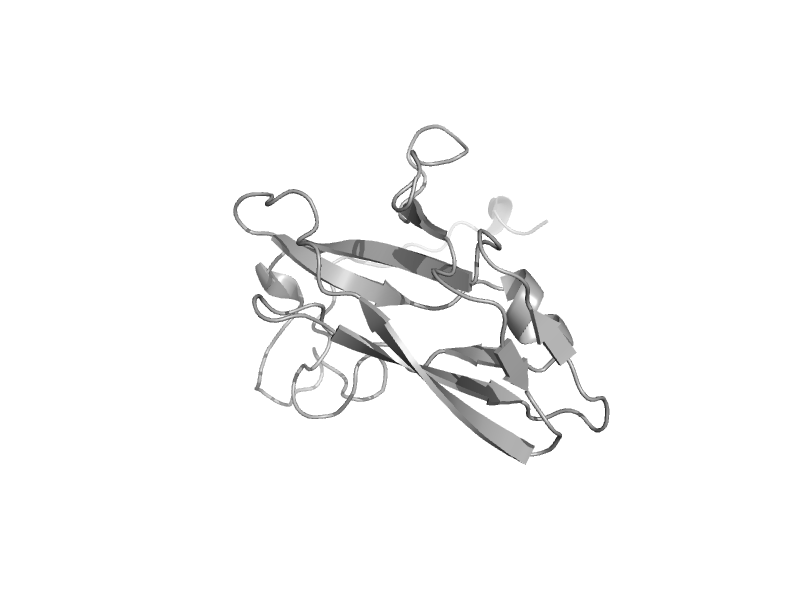 | view | |
| Deoxycytidine triphosphate deaminase (dCTP deaminase) | dUTPase-like | Mycobacterium tuberculosis [TaxId: 83332] | 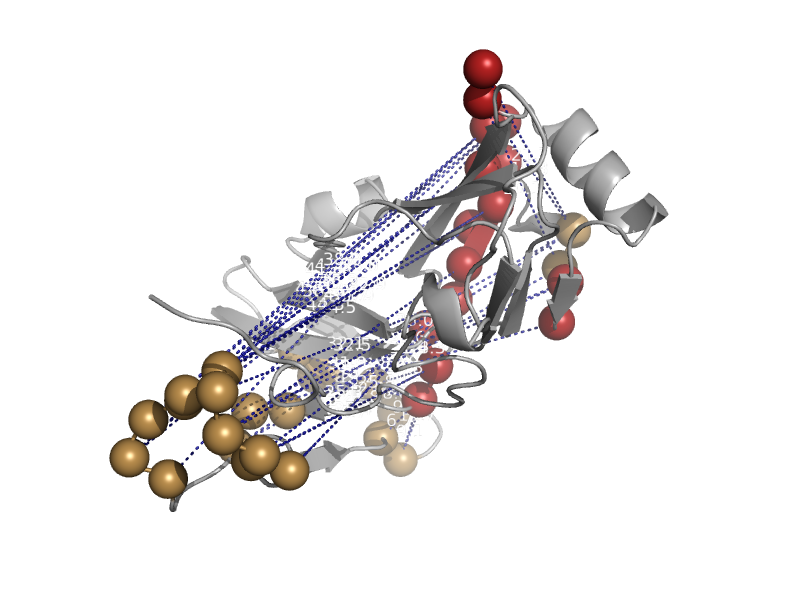 | view | |
| automated matches | automated matches | Entamoeba histolytica [TaxId: 294381] | 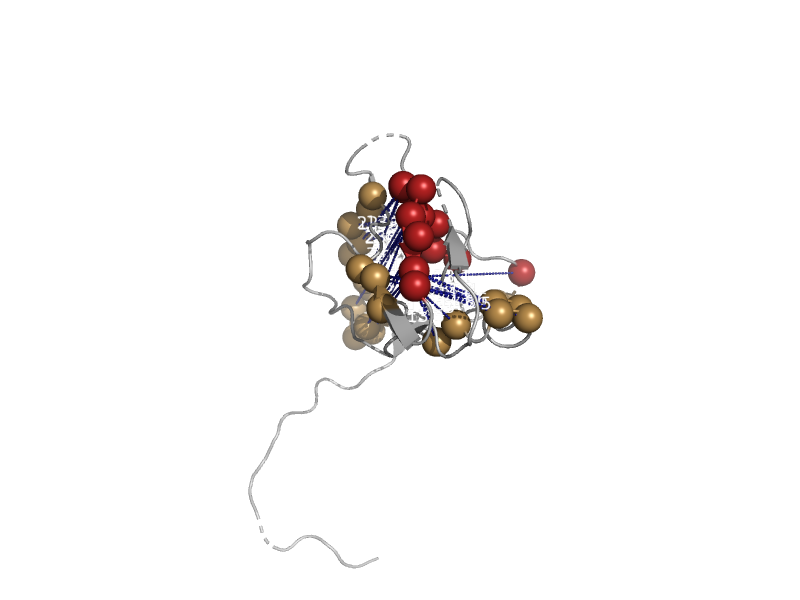 | view | |
| automated matches | dUTPase-like | Plasmodium falciparum [TaxId: 36329] |  | view | |
| automated matches | automated matches | Mason-pfizer monkey virus [TaxId: 11855] |  | view | |
| automated matches | automated matches | Bacillus subtilis [TaxId: 1423] |  | view | |
| automated matches | automated matches | Staphylococcus phage [TaxId: 12360] |  | view | |
| automated matches | automated matches | Bacillus halodurans [TaxId: 272558] |  | view | |
| automated matches | automated matches | White spot syndrome virus (isolate shrimp/china/tongan/1996) [TaxId: 654913] |  | view | |
| automated matches | automated matches | Rickettsia prowazekii [TaxId: 272947] |  | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | 230 | 240 | 250 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | M | K | K | I | D | V | K | I | L | D | P | - | - | - | R | V | G | K | E | F | P | L | P | T | Y | A | T | S | G | S | - | - | - | A | G | L | D | L | R | A | C | L | N | D | A | V | E | L | A | P | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | T | T | L | V | P | T | G | L | A | I | H | I | A | D | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | L | A | A | M | M | L | P | R | S | G | L | G | H | K | H | G | I | V | L | G | N | L | V | G | L | I | D | S | D | Y | Q | - | - | - | G | Q | L | M | I | S | V | W | N | R | G | Q | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | S | F | T | I | Q | P | G | E | R | I | A | Q | M | I | F | V | P | V | V | - | - | - | - | - | - | - | - | - | Q | A | - | E | F | N | L | V | E | D | F | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | M | I | I | E | G | - | - | - | - | - | - | - | - | - | - | D | G | I | L | D | K | R | S | - | E | - | D | - | - | - | A | G | Y | D | L | L | A | A | K | - | - | E | I | H | L | L | P | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | V | K | V | I | P | T | G | V | K | L | M | L | - | P | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | Y | W | G | L | I | I | G | K | S | S | I | G | S | K | - | G | L | D | V | L | - | - | G | G | V | I | D | E | G | Y | R | - | - | - | G | E | I | G | V | I | M | I | N | V | S | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | S | I | T | L | M | E | R | Q | K | I | A | Q | L | I | I | L | P | C | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | H | - | - | - | - | - | - | - | - | E | V | L | E | Q | G | K | V | - | - | - | V | M |
| - | - | - | - | - | - | M | I | L | S | D | K | D | I | I | D | Y | V | T | S | K | R | I | I | I | K | P | F | N | K | D | F | V | G | P | C | S | Y | D | V | T | L | G | D | E | F | I | I | Y | D | D | - | - | - | E | V | Y | D | L | S | K | E | L | N | - | - | Y | K | R | I | K | I | K | - | - | N | S | I | L | V | C | P | L | N | Y | N | L | T | E | E | K | I | N | Y | F | K | E | K | Y | N | V | D | Y | V | V | E | G | G | V | L | G | T | T | N | E | Y | I | E | L | P | N | D | I | S | A | Q | Y | Q | G | R | S | S | L | G | R | - | V | F | L | T | S | H | Q | T | A | G | W | I | D | A | G | F | - | - | - | K | G | K | I | T | L | E | I | V | A | F | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | P | V | I | L | Y | K | N | Q | R | I | G | Q | L | I | F | S | K | L | L | S | P | A | D | - | - | V | G | Y | S | E | R | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | T | - | - | - | - | - | - | - | - |
| M | - | S | T | T | L | A | I | V | R | L | D | P | - | - | - | G | - | - | - | - | L | P | L | P | S | R | A | H | D | G | D | - | - | - | A | G | V | D | L | Y | S | A | E | - | - | D | V | E | L | A | P | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | R | A | L | V | R | T | G | V | A | V | A | V | - | P | F | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | M | V | G | L | V | H | P | R | S | G | L | A | T | R | V | G | L | S | I | V | N | S | P | G | T | I | D | A | G | Y | R | - | - | - | G | E | I | K | V | A | L | I | N | L | D | P | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | P | I | V | V | H | R | G | D | R | I | A | Q | L | L | V | Q | R | V | E | - | - | - | - | - | - | - | - | - | L | V | - | E | L | V | E | V | S | S | F | D | E | A | G | - | - | - | - | - | - | - | - | L | A | S | T | S | R | G | D | - | - | - | G | G |
| - | - | C | P | H | I | R | Y | A | F | Q | N | D | - | - | - | - | - | - | - | K | L | L | L | Q | Q | A | S | V | - | - | - | - | - | - | G | R | L | T | L | V | N | K | T | - | - | T | I | L | L | R | P | M | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | T | T | T | V | D | L | G | L | Y | A | R | P | - | P | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | H | G | L | M | L | W | G | S | T | S | - | - | - | - | R | P | V | T | S | H | - | - | V | G | I | I | D | P | G | Y | T | - | - | - | G | E | L | R | L | I | L | Q | N | Q | R | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Y | N | S | T | L | R | P | S | E | L | K | I | H | L | A | A | F | R | Y | A | - | - | - | - | - | - | - | - | - | T | P | Q | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | P | I | N | H | P | Q | Y | P | - | - | - | - | - | - | - | - | - | - | - | - | G | D | V | G | L | D | V | S | L | P | K | - | - | D | L | A | L | F | P | H | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | T | V | S | V | T | L | T | V | P | P | P | S | I | P | H | H | R | P | T | I | F | G | R | S | G | L | A | M | - | Q | G | I | L | V | K | - | - | P | C | R | W | R | R | G | - | - | - | - | - | - | G | V | D | V | S | L | T | N | F | S | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | T | V | F | L | N | K | Y | R | R | F | C | Q | L | V | Y | L | H | - | - | - | - | - | K | H | H | L | T | S | F | Y | S | P | H | S | D | A | G | V | L | G | P | R | S | L | F | R | W | A | S | - | - | - | - | C | T | F | E | E | V | P | S | L | A | M |
| - | - | - | - | - | - | M | L | L | S | D | R | D | L | R | A | E | I | S | S | G | R | L | G | I | D | P | F | D | D | T | L | V | Q | P | S | S | I | D | V | R | L | D | C | L | F | R | V | F | N | N | T | R | Y | T | H | I | D | P | A | K | Q | Q | D | E | L | T | S | L | V | Q | P | V | D | G | E | P | F | V | L | H | P | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | F | V | L | G | S | T | L | E | L | F | T | L | P | D | N | L | A | G | R | L | E | G | K | S | S | L | G | R | - | L | G | L | L | T | H | S | T | A | G | F | I | D | P | G | F | - | - | - | S | G | H | I | T | L | E | L | S | N | V | A | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | P | I | T | L | W | P | G | M | K | I | G | Q | L | C | M | L | R | L | T | S | P | S | E | H | P | Y | G | S | - | - | - | S | R | A | G | S | K | Y | Q | G | Q | R | G | P | T | P | S | R | S | Y | Q | N | F | I | R | S | - | - | - | - | - | - | - | - |
| - | - | D | E | V | L | L | V | K | K | L | V | - | - | - | - | - | - | - | - | D | A | I | V | P | T | R | G | - | S | C | A | - | - | - | A | G | I | D | L | Y | S | N | T | - | - | N | F | I | I | Q | P | H | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | R | F | L | V | S | T | G | V | S | V | Q | I | - | P | H | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | C | Y | G | R | I | A | P | R | S | S | L | A | L | K | Y | G | I | D | V | G | - | - | A | G | V | I | D | E | D | Y | R | - | - | - | G | E | I | K | V | I | L | F | N | H | S | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | I | F | N | G | K | - | G | D | R | I | A | Q | L | I | I | E | R | I | S | - | - | - | - | - | - | - | - | - | Y | C | - | R | I | S | E | V | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | N | T | T | - | - | - | - | - | - | - | - | - |
| - | - | - | M | H | L | K | I | V | C | L | S | D | E | V | R | E | M | Y | K | N | H | K | T | H | H | - | - | - | E | G | D | - | - | - | S | G | L | D | L | F | I | V | K | - | - | D | E | V | L | K | P | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | T | T | F | V | K | L | G | I | K | A | I | A | L | Q | Y | K | S | N | Y | Y | Y | K | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | I | V | N | T | S | F | L | L | F | P | R | S | S | I | S | K | - | T | P | L | R | L | A | N | S | I | G | L | I | D | A | G | Y | R | - | - | - | G | E | I | I | A | A | L | D | N | T | S | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | E | Y | H | I | K | K | N | D | K | L | V | Q | L | V | S | F | T | G | E | - | - | - | - | - | - | - | - | - | P | L | S | F | E | L | V | E | E | L | D | E | T | S | R | G | E | G | G | F | G | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | Q | P | I | S | L | T | R | A | T | P | G | S | - | - | - | A | G | L | D | L | C | S | T | S | - | - | H | T | V | L | T | P | E | M | - | - | - | - | - | - | - | - | - | - | - | - | - | G | P | Q | A | L | S | T | G | I | Y | G | P | L | - | P | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | T | F | G | L | I | L | G | R | S | S | I | T | M | - | - | G | L | Q | V | Y | - | - | P | G | V | I | D | N | D | Y | T | - | - | - | G | E | I | K | I | M | A | K | A | V | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | I | V | T | V | S | G | - | - | N | I | A | Q | L | I | L | L | P | L | I | - | - | - | - | - | - | - | - | - | T | D | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | V |
| - | - | T | M | Q | I | K | I | K | Y | L | D | E | T | Q | T | - | - | - | - | - | R | I | S | K | I | - | - | - | E | Q | G | - | - | - | D | W | I | D | L | R | A | A | E | - | - | D | V | T | I | K | K | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | F | K | L | V | P | L | G | V | A | M | E | L | P | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | Y | E | A | H | V | V | P | R | S | S | T | Y | K | N | F | G | V | I | Q | T | N | S | M | G | V | I | D | E | S | Y | K | G | D | N | D | F | W | F | F | P | A | Y | A | L | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | T | E | I | K | K | G | D | R | I | C | Q | F | R | I | M | K | K | M | - | - | - | - | - | - | - | - | - | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | V | E | L | V | E | V | E | H | - | - | - | L | G |
| - | - | - | N | T | L | Q | V | R | L | L | S | - | - | - | - | - | - | - | - | N | A | R | M | P | E | R | N | - | H | T | D | - | - | - | A | G | Y | D | I | F | S | A | E | - | - | T | V | V | L | E | P | Q | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | K | A | V | I | K | T | D | V | A | V | S | I | - | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | Y | V | G | L | L | T | S | R | S | G | V | S | S | T | - | H | L | V | I | E | - | - | T | G | K | I | D | A | G | Y | H | - | - | - | G | N | L | G | I | N | I | K | N | D | A | I | A | G | I | P | G | V | F | D | I | G | E | I | D | S | D | A | I | R | Q | Y | G | T | Y | Q | I | N | E | G | D | K | L | A | Q | L | V | I | V | P | I | W | - | - | - | - | - | - | - | - | - | T | P | L | - | - | - | - | - | - | Q | V | E | F | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | M | I | L | S | G | K | T | I | S | E | K | L | T | E | K | E | L | E | I | T | P | L | T | E | E | Q | I | Q | P | A | S | V | D | L | R | L | G | P | H | F | V | T | I | D | D | S | K | E | A | V | I | S | F | E | R | P | I | R | - | - | Y | R | E | W | T | T | S | D | - | E | T | I | V | L | P | P | H | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | T | F | L | L | A | T | T | M | E | T | V | K | L | P | N | H | L | T | A | F | V | E | G | R | S | S | V | G | R | - | L | G | L | F | I | Q | N | - | A | G | W | V | D | P | G | F | - | - | - | N | G | Q | I | T | L | E | L | F | N | A | N | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | P | I | E | L | P | I | G | R | R | I | C | Q | L | V | F | A | E | V | T | G | E | V | - | A | P | Y | Q | - | - | - | - | - | - | - | - | G | K | Y | L | F | Q | K | G | A | T | M | S | E | I | Y | K | D | A | F | - | - | - | - | - | - | - | - | - | - |
| - | S | S | A | S | V | V | F | M | R | F | A | P | - | - | - | P | G | E | E | T | A | L | P | P | R | R | A | T | P | G | S | - | - | - | V | A | Y | D | L | F | P | S | E | - | - | E | M | D | I | E | P | M | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | L | A | K | I | S | T | G | Y | G | I | D | K | F | P | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | C | Y | G | Q | I | V | S | R | S | G | M | T | W | K | N | N | T | S | V | P | - | - | T | G | T | I | N | V | D | Y | R | - | - | - | G | E | L | K | V | I | L | R | N | H | S | A | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | S | V | P | I | R | K | G | T | S | I | A | Q | L | I | F | L | R | Y | C | - | - | - | - | - | - | - | - | - | D | V | E | E | E | Q | I | V | Y | I | N | E | T | T | G | - | - | - | - | - | - | - | - | E | R | T | I | I | D | S | S | - | - | - | S | K |
| M | T | I | I | E | V | K | I | K | K | L | N | F | - | - | - | G | - | - | - | - | - | - | L | P | E | Y | A | T | E | H | S | - | - | - | A | G | M | D | L | V | A | A | N | Q | - | S | I | T | I | K | V | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | I | Q | L | I | P | T | G | I | A | I | A | L | - | P | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | F | E | A | Q | I | R | P | R | S | G | L | A | V | K | H | G | I | T | V | A | N | S | P | G | T | I | D | A | D | Y | R | - | - | - | G | E | I | K | V | L | L | I | N | L | G | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | F | I | I | E | - | G | M | R | I | A | Q | M | I | I | A | - | Y | E | - | - | - | - | - | - | - | - | - | R | V | - | L | W | A | E | T | S | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
