solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1e0ta1 ( 70 ) peIrTmkL
d1o65a1 ( 4 ) flvereqmrypVdVytgakiqvdg--elmLtelgLegdeqa---------
d1orua- ( 2 ) wkrtakAeGLYiAdtksfvTkqdkldFdygGIpgDlhfgltkkAgar
d2g50a1 ( 116 ) peIrTGlI
d3hqna2 ( 88 ) eIrTgqF
60 70 80 90 100
d1e0ta1 ( 78 ) egg--ndvsL-agqtFtFTtdksv--------------iGns-emVAVty
d1o65a1 ( 60 ) --------hggpdrALcHYPrehylyWarefpeqaelFvApaFgENLSTd
d1orua- ( 51 ) epfsrgteifNr-rQIsIVSieECneIAlk---gVprIlpeWLgANVAvs
d2g50a1 ( 124 ) kgsgtaeVeLkkgatLkITldnayme------------kCde-niLWLdY
d3hqna2 ( 95 ) vg---gdavMergatCyVTTdpafad------------kGtk-dkFyIdY
bb bbbb b
110 120 130 140 150
d1e0ta1 ( 111 ) egFttdLsvgntVlVddglIgMeVtai-eg--------------------
d1o65a1 ( 102 ) gLtesnVyMGDiFrWge--AlIqVSqPrsPcyklNyhF-----disdIAq
d1orua- ( 99 ) gpdlTslkegSrIiFpsg-AaLlCegeNdPciqpGevIqsyypdqpklas
d2g50a1 ( 161 ) knIckvVdvgskVyVddglISLqVkqk-gp--------------------
d3hqna2 ( 129 ) qnLskvVrpgnyIyIddgiLiLqVqshede--------------------
bbbb bbbbbbbb
160 170 180 190 200
d1e0ta1 ( 140 ) ---------nkViCkVlnngdLgenkgVnLpgvsial
d1o65a1 ( 145 ) lMQnt--gkVGWlYsViapgkVsadapLeLvsrvsdVtVqeAAaIAWhmp
d1orua- ( 149 ) aFvrhAlgiRGIVCiVerpgaVytgdeIevhsyq
d2g50a1 ( 190 ) ---------dfLvTeVenggfLgskkgVnLpgaavdl
d3hqna2 ( 159 ) ---------qTLeCtVtnshtIsdrrgVnLpgCdvdl
bbbbb bbb bb
210 220 230 240
d1e0ta1
d1o65a1 ( 193 ) fdddqYhrLlsAagLSkswtrtMqkRrlsgkiedfsrrLwg
d1orua-
d2g50a1
d3hqna2
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Pyruvate kinase (PK) | Pyruvate kinase beta-barrel domain | Escherichia coli [TaxId: 562] | 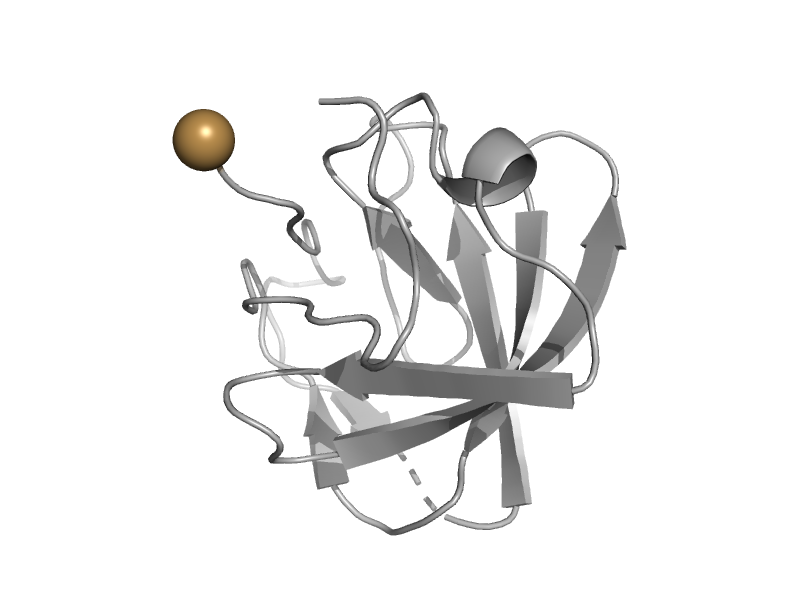 | view | |
| Hypothetical protein YiiM | MOSC (MOCO sulphurase C-terminal) domain | Escherichia coli [TaxId: 562] | 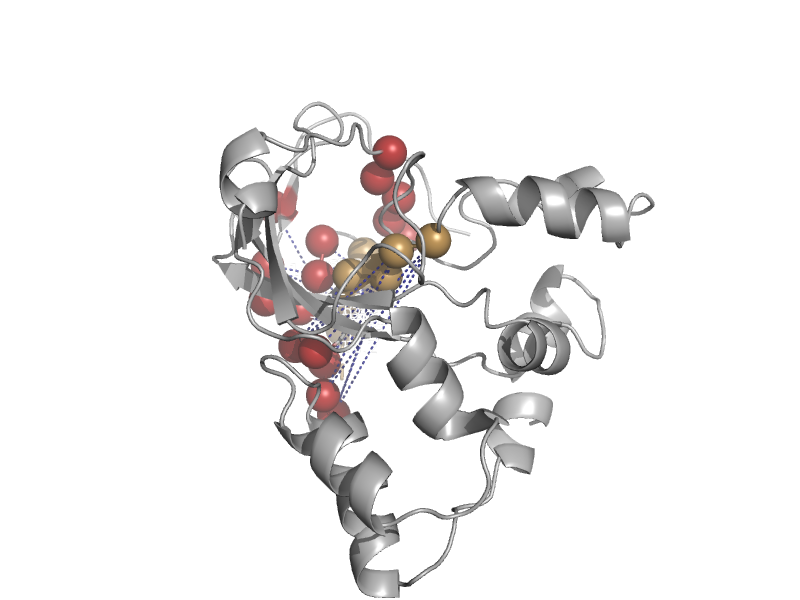 | view | |
| Hypothetical protein YuaD | MOSC (MOCO sulphurase C-terminal) domain | Bacillus subtilis [TaxId: 1423] | 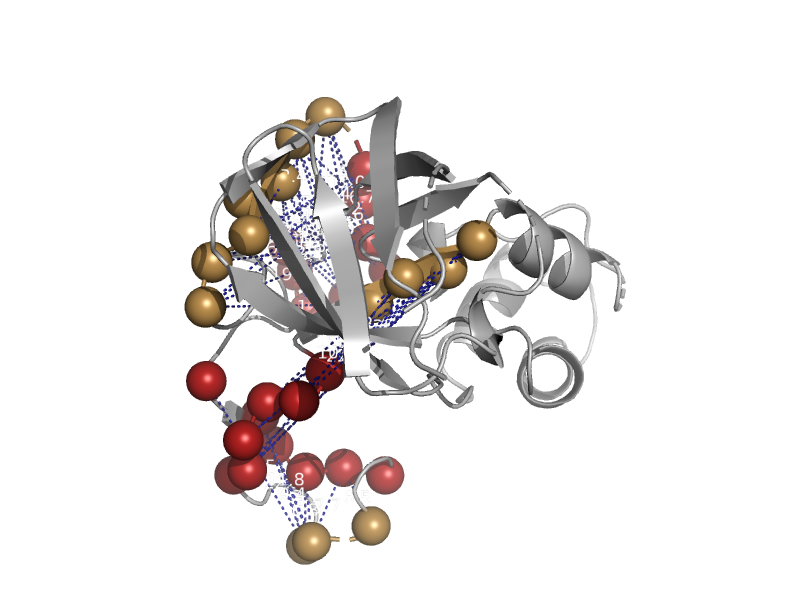 | view | |
| Pyruvate kinase (PK) | Pyruvate kinase beta-barrel domain | Rabbit (Oryctolagus cuniculus) [TaxId: 9986] | 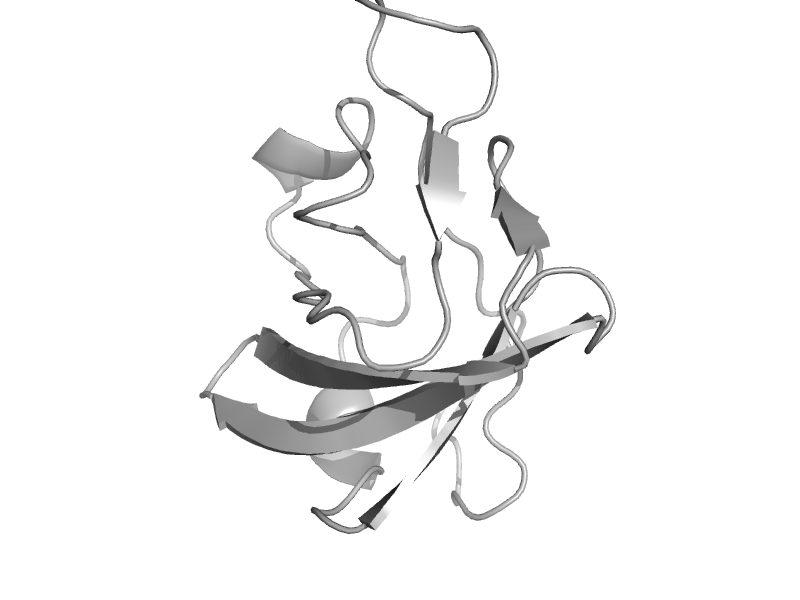 | view | |
| Pyruvate kinase (PK) | Pyruvate kinase beta-barrel domain | Trypanosome (Leishmania mexicana) [TaxId: 5665] | 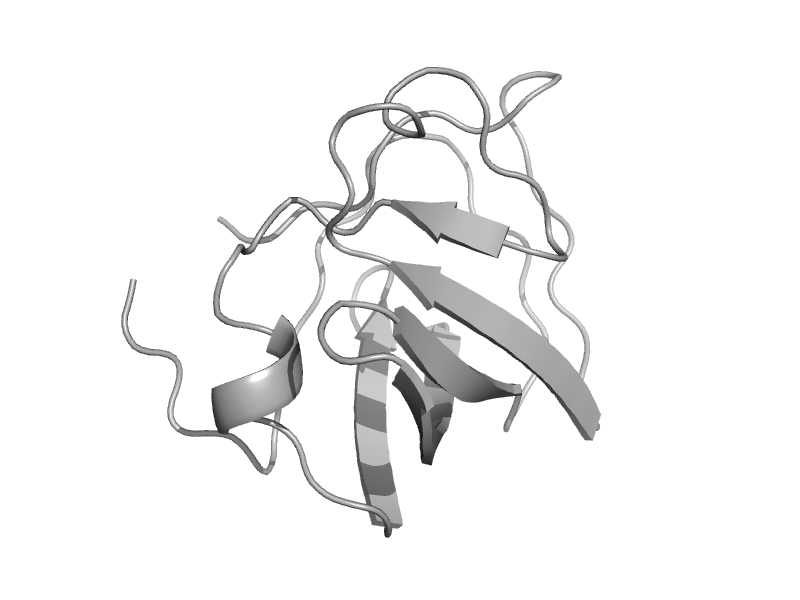 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | 230 | 240 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | E | I | R | T | M | K | L | E | G | G | - | - | N | D | V | S | L | - | A | G | Q | T | F | T | F | T | T | D | K | S | V | - | - | - | - | - | - | - | - | - | - | - | - | - | - | I | G | N | S | - | E | M | V | A | V | T | Y | E | G | F | T | T | D | L | S | V | G | N | T | V | L | V | D | D | G | L | I | G | M | E | V | T | A | I | - | E | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | K | V | I | C | K | V | L | N | N | G | D | L | G | E | N | K | G | V | N | L | P | G | V | S | I | A | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| F | L | V | E | R | E | Q | M | R | Y | P | V | D | V | Y | T | G | A | K | I | Q | V | D | G | - | - | E | L | M | L | T | E | L | G | L | E | G | D | E | Q | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | H | G | G | P | D | R | A | L | C | H | Y | P | R | E | H | Y | L | Y | W | A | R | E | F | P | E | Q | A | E | L | F | V | A | P | A | F | G | E | N | L | S | T | D | G | L | T | E | S | N | V | Y | M | G | D | I | F | R | W | G | E | - | - | A | L | I | Q | V | S | Q | P | R | S | P | C | Y | K | L | N | Y | H | F | - | - | - | - | - | D | I | S | D | I | A | Q | L | M | Q | N | T | - | - | G | K | V | G | W | L | Y | S | V | I | A | P | G | K | V | S | A | D | A | P | L | E | L | V | S | R | V | S | D | V | T | V | Q | E | A | A | A | I | A | W | H | M | P | F | D | D | D | Q | Y | H | R | L | L | S | A | A | G | L | S | K | S | W | T | R | T | M | Q | K | R | R | L | S | G | K | I | E | D | F | S | R | R | L | W | G |
| - | - | - | W | K | R | T | A | K | A | E | G | L | Y | I | A | D | T | K | S | F | V | T | K | Q | D | K | L | D | F | D | Y | G | G | I | P | G | D | L | H | F | G | L | T | K | K | A | G | A | R | E | P | F | S | R | G | T | E | I | F | N | R | - | R | Q | I | S | I | V | S | I | E | E | C | N | E | I | A | L | K | - | - | - | G | V | P | R | I | L | P | E | W | L | G | A | N | V | A | V | S | G | P | D | L | T | S | L | K | E | G | S | R | I | I | F | P | S | G | - | A | A | L | L | C | E | G | E | N | D | P | C | I | Q | P | G | E | V | I | Q | S | Y | Y | P | D | Q | P | K | L | A | S | A | F | V | R | H | A | L | G | I | R | G | I | V | C | I | V | E | R | P | G | A | V | Y | T | G | D | E | I | E | V | H | S | Y | Q | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | E | I | R | T | G | L | I | K | G | S | G | T | A | E | V | E | L | K | K | G | A | T | L | K | I | T | L | D | N | A | Y | M | E | - | - | - | - | - | - | - | - | - | - | - | - | K | C | D | E | - | N | I | L | W | L | D | Y | K | N | I | C | K | V | V | D | V | G | S | K | V | Y | V | D | D | G | L | I | S | L | Q | V | K | Q | K | - | G | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | F | L | V | T | E | V | E | N | G | G | F | L | G | S | K | K | G | V | N | L | P | G | A | A | V | D | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | I | R | T | G | Q | F | V | G | - | - | - | G | D | A | V | M | E | R | G | A | T | C | Y | V | T | T | D | P | A | F | A | D | - | - | - | - | - | - | - | - | - | - | - | - | K | G | T | K | - | D | K | F | Y | I | D | Y | Q | N | L | S | K | V | V | R | P | G | N | Y | I | Y | I | D | D | G | I | L | I | L | Q | V | Q | S | H | E | D | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | T | L | E | C | T | V | T | N | S | H | T | I | S | D | R | R | G | V | N | L | P | G | C | D | V | D | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
