solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1n10a2 (1003 )
d2enga- ( 1 )
d2g5da1 ( 40 ) pdrpagipdpagttvagg---------------gAvYtVvphlsMphWaa
d2pi8a- ( 3 ) skptdrGqqykdgkftqpfslvn--qPdAvga
d2pnwa1 ( 5 ) fsidevsfrdLpgWgq
d3czba- ( 58 ) mpAalsfagLagWae
d4jp6a- ( 2 )
d5xbxa1 ( 1 )
60 70 80 90 100
d1n10a2 (1003 )
d2enga- ( 1 )
d2g5da1 ( 75 ) Q-dFakSLqSFrlGÇanLknrq------------gWqdVÇaqAfqt-pih
d2pi8a- ( 33 ) PiNagdFaeQInhIrnssprlyg-----------nqsnvYnaVqeWl---
d2pnwa1 ( 21 ) d-dPrkLfpAMatILshLrnakpyrtGalgItAaeLvsLLelAergqvns
d3czba- ( 73 ) E-dHlaALnAFraGÇgvsk-------------dpaaarVçglAkatkdld
d4jp6a- ( 2 )
d5xbxa1 ( 1 )
110 120 130 140 150
d1n10a2 (1003 )
d2enga- ( 1 )
d2g5da1 ( 111 ) sfqAkrFFeryFtPWqVag-ngslagtVtGyyEpvLkGdgrrterArfpI
d2pi8a- ( 69 ) ragGdTrnrqfgidAwQeGadnyGNVqFtGyyTpviqArhtrqgefqypi
d2pnwa1 ( 70 ) peqArqfFetnSVPFrIsp-----sGfVtAfYEpeLeVsatpddvwrypI
d3czba- ( 109 ) vsgAkafIeanFrVea---VdgGgdglLtAyfaPqyeArmsrnaeFsapL
d4jp6a- ( 2 )
d5xbxa1 ( 1 )
160 170 180 190 200
d1n10a2 (1003 )
d2enga- ( 1 )
d2g5da1 ( 160 ) YGiPddFIsvplplvrIrqtgknsGthtAdlsrFpitarTtaIkGrfegs
d2pi8a- ( 121 ) yrPp--------------------------krgrl---------------
d2pnwa1 ( 119 ) YrrPpeLvdIdn---------dnrpd-gFdp-------sya--fGkadee
d3czba- ( 156 ) rglpadLvvldlGpfe--------------palvg-----kkitGhvegs
d4jp6a- ( 2 )
d5xbxa1 ( 1 )
210 220 230 240 250
d1n10a2 (1003 ) pkvppgp---------nitatygd----kw
d2enga- ( 1 )
d2g5da1 ( 225 ) rFlpYhtRnqInggaLdgkApilGyAeDpVelFfMHiqgsGRLktpsg-k
d2pi8a- ( 131 ) -----ssRaeIyagaLsd-kyIlAySnsl-dnfi-DvqgsgyIdfgdgsp
d2pnwa1 ( 150 ) gisyFpdRraIdegcLrgrgLeIAwArskvdlFfVHvqgaArLvfpdg-a
d3czba- ( 187 ) tfvpypdRaeieatpsd----plAwM-rpEeLFfLQiqgsGvLvLpdg-r
d4jp6a- ( 2 ) sa
d5xbxa1 ( 1 ) aqlÇqpd---------ahgVrrfn----gr
260 270 280 290 300
d1n10a2 (1020 ) ldAkStwyg----------------------ggaÇ--gykdvd-------
d2enga- ( 1 ) adgrStryWdÇçKpsÇGwak---------kApvnqpVfsÇnanfqritdf
d2g5da1 ( 274 ) yirIgyAdkNehpyvsIGryMadkgylklgqtsmqgIkayMrqnp-qrla
d2pi8a- ( 175 ) lnFFsyagkNghayrsIgkVLidrgev------sqairhwgethseaevr
d2pnwa1 ( 199 ) ikrITyaAkAGHvfspIgrlLldrgeldpktismqtirqwLadhp-deVd
d3czba- ( 232 ) rvrAvfagtNgkpfvgiaiaMrdkglla------dairtwLaehrgpeAd
d4jp6a- ( 4 ) snVrAtyhfYnAqqngwdLr---------kvsAyÇatwdAdkp-------
d5xbxa1 ( 18 ) pÇASTtryvdghkgaÇGÇGq---------k-----------------gsd
bbbbbbbb
310 320 330 340 350
d1n10a2 (1049 ) ---------------------------kppfs-gmTGcGNtp-IfksgrG
d2enga- ( 42 ) daksGçepgGvAYsÇadQtPwavn-----ddfALGFAaTs-iagsneagw
d2g5da1 ( 323 ) eVlGqNpsYiFFrel---dgpvgaLgTpL--mgeYAGAIDr-----hyIT
d2pi8a- ( 225 ) eLleqNpSfvFfkpqs-fapVkgasaVpL--vgrASvASdr-----siIp
d2pnwa1 ( 248 ) gvLwhNrSYiFFrea----gPiaaAkVpL--vagrALAVdr-----liHT
d3czba- ( 282 ) aiMrlNprYVFFrtvpDdgePagaAgvaL--ppgrAIAVdp-----gyHa
d4jp6a- ( 38 ) ---------------------------yswrskygWTAFçGpvgphgraA
d5xbxa1 ( 42 ) tPfpwNl-qkhVTAPSerYFDdggsnlwçGknÇGkÇVrLt-PTgGfvpg-
bbb
360 370 380 390 400
d1n10a2 (1070 ) çgSÇfeIkÇtk-peAÇsg---epvvVhItddneepiapyHFdLsghAFGa
d2enga- ( 86 ) ÇÇAÇYeLtFts-gpVa-g---kkMVVQSTstnhFdLnIPgGgvgifdGÇt
d2g5da1 ( 368 ) LGAPLFVaTa---hPvt-rkaLNrLIMAQdtgs-----------------
d2pi8a- ( 267 ) pGtTLIaEvPlLdnnGkfngqyeLrLVALdvgg-----------------
d2pnwa1 ( 294 ) fGLPFFIhAptLthldd-gkpFarLMLALdtgs-----------------
d3czba- ( 326 ) yGgFYWLdAa--------fpvyrrAVTALdtgg-----------------
d4jp6a- ( 61 ) ÇgkÇLrVtNtk-----tr---aettVRIVdqç---sng-GLdLDwsvFkk
d5xbxa1 ( 89 ) ----kGgaPpn------h---npvvFMVTnAçpin---------------
bbbbbb bbbbbbbb
410 420 430 440 450
d1n10a2 (1116 ) MAkk------------------------------------gdeqkLrsag
d2enga- ( 137 ) pQfggLpgqrygGIssrneÇdrFpdaLkp--------GÇyWRfdWFknAd
d2g5da1 ( 397 ) -----------aIk------------gAVrVdYFWgyg-deagelAgkQ-
d2pi8a- ( 301 ) -----------aik------------g-qhFAIYQgig-peAGhrAgwY-
d2pnwa1 ( 326 ) -----------aIv------------gpArGdIFTgsg-feaGelAgtV-
d3czba- ( 358 ) -----------aik------------geVrAdLYmgsg-avagveAgrV-
d4jp6a- ( 99 ) LDtd------------------------------------g---sGylrg
d5xbxa1 ( 111 ) ---g---neewÇGIsgkpgtnhvNshgYEVHFdLQ--dqvgqVeal-hWd
aaaaa
460 470 480
d1n10a2 (1130 ) eLeLqFrrvkçkypeg
d2enga- ( 179 ) nPsFsfrqVqÇpaeLvarTgÇrRndDgnfpav
d2g5da1 ( 422 ) kttGyVWQLLpNgmkPeyrp
d2pi8a- ( 325 ) nhyGrVWvlktap
d2pnwa1 ( 351 ) rneAdFYILLPriAAeryr
d3czba- ( 383 ) rhtLrLYrLtpnp
d4jp6a- ( 110 ) hLiVnYqfvnçg---n
d5xbxa1 ( 152 ) nPEVTweevpçpgdLqanyq-qÇeÇhns
bbbbbbbb
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Pollen allergen PHL P 1 N-terminal domain | Pollen allergen PHL P 1 N-terminal domain | Timothy grass (Phleum pratense) [TaxId: 15957] |  | view | |
| Endoglucanase V (Eng V) | Eng V-like | Fungus (Humicola insolens) [TaxId: 34413] |  | view | |
| Membrane-bound lytic murein transglycosylase A, MLTA | MLTA-like | Neisseria gonorrhoeae [TaxId: 485] |  | view | |
| Membrane-bound lytic murein transglycosylase A, MLTA | MLTA-like | Escherichia coli [TaxId: 562] |  | view | |
| Membrane-bound lytic murein transglycosylase A, MLTA | MLTA-like | Agrobacterium tumefaciens [TaxId: 358] | 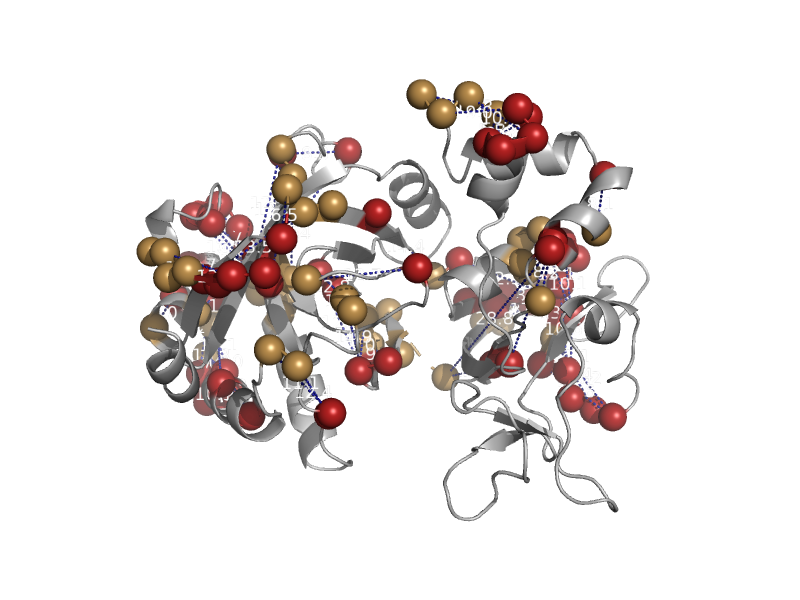 | view | |
| automated matches | automated matches | Caulobacter crescentus [TaxId: 190650] | 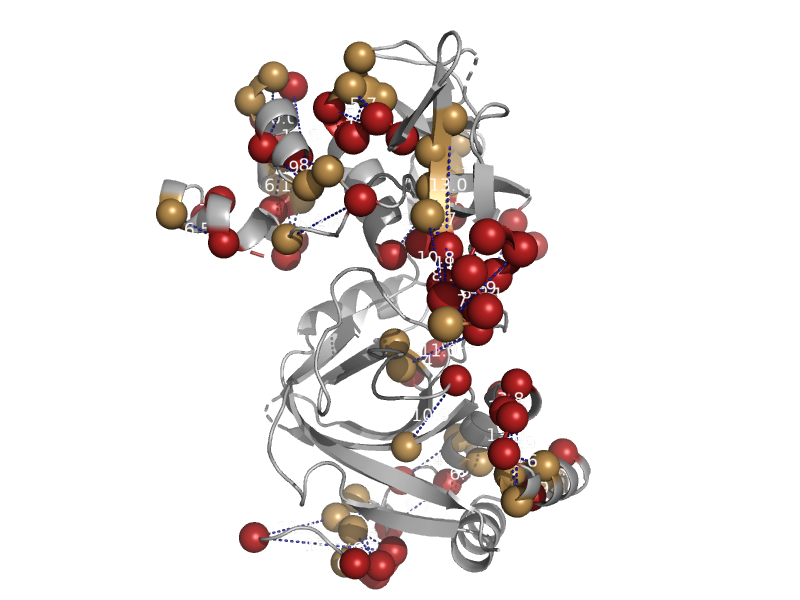 | view | |
| automated matches | Barwin | Papaya (Carica papaya) [TaxId: 3649] | 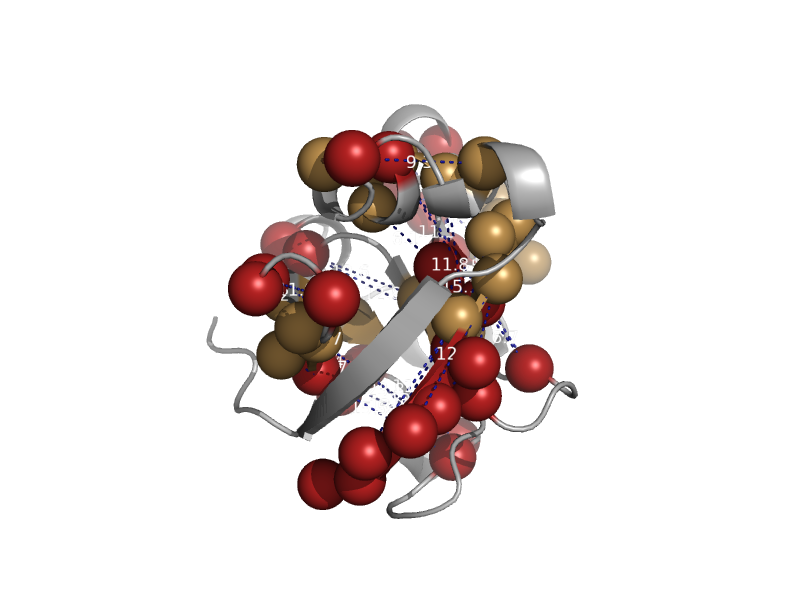 | view | |
| automated matches | automated matches | Ampullaria crossean [TaxId: 228791] | 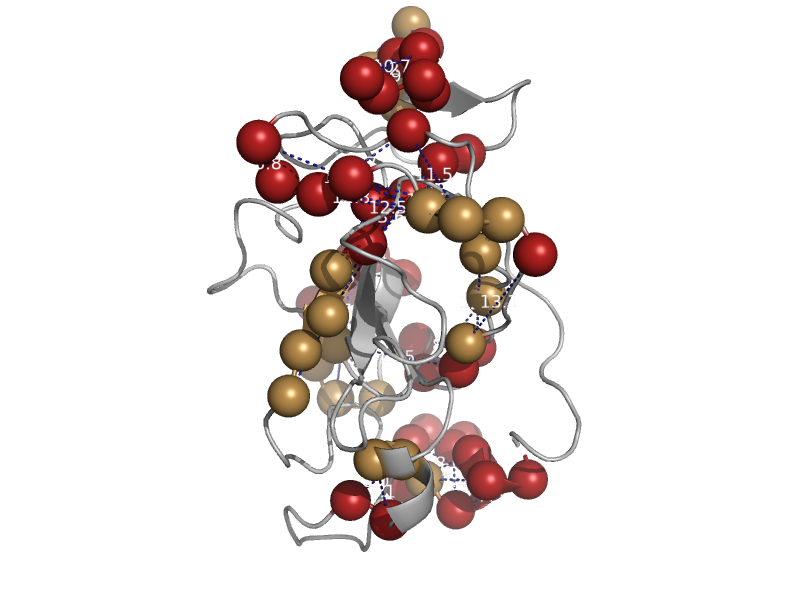 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | 230 | 240 | 250 | 260 | 270 | 280 | 290 | 300 | 310 | 320 | 330 | 340 | 350 | 360 | 370 | 380 | 390 | 400 | 410 | 420 | 430 | 440 | 450 | 460 | 470 | 480 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | K | V | P | P | G | P | - | - | - | - | - | - | - | - | - | N | I | T | A | T | Y | G | D | - | - | - | - | K | W | L | D | A | K | S | T | W | Y | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | G | A | C | - | - | G | Y | K | D | V | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | P | P | F | S | - | G | M | T | G | C | G | N | T | P | - | I | F | K | S | G | R | G | C | G | S | C | F | E | I | K | C | T | K | - | P | E | A | C | S | G | - | - | - | E | P | V | V | V | H | I | T | D | D | N | E | E | P | I | A | P | Y | H | F | D | L | S | G | H | A | F | G | A | M | A | K | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | D | E | Q | K | L | R | S | A | G | E | L | E | L | Q | F | R | R | V | K | C | K | Y | P | E | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | D | G | R | S | T | R | Y | W | D | C | C | K | P | S | C | G | W | A | K | - | - | - | - | - | - | - | - | - | K | A | P | V | N | Q | P | V | F | S | C | N | A | N | F | Q | R | I | T | D | F | D | A | K | S | G | C | E | P | G | G | V | A | Y | S | C | A | D | Q | T | P | W | A | V | N | - | - | - | - | - | D | D | F | A | L | G | F | A | A | T | S | - | I | A | G | S | N | E | A | G | W | C | C | A | C | Y | E | L | T | F | T | S | - | G | P | V | A | - | G | - | - | - | K | K | M | V | V | Q | S | T | S | T | N | H | F | D | L | N | I | P | G | G | G | V | G | I | F | D | G | C | T | P | Q | F | G | G | L | P | G | Q | R | Y | G | G | I | S | S | R | N | E | C | D | R | F | P | D | A | L | K | P | - | - | - | - | - | - | - | - | G | C | Y | W | R | F | D | W | F | K | N | A | D | N | P | S | F | S | F | R | Q | V | Q | C | P | A | E | L | V | A | R | T | G | C | R | R | N | D | D | G | N | F | P | A | V |
| P | D | R | P | A | G | I | P | D | P | A | G | T | T | V | A | G | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | A | V | Y | T | V | V | P | H | L | S | M | P | H | W | A | A | Q | - | D | F | A | K | S | L | Q | S | F | R | L | G | C | A | N | L | K | N | R | Q | - | - | - | - | - | - | - | - | - | - | - | - | G | W | Q | D | V | C | A | Q | A | F | Q | T | - | P | I | H | S | F | Q | A | K | R | F | F | E | R | Y | F | T | P | W | Q | V | A | G | - | N | G | S | L | A | G | T | V | T | G | Y | Y | E | P | V | L | K | G | D | G | R | R | T | E | R | A | R | F | P | I | Y | G | I | P | D | D | F | I | S | V | P | L | P | L | V | R | I | R | Q | T | G | K | N | S | G | T | H | T | A | D | L | S | R | F | P | I | T | A | R | T | T | A | I | K | G | R | F | E | G | S | R | F | L | P | Y | H | T | R | N | Q | I | N | G | G | A | L | D | G | K | A | P | I | L | G | Y | A | E | D | P | V | E | L | F | F | M | H | I | Q | G | S | G | R | L | K | T | P | S | G | - | K | Y | I | R | I | G | Y | A | D | K | N | E | H | P | Y | V | S | I | G | R | Y | M | A | D | K | G | Y | L | K | L | G | Q | T | S | M | Q | G | I | K | A | Y | M | R | Q | N | P | - | Q | R | L | A | E | V | L | G | Q | N | P | S | Y | I | F | F | R | E | L | - | - | - | D | G | P | V | G | A | L | G | T | P | L | - | - | M | G | E | Y | A | G | A | I | D | R | - | - | - | - | - | H | Y | I | T | L | G | A | P | L | F | V | A | T | A | - | - | - | H | P | V | T | - | R | K | A | L | N | R | L | I | M | A | Q | D | T | G | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | I | K | - | - | - | - | - | - | - | - | - | - | - | - | G | A | V | R | V | D | Y | F | W | G | Y | G | - | D | E | A | G | E | L | A | G | K | Q | - | K | T | T | G | Y | V | W | Q | L | L | P | N | G | M | K | P | E | Y | R | P | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | K | P | T | D | R | G | Q | Q | Y | K | D | G | K | F | T | Q | P | F | S | L | V | N | - | - | Q | P | D | A | V | G | A | P | I | N | A | G | D | F | A | E | Q | I | N | H | I | R | N | S | S | P | R | L | Y | G | - | - | - | - | - | - | - | - | - | - | - | N | Q | S | N | V | Y | N | A | V | Q | E | W | L | - | - | - | R | A | G | G | D | T | R | N | R | Q | F | G | I | D | A | W | Q | E | G | A | D | N | Y | G | N | V | Q | F | T | G | Y | Y | T | P | V | I | Q | A | R | H | T | R | Q | G | E | F | Q | Y | P | I | Y | R | P | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | R | G | R | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | S | R | A | E | I | Y | A | G | A | L | S | D | - | K | Y | I | L | A | Y | S | N | S | L | - | D | N | F | I | - | D | V | Q | G | S | G | Y | I | D | F | G | D | G | S | P | L | N | F | F | S | Y | A | G | K | N | G | H | A | Y | R | S | I | G | K | V | L | I | D | R | G | E | V | - | - | - | - | - | - | S | Q | A | I | R | H | W | G | E | T | H | S | E | A | E | V | R | E | L | L | E | Q | N | P | S | F | V | F | F | K | P | Q | S | - | F | A | P | V | K | G | A | S | A | V | P | L | - | - | V | G | R | A | S | V | A | S | D | R | - | - | - | - | - | S | I | I | P | P | G | T | T | L | I | A | E | V | P | L | L | D | N | N | G | K | F | N | G | Q | Y | E | L | R | L | V | A | L | D | V | G | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | I | K | - | - | - | - | - | - | - | - | - | - | - | - | G | - | Q | H | F | A | I | Y | Q | G | I | G | - | P | E | A | G | H | R | A | G | W | Y | - | N | H | Y | G | R | V | W | V | L | K | T | A | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | F | S | I | D | E | V | S | F | R | D | L | P | G | W | G | Q | D | - | D | P | R | K | L | F | P | A | M | A | T | I | L | S | H | L | R | N | A | K | P | Y | R | T | G | A | L | G | I | T | A | A | E | L | V | S | L | L | E | L | A | E | R | G | Q | V | N | S | P | E | Q | A | R | Q | F | F | E | T | N | S | V | P | F | R | I | S | P | - | - | - | - | - | S | G | F | V | T | A | F | Y | E | P | E | L | E | V | S | A | T | P | D | D | V | W | R | Y | P | I | Y | R | R | P | P | E | L | V | D | I | D | N | - | - | - | - | - | - | - | - | - | D | N | R | P | D | - | G | F | D | P | - | - | - | - | - | - | - | S | Y | A | - | - | F | G | K | A | D | E | E | G | I | S | Y | F | P | D | R | R | A | I | D | E | G | C | L | R | G | R | G | L | E | I | A | W | A | R | S | K | V | D | L | F | F | V | H | V | Q | G | A | A | R | L | V | F | P | D | G | - | A | I | K | R | I | T | Y | A | A | K | A | G | H | V | F | S | P | I | G | R | L | L | L | D | R | G | E | L | D | P | K | T | I | S | M | Q | T | I | R | Q | W | L | A | D | H | P | - | D | E | V | D | G | V | L | W | H | N | R | S | Y | I | F | F | R | E | A | - | - | - | - | G | P | I | A | A | A | K | V | P | L | - | - | V | A | G | R | A | L | A | V | D | R | - | - | - | - | - | L | I | H | T | F | G | L | P | F | F | I | H | A | P | T | L | T | H | L | D | D | - | G | K | P | F | A | R | L | M | L | A | L | D | T | G | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | I | V | - | - | - | - | - | - | - | - | - | - | - | - | G | P | A | R | G | D | I | F | T | G | S | G | - | F | E | A | G | E | L | A | G | T | V | - | R | N | E | A | D | F | Y | I | L | L | P | R | I | A | A | E | R | Y | R | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | P | A | A | L | S | F | A | G | L | A | G | W | A | E | E | - | D | H | L | A | A | L | N | A | F | R | A | G | C | G | V | S | K | - | - | - | - | - | - | - | - | - | - | - | - | - | D | P | A | A | A | R | V | C | G | L | A | K | A | T | K | D | L | D | V | S | G | A | K | A | F | I | E | A | N | F | R | V | E | A | - | - | - | V | D | G | G | G | D | G | L | L | T | A | Y | F | A | P | Q | Y | E | A | R | M | S | R | N | A | E | F | S | A | P | L | R | G | L | P | A | D | L | V | V | L | D | L | G | P | F | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | A | L | V | G | - | - | - | - | - | K | K | I | T | G | H | V | E | G | S | T | F | V | P | Y | P | D | R | A | E | I | E | A | T | P | S | D | - | - | - | - | P | L | A | W | M | - | R | P | E | E | L | F | F | L | Q | I | Q | G | S | G | V | L | V | L | P | D | G | - | R | R | V | R | A | V | F | A | G | T | N | G | K | P | F | V | G | I | A | I | A | M | R | D | K | G | L | L | A | - | - | - | - | - | - | D | A | I | R | T | W | L | A | E | H | R | G | P | E | A | D | A | I | M | R | L | N | P | R | Y | V | F | F | R | T | V | P | D | D | G | E | P | A | G | A | A | G | V | A | L | - | - | P | P | G | R | A | I | A | V | D | P | - | - | - | - | - | G | Y | H | A | Y | G | G | F | Y | W | L | D | A | A | - | - | - | - | - | - | - | - | F | P | V | Y | R | R | A | V | T | A | L | D | T | G | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | I | K | - | - | - | - | - | - | - | - | - | - | - | - | G | E | V | R | A | D | L | Y | M | G | S | G | - | A | V | A | G | V | E | A | G | R | V | - | R | H | T | L | R | L | Y | R | L | T | P | N | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | A | S | N | V | R | A | T | Y | H | F | Y | N | A | Q | Q | N | G | W | D | L | R | - | - | - | - | - | - | - | - | - | K | V | S | A | Y | C | A | T | W | D | A | D | K | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Y | S | W | R | S | K | Y | G | W | T | A | F | C | G | P | V | G | P | H | G | R | A | A | C | G | K | C | L | R | V | T | N | T | K | - | - | - | - | - | T | R | - | - | - | A | E | T | T | V | R | I | V | D | Q | C | - | - | - | S | N | G | - | G | L | D | L | D | W | S | V | F | K | K | L | D | T | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | - | - | - | S | G | Y | L | R | G | H | L | I | V | N | Y | Q | F | V | N | C | G | - | - | - | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | Q | L | C | Q | P | D | - | - | - | - | - | - | - | - | - | A | H | G | V | R | R | F | N | - | - | - | - | G | R | P | C | A | S | T | T | R | Y | V | D | G | H | K | G | A | C | G | C | G | Q | - | - | - | - | - | - | - | - | - | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | S | D | T | P | F | P | W | N | L | - | Q | K | H | V | T | A | P | S | E | R | Y | F | D | D | G | G | S | N | L | W | C | G | K | N | C | G | K | C | V | R | L | T | - | P | T | G | G | F | V | P | G | - | - | - | - | - | K | G | G | A | P | P | N | - | - | - | - | - | - | H | - | - | - | N | P | V | V | F | M | V | T | N | A | C | P | I | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | - | - | - | N | E | E | W | C | G | I | S | G | K | P | G | T | N | H | V | N | S | H | G | Y | E | V | H | F | D | L | Q | - | - | D | Q | V | G | Q | V | E | A | L | - | H | W | D | N | P | E | V | T | W | E | E | V | P | C | P | G | D | L | Q | A | N | Y | Q | - | Q | C | E | C | H | N | S | - | - | - | - |
