solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1ffya2 ( 201 ) hdkrSAsIyVAFnVkddkGvvdadAkfIIwtttpwtipSnVaITvhpe--
d1h3na2 ( 226 ) rseGAeIlFpVeg------kevrIpVFttrPDTLfGAtFLVLAPehp
d1wkaa- ( 195 ) gklyTLrYeVeg----g---gfIeIAtvrPETVFADqAIAVhpeDe
d1wnya- ( 202 ) dpsVyVrFpLkepkklgLekASLLIwtttPwtLPGNVAAAVhpeyt
bbbbbbbb bbbbbb 333333 bbbb
60 70 80 90 100
d1ffya2 ( 249 ) ---lkyGqYnvngekYiiaeaLsdaVaeaLdwdka-----siklekeytg
d1h3na2 ( 267 ) lTleLAa---pek------reeVlayveaAkrkteierqaegrektGvf-
d1wkaa- ( 234 ) ------------------------------------------------ry
d1wnya- ( 248 ) ----YAAFqvgd-eALILEeglGrkllge-----------gtpvlktfpG
aaaa
110 120 130 140 150
d1ffya2 ( 291 ) kelewvvAqHpfldr--esLvINgdhvttda-gTgcvHtAPghgeddyiv
d1h3na2 ( 307 ) ---LgayAlNpAt---gerIpIwTAdyVlfgygtGAiMAVPAHdqrDYeF
d1wkaa- ( 236 ) rhllgkrArIPlt---evwIpIlaDpaVekdfgtGAlkVTPahdpldyeI
d1wnya- ( 282 ) kaLeglpYtPPYpqaLekGyfVVladyVsqedgTGIvHQAPafgaedlet
bbb bbb aaaaaa
160 170 180 190 200
d1ffya2 ( 338 ) gqqyelpvISpidd---------------kgvfte--eggqfegmfydkA
d1h3na2 ( 351 ) ArkfglpikkVIerpgepLpeplerAyeepgiMvn---SgpfdgteSeeG
d1wkaa- ( 283 ) GerhglkpvsVinl---------------egrMegerVpeaLrgldrfeA
d1wnya- ( 332 ) ArvygLpllkTvde---------------egkLlve----pfkglyfreA
aaaa aaaa
210 220
d1ffya2 ( 371 ) NkavTdllTekgAlLkldfithsy
d1h3na2 ( 398 ) kr-kViawLeekglgkgrvty
d1wkaa- ( 318 ) rr-kAVelFreaghLvkeedy
d1wnya- ( 363 ) nr-aIlrdLrgrglLfkees
aa aaaaaaaa bbbbbb
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Isoleucyl-tRNA synthetase (IleRS) | ValRS/IleRS/LeuRS editing domain | Staphylococcus aureus [TaxId: 1280] | 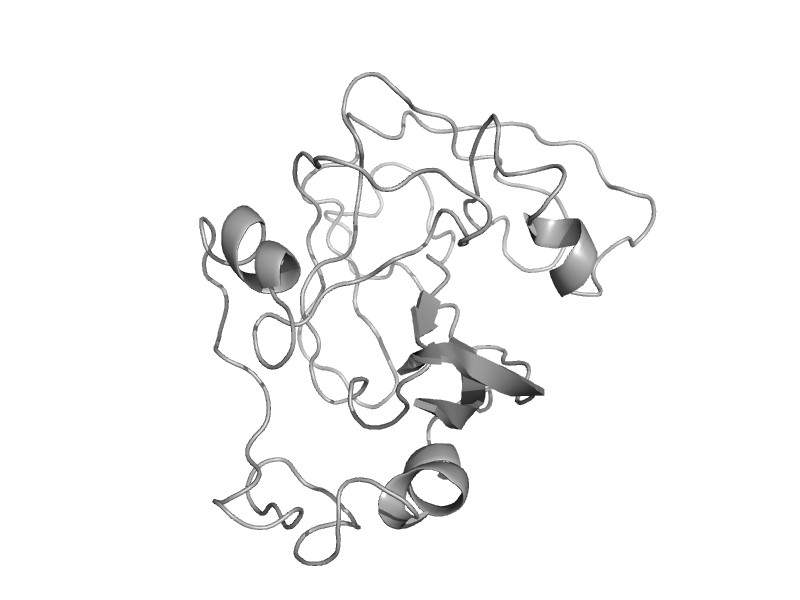 | view | |
| Leucyl-tRNA synthetase (LeuRS) | ValRS/IleRS/LeuRS editing domain | Thermus thermophilus [TaxId: 274] | 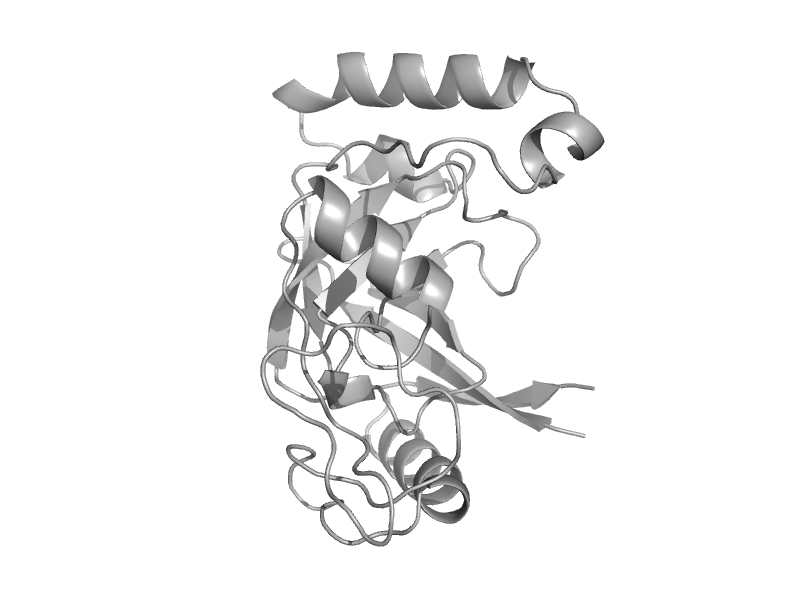 | view | |
| automated matches | ValRS/IleRS/LeuRS editing domain | Thermus thermophilus [TaxId: 274] | 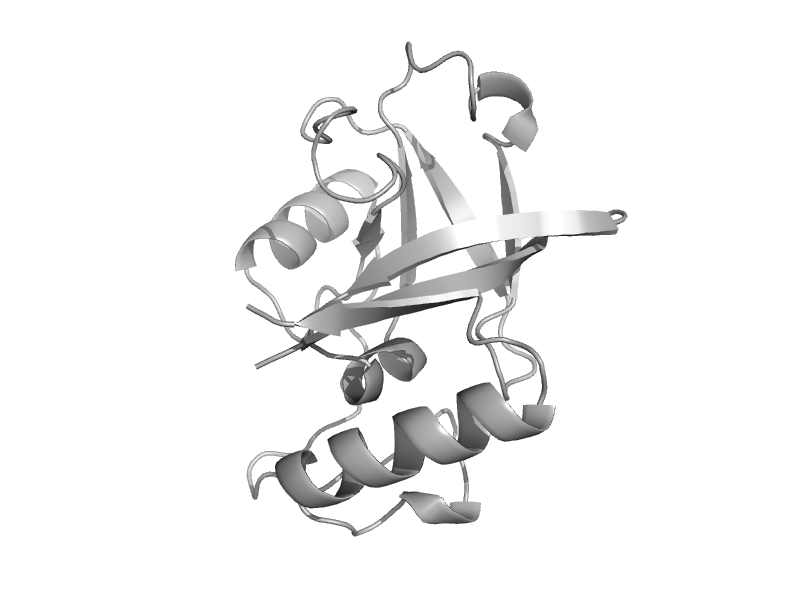 | view | |
| automated matches | ValRS/IleRS/LeuRS editing domain | Thermus thermophilus [TaxId: 274] | 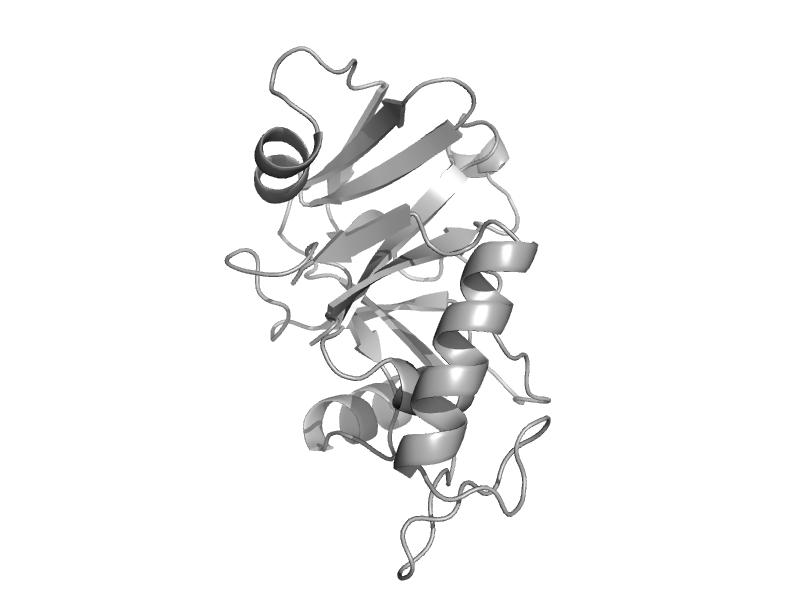 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| H | D | K | R | S | A | S | I | Y | V | A | F | N | V | K | D | D | K | G | V | V | D | A | D | A | K | F | I | I | W | T | T | T | P | W | T | I | P | S | N | V | A | I | T | V | H | P | E | - | - | - | - | - | L | K | Y | G | Q | Y | N | V | N | G | E | K | Y | I | I | A | E | A | L | S | D | A | V | A | E | A | L | D | W | D | K | A | - | - | - | - | - | S | I | K | L | E | K | E | Y | T | G | K | E | L | E | W | V | V | A | Q | H | P | F | L | D | R | - | - | E | S | L | V | I | N | G | D | H | V | T | T | D | A | - | G | T | G | C | V | H | T | A | P | G | H | G | E | D | D | Y | I | V | G | Q | Q | Y | E | L | P | V | I | S | P | I | D | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | G | V | F | T | E | - | - | E | G | G | Q | F | E | G | M | F | Y | D | K | A | N | K | A | V | T | D | L | L | T | E | K | G | A | L | L | K | L | D | F | I | T | H | S | Y |
| - | - | - | R | S | E | G | A | E | I | L | F | P | V | E | G | - | - | - | - | - | - | K | E | V | R | I | P | V | F | T | T | R | P | D | T | L | F | G | A | T | F | L | V | L | A | P | E | H | P | L | T | L | E | L | A | A | - | - | - | P | E | K | - | - | - | - | - | - | R | E | E | V | L | A | Y | V | E | A | A | K | R | K | T | E | I | E | R | Q | A | E | G | R | E | K | T | G | V | F | - | - | - | - | L | G | A | Y | A | L | N | P | A | T | - | - | - | G | E | R | I | P | I | W | T | A | D | Y | V | L | F | G | Y | G | T | G | A | I | M | A | V | P | A | H | D | Q | R | D | Y | E | F | A | R | K | F | G | L | P | I | K | K | V | I | E | R | P | G | E | P | L | P | E | P | L | E | R | A | Y | E | E | P | G | I | M | V | N | - | - | - | S | G | P | F | D | G | T | E | S | E | E | G | K | R | - | K | V | I | A | W | L | E | E | K | G | L | G | K | G | R | V | T | Y | - | - | - |
| - | - | - | - | G | K | L | Y | T | L | R | Y | E | V | E | G | - | - | - | - | G | - | - | - | G | F | I | E | I | A | T | V | R | P | E | T | V | F | A | D | Q | A | I | A | V | H | P | E | D | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | Y | R | H | L | L | G | K | R | A | R | I | P | L | T | - | - | - | E | V | W | I | P | I | L | A | D | P | A | V | E | K | D | F | G | T | G | A | L | K | V | T | P | A | H | D | P | L | D | Y | E | I | G | E | R | H | G | L | K | P | V | S | V | I | N | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | G | R | M | E | G | E | R | V | P | E | A | L | R | G | L | D | R | F | E | A | R | R | - | K | A | V | E | L | F | R | E | A | G | H | L | V | K | E | E | D | Y | - | - | - |
| - | - | - | - | D | P | S | V | Y | V | R | F | P | L | K | E | P | K | K | L | G | L | E | K | A | S | L | L | I | W | T | T | T | P | W | T | L | P | G | N | V | A | A | A | V | H | P | E | Y | T | - | - | - | - | Y | A | A | F | Q | V | G | D | - | E | A | L | I | L | E | E | G | L | G | R | K | L | L | G | E | - | - | - | - | - | - | - | - | - | - | - | G | T | P | V | L | K | T | F | P | G | K | A | L | E | G | L | P | Y | T | P | P | Y | P | Q | A | L | E | K | G | Y | F | V | V | L | A | D | Y | V | S | Q | E | D | G | T | G | I | V | H | Q | A | P | A | F | G | A | E | D | L | E | T | A | R | V | Y | G | L | P | L | L | K | T | V | D | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | G | K | L | L | V | E | - | - | - | - | P | F | K | G | L | Y | F | R | E | A | N | R | - | A | I | L | R | D | L | R | G | R | G | L | L | F | K | E | E | S | - | - | - | - |
