solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1bu8a1 ( 337 ) rwrYkVsVtLsg------akklsgyIlVALyGnn--------gnS
d1ca1a2 ( 250 ) svgknVkeLvAyIsTSgek----dAgTdDyMyFGIkTkd--------gkt
d2p0ma2 ( 2 ) gvyrVcVsTGasi----yAgSkNkVeLwLvGqh--------geV
d3pzwa1 ( 7 ) kikGtVvLmpknelnlnaflgrsVSLqLiSatkadahgkgkvg
d3v98a1 ( 5 ) sYtvtvAtGsqe----hAgtdDyiylSlvGsa--------gcS
bbbbbbb bbbbbbbb
60 70 80 90 100
d1bu8a1 ( 367 ) kqyeIfk-----gsLkpearhvrdIdVd---inVgeIqkVkFlWn-----
d1ca1a2 ( 288 ) qeweMdnp---gndFmtgskdtYtFkLkdenlkiddIqnMwIrKrkytaf
d2p0ma2 ( 34 ) eLgscl-------rPtrnkeeefkvnvs---kyLgsLlfVrLrKkhfl-k
d3pzwa1 ( 60 ) kdtfLegintslptlgagesAFnIhFeWdgsmg--iPgAFyIkNymq---
d3v98a1 ( 36 ) ekhlldk-----gsFergavdsydvtvd---eelgeiqlvrieKrkyg-s
bb bbbbbbb bbbbbbbbb
110 120 130 140
d1bu8a1 ( 413 ) -ptLGAsqItVqsg-vdgkeynFçssdtvred----vlQsLypç
d1ca1a2 ( 335 ) pdaYkPenIkViAng--kvvvdkdInewIsgn----stynIk
d2p0ma2 ( 73 ) edaWfCnwISVqAlgaaedkywFpCyrwVvgd----gvqsLpvg
d3pzwa1 ( 105 ) -veFfLksLtLea-----gtirFvCnswVyntklyksvriffAnhty
d3v98a1 ( 77 ) nddWylkyitlktph--gdyieFpCyrwitGd----vevvlrdg
bbbbbbbb bbbb bbbb
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Pancreatic lipase, C-terminal domain | Colipase-binding domain | Norway rat (Rattus norvegicus) [TaxId: 10116] |  | view | |
| Alpha-toxin, C-terminal domain | Alpha-toxin, C-terminal domain | Clostridium perfringens, different strains [TaxId: 1502] | 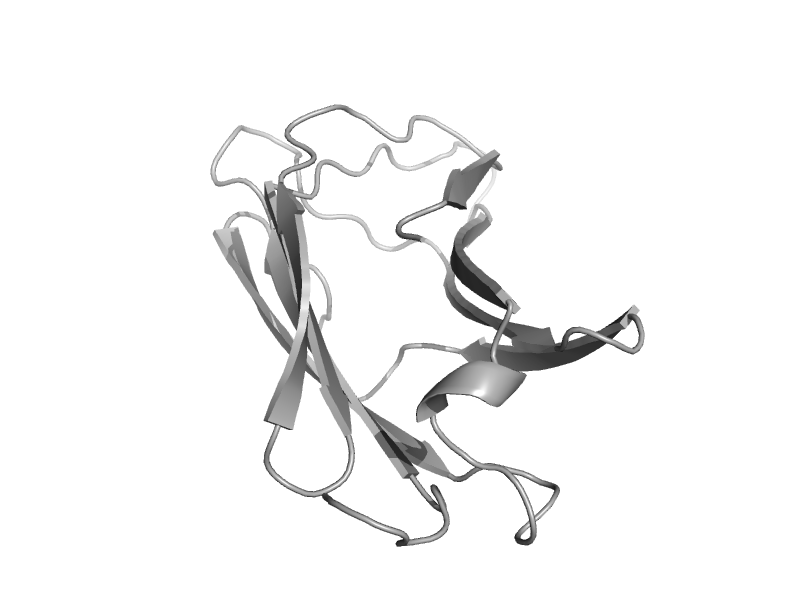 | view | |
| automated matches | automated matches | Rabbit (Oryctolagus cuniculus) [TaxId: 9986] | 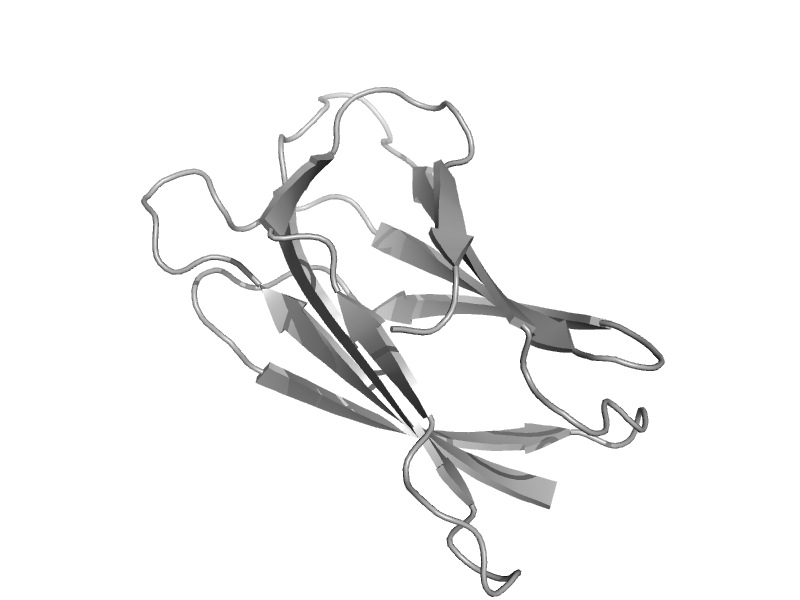 | view | |
| Plant lipoxigenase | Lipoxigenase N-terminal domain | Soybean (Glycine max), isozyme L1 [TaxId: 3847] | 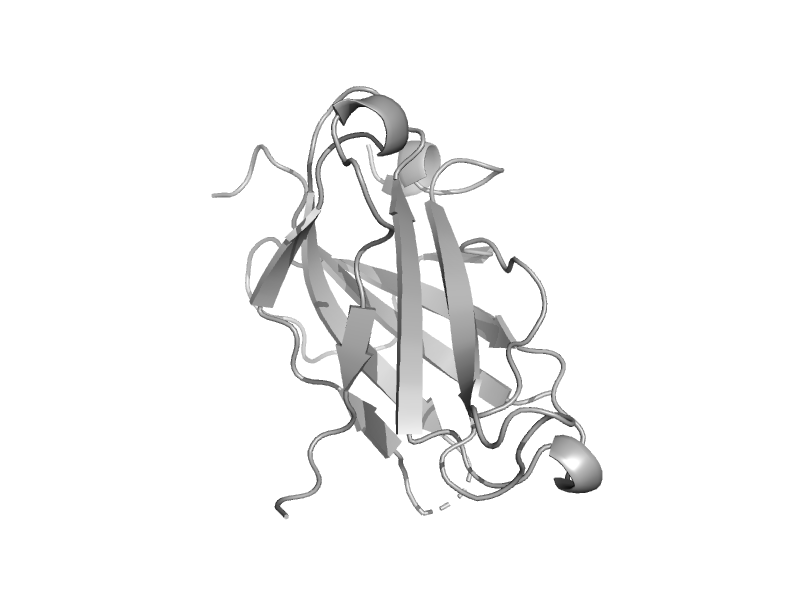 | view | |
| automated matches | automated matches | Human (Homo sapiens) [TaxId: 9606] | 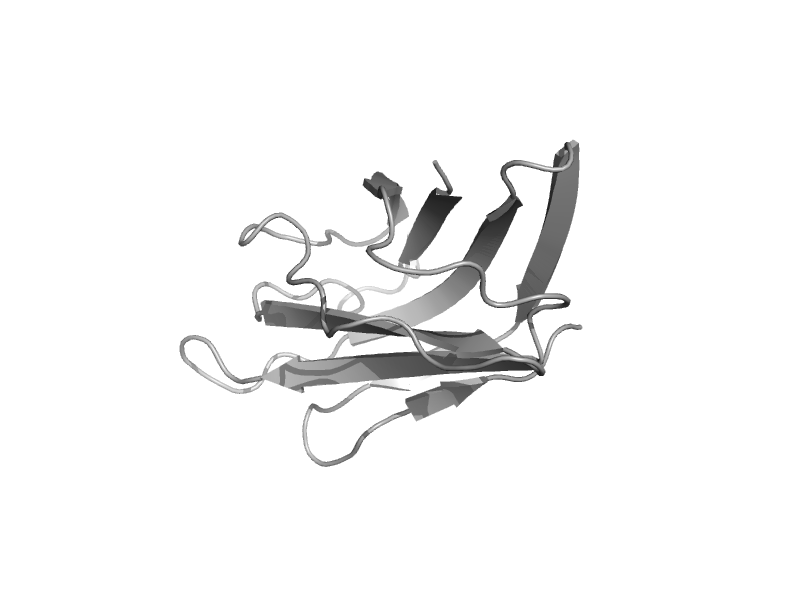 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | R | W | R | Y | K | V | S | V | T | L | S | G | - | - | - | - | - | - | A | K | K | L | S | G | Y | I | L | V | A | L | Y | G | N | N | - | - | - | - | - | - | - | - | G | N | S | K | Q | Y | E | I | F | K | - | - | - | - | - | G | S | L | K | P | E | A | R | H | V | R | D | I | D | V | D | - | - | - | I | N | V | G | E | I | Q | K | V | K | F | L | W | N | - | - | - | - | - | - | P | T | L | G | A | S | Q | I | T | V | Q | S | G | - | V | D | G | K | E | Y | N | F | C | S | S | D | T | V | R | E | D | - | - | - | - | V | L | Q | S | L | Y | P | C | - | - | - |
| S | V | G | K | N | V | K | E | L | V | A | Y | I | S | T | S | G | E | K | - | - | - | - | D | A | G | T | D | D | Y | M | Y | F | G | I | K | T | K | D | - | - | - | - | - | - | - | - | G | K | T | Q | E | W | E | M | D | N | P | - | - | - | G | N | D | F | M | T | G | S | K | D | T | Y | T | F | K | L | K | D | E | N | L | K | I | D | D | I | Q | N | M | W | I | R | K | R | K | Y | T | A | F | P | D | A | Y | K | P | E | N | I | K | V | I | A | N | G | - | - | K | V | V | V | D | K | D | I | N | E | W | I | S | G | N | - | - | - | - | S | T | Y | N | I | K | - | - | - | - | - |
| - | - | - | - | - | - | G | V | Y | R | V | C | V | S | T | G | A | S | I | - | - | - | - | Y | A | G | S | K | N | K | V | E | L | W | L | V | G | Q | H | - | - | - | - | - | - | - | - | G | E | V | E | L | G | S | C | L | - | - | - | - | - | - | - | R | P | T | R | N | K | E | E | E | F | K | V | N | V | S | - | - | - | K | Y | L | G | S | L | L | F | V | R | L | R | K | K | H | F | L | - | K | E | D | A | W | F | C | N | W | I | S | V | Q | A | L | G | A | A | E | D | K | Y | W | F | P | C | Y | R | W | V | V | G | D | - | - | - | - | G | V | Q | S | L | P | V | G | - | - | - |
| - | - | - | - | - | - | - | K | I | K | G | T | V | V | L | M | P | K | N | E | L | N | L | N | A | F | L | G | R | S | V | S | L | Q | L | I | S | A | T | K | A | D | A | H | G | K | G | K | V | G | K | D | T | F | L | E | G | I | N | T | S | L | P | T | L | G | A | G | E | S | A | F | N | I | H | F | E | W | D | G | S | M | G | - | - | I | P | G | A | F | Y | I | K | N | Y | M | Q | - | - | - | - | V | E | F | F | L | K | S | L | T | L | E | A | - | - | - | - | - | G | T | I | R | F | V | C | N | S | W | V | Y | N | T | K | L | Y | K | S | V | R | I | F | F | A | N | H | T | Y |
| - | - | - | - | - | - | - | S | Y | T | V | T | V | A | T | G | S | Q | E | - | - | - | - | H | A | G | T | D | D | Y | I | Y | L | S | L | V | G | S | A | - | - | - | - | - | - | - | - | G | C | S | E | K | H | L | L | D | K | - | - | - | - | - | G | S | F | E | R | G | A | V | D | S | Y | D | V | T | V | D | - | - | - | E | E | L | G | E | I | Q | L | V | R | I | E | K | R | K | Y | G | - | S | N | D | D | W | Y | L | K | Y | I | T | L | K | T | P | H | - | - | G | D | Y | I | E | F | P | C | Y | R | W | I | T | G | D | - | - | - | - | V | E | V | V | L | R | D | G | - | - | - |
