solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1a3qa2 ( 37 ) gpyLvIveqPkqrgfrFry-
d1bg1a2 ( 322 ) vVe-rqPcMpmhpdrpLVIktgvqFtTkVr
d1eaqa- ( 50 ) svevladhpgeLvrTdspnFlSS-vLpt----hwrsnktLp-iaFkVVal
d1mnna- ( 33 ) viltqlnedgttSnyF
d1nfia2 ( 20 ) yVeiieqPkqrgmrFry-
d1t4wa- ( 223 ) ekwMeIdVlkqkVa-kssdmAFaissehekyLWTkmgclVPIqVk
d1uura2 ( 360 ) pnVaLvLk-sqpFP--vViskgkqLgenqLvVlVl
d2as5m2 ( 396 ) plewplssqsgsyeLrievqPkp-hhrAhy-
d3brfa2 ( 197 )
d3d06a- ( 97 ) vpsqtygsygFrLgFltctySpalnkMFCqlatCpVqLwvd
d5qs9a- ( 41 )
b
60 70 80 90 100
d1a3qa2 ( 56 ) -g-------cegpshgGLpGasSek--------grktyPtVkIcnye---
d1bg1a2 ( 351 ) llvkfpeLn---yqLkIkVcIdk-------dsgdva---------alrGs
d1eaqa- ( 95 ) g-------------------------------------------------
d1mnna- ( 49 ) dkrklkiaprstlqfKvGPpFelvr-----------dyCpVVeshtgrt-
d1nfia2 ( 37 ) -k-------cegrsagsIpGerStd--------ttkthPtIkIngyt---
d1t4wa- ( 267 ) Wkldkrhfn---snLsLrIRFVkydkkenveyAIrnprsdVmKcrshter
d1uura2 ( 392 ) tga----------------------------------------------r
d2as5m2 ( 425 ) -e-------teg-srgaVkAptg-------------ghPvVqLhgYmen-
d3brfa2 ( 197 ) sltsdrmidfls-----------------------------nkeky-
d3d06a- ( 149 ) stPpp--------gTrVrAmAIykqsqhmt--------evVrrCphhe--
d5qs9a- ( 41 )
110 120 130 140 150
d1a3qa2 ( 87 ) ---gpAkIeVdLVthsd----ppraHA---HsLvgk--------------
d1bg1a2 ( 382 ) rkFnIlgtntkvMnmeesnngsLsAeFk-hLtLreqrcgnggran----c
d1eaqa- ( 96 ) --dvpdgtlVtVAgnden----ysAelr-nataak---------------
d1mnna- ( 87 ) ---LdlrIiprIDrgFdhideewVGYkrnyFTLVSTFeTa-ncdldtFLk
d1nfia2 ( 68 ) ---gpGtVrISLVTkdp----phrPHP---HeLvgk--------------
d1t4wa- ( 314 ) EqhfpfdSFFYIr----nSe-HefsysaekgstFtLiMyp--gA------
d1uura2 ( 396 ) snFhingpVkAtmicd-----shttpLe-mdsqpiyp------------a
d2as5m2 ( 452 ) ---kplgLqIFIGTAder---ilkpHa--FYqVhritg--------ktVt
d3brfa2 ( 214 ) ---ecvisifhakvaqkSygnekrffc---ppPcIyLigqGWklkkdrva
d3d06a- ( 188 ) ---lAppHLIrVe----gnlrVeYlddntfRhSVVVpyeppvgs------
d5qs9a- ( 41 ) elrVgLeeselWlrFkelt-
bbb bbbb
160 170 180 190 200
d1a3qa2 ( 113 ) ---qCse-------------------lGiCavsVgpk-------------
d1bg1a2 ( 427 ) dasli-----------------vtEELHlItFete---vyhq--------
d1eaqa- ( 126 ) nqvArFndLrFvgrSg------rgksFtLtItVft---n-----------
d1mnna- ( 133 ) ssFdLlvgrlrVq------------yFAIkIkAknddddteinLvqhtak
d1nfia2 ( 94 ) ---dCr--------------------dGFyeaeLcpd-------------
d1t4wa- ( 351 ) -vqAnF-dIiFmCqekcldlddrrktMCLAVFLdd-----en--------
d1uura2 ( 435 ) tlTAhF-pLkFlagt-------rkcSVnLkFgVnI---rdld--------
d2as5m2 ( 486 ) ttSyekivg----------------ntkVLeIpLepkn------------
d3brfa2 ( 258 ) qlyktlt-------------------elvAyIgIgsd----tserqqLdf
d3d06a- ( 228 ) -dctti-hYnYmCnsscm--ggMnspIlTIItLed-----ss--------
d5qs9a- ( 60 ) -NEMiVtknGrrM------------fPvLkVnVsgLdpnamysFlLdFva
bbbbb
210 220 230 240 250
d1a3qa2 ( 128 ) ------dMtaqFnnLgVlhVtkknMmgtMiqkLqrqrlrsrpqglt----
d1bg1a2 ( 449 ) --glkidleth-SlPVVVIsnicqmpnAwASIlWYnmLtnnpknvnFFt-
d1eaqa- ( 156 ) -----ppqvAtYhraIkItv---dGp
d1mnna- ( 177 ) rdkgpqfcPsvcpLVPsplpkHqtIreAsnvnitkmkkydstFylhrdhv
d1nfia2 ( 108 ) ------rcihsfqnLgIqCvkkrdleqAIsqRiqtnNNPf---qvp----
d1t4wa- ( 386 ) --gn--eilhayIkqVrIva---yPrrdwknfceredakq
d1uura2 ( 466 ) --nvtttveSdaSnpFVVItnecqWegSaGvLLkkdAFdg----------
d2as5m2 ( 508 ) ------nMratIDCAGIlklrnadielrk---------------------
d3brfa2 ( 319 ) p-------------------------------------------------
d3d06a- ( 262 ) --gn--llGrnsF-eVrVCa---cpgrdrteen
d5qs9a- ( 97 ) adnhrwkyvngeWvpGg-----------------------------kpep
b bb aa
260 270 280 290 300
d1a3qa2 ( 168 ) ----------------eaeqr----------------eLeqeAkelk--k
d1bg1a2 ( 495 ) --kppiGtwdqVa--evLswqfssttk-------rglsieqlttlAekLl
d1eaqa-
d1mnna- ( 228 ) ny------eeygvdSLLfsYpe--dsIqkVArYerVqFassisvkkp--s
d1nfia2 ( 145 ) ----------------ieeQr----------------g------------
d1t4wa-
d1uura2 ( 504 ) ---qleItwaqFi--ntLqrhfliAtkqdpvrpkrplssydlkyIqthfF
d2as5m2 ( 531 ) ----------------------------------------getdiGr--k
d3brfa2 ( 320 ) -niydyCaaktLyIsdsdkrk--------yFdL----------------n
d3d06a-
d5qs9a- ( 118 ) qapscvyihpdSpnfGahWmka--pVsFskVkLTnklngg---------g
310 320 330 340 350
d1a3qa2 ( 184 ) ---vMdlsiVrLrFsAflr-s---------------lplkpviSqp-IhD
d1bg1a2 ( 534 ) gpg------------------vnysgcqItWakFCkenma---------g
d1eaqa-
d1mnna- ( 268 ) ---qqnkhFSLhVILGAVV-dpgipydelalgsGmFVyLqEmkTppLIIr
d1nfia2 ( 151 ) ---dYdlnAVrLCFqVtVr-dp----------sgrplrlppvlPhp-IfD
d1t4wa-
d1uura2 ( 549 ) gnr------------------s-----iIhqq------------------
d2as5m2 ( 539 ) ------ntrVrLVFRVhIp-es----------sgrivsL-qtaSnp-IeC
d3brfa2 ( 345 ) AqFfYgcgmeiggfvSq-----------------------rIkviskpsk
d3d06a-
d5qs9a- ( 157 ) qImLnslhkYePrIhIvrvgdpqrmitshcFpeT------qFIAVtayqn
360
d1a3qa2 ( 220 ) skspgAs
d1bg1a2 ( 557 ) kgfSFWvwLdniIdlvkky
d1eaqa-
d1mnna- ( 325 ) grspsnyassq
d1nfia2 ( 186 ) nrap
d1t4wa-
d1uura2 ( 558 ) dFdkFwvwfGksMqtlryq
d2as5m2 ( 570 ) sq-rsah
d3brfa2 ( 372 ) kkqsd
d3d06a-
d5qs9a- ( 201 ) eeItalkikyn
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| p52 subunit of NF-kappa B (NFKB), N-terminal domain | Rel/Dorsal transcription factors, DNA-binding domain | Human (Homo sapiens) [TaxId: 9606] | 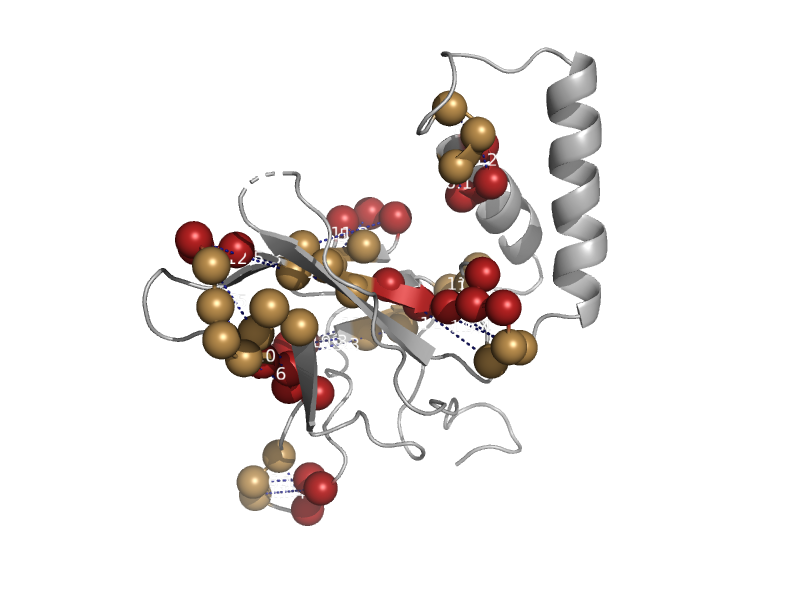 | view | |
| STAT3b | STAT DNA-binding domain | Mouse (Mus musculus) [TaxId: 10090] |  | view | |
| Acute myeloid leukemia 1 protein (AML1), RUNT domain | RUNT domain | Mouse (Mus musculus) [TaxId: 10090] | 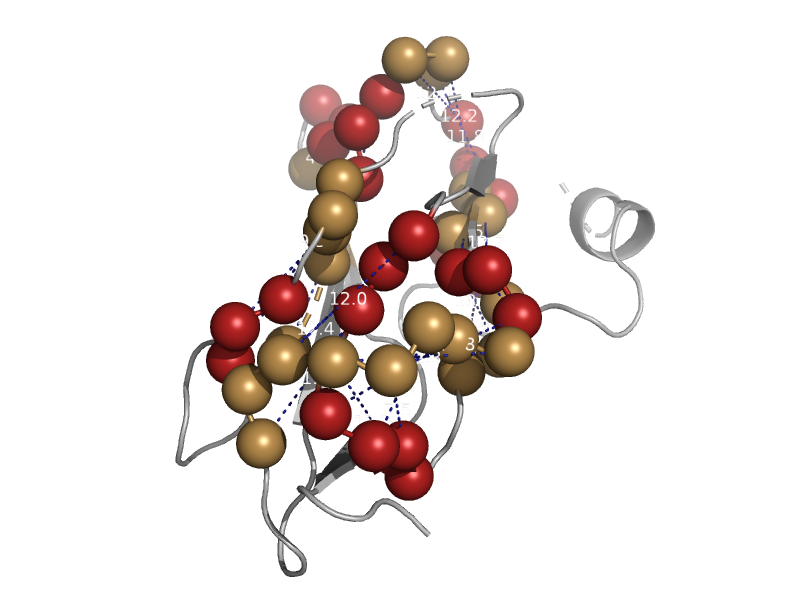 | view | |
| DNA-binding domain from NDT80 | DNA-binding domain from NDT80 | Baker's yeast (Saccharomyces cerevisiae) [TaxId: 4932] | 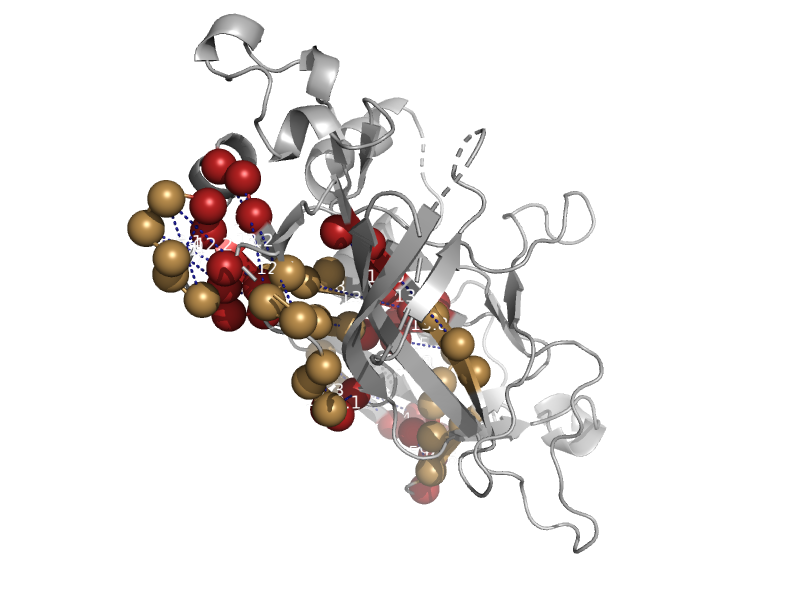 | view | |
| p65 subunit of NF-kappa B (NFKB), N-terminal domain | Rel/Dorsal transcription factors, DNA-binding domain | Human (Homo sapiens) [TaxId: 9606] | 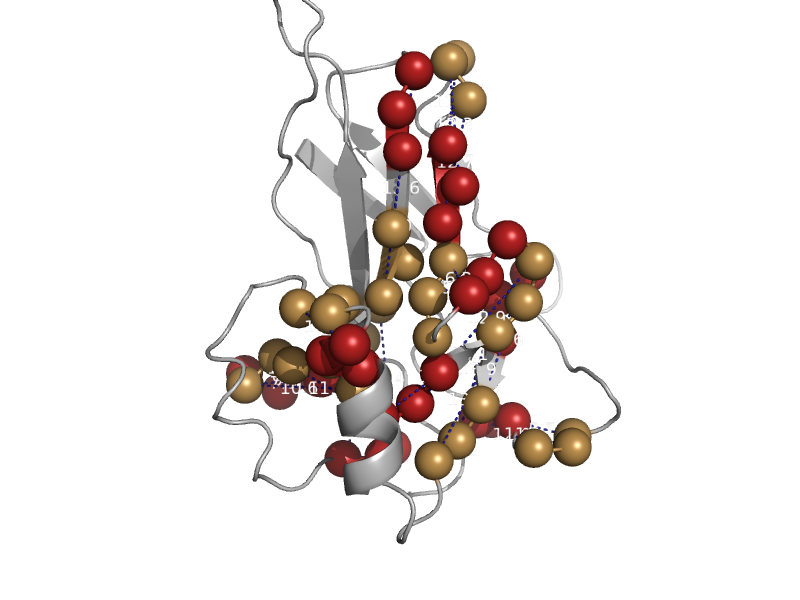 | view | |
| Transcription factor CEP-1 | p53 DNA-binding domain-like | Nematode (Caenorhabditis elegans) [TaxId: 6239] |  | view | |
| STAT homologue | STAT DNA-binding domain | Slime mold (Dictyostelium discoideum) [TaxId: 44689] | 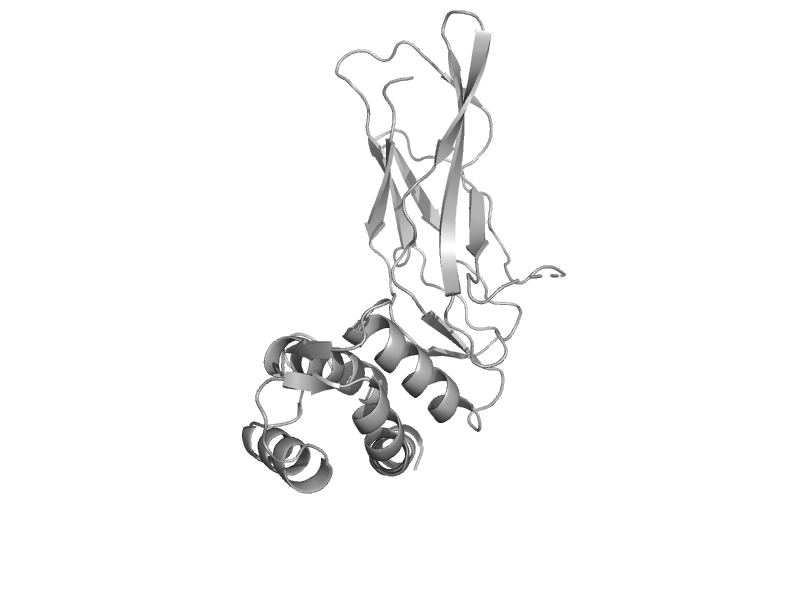 | view | |
| T-cell transcription factor NFAT1 (NFATC), DNA-binding domain | Rel/Dorsal transcription factors, DNA-binding domain | Human (Homo sapiens) [TaxId: 9606] | 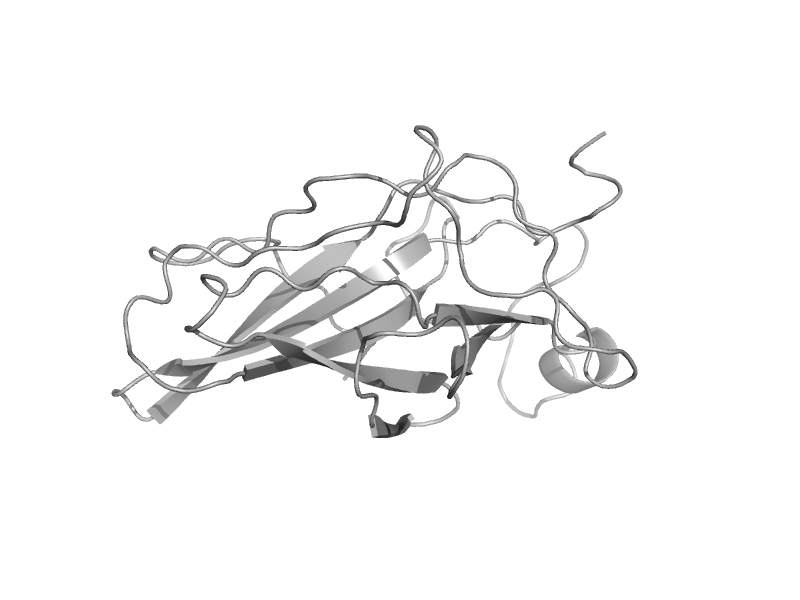 | view | |
| DNA-binding protein LAG-1 (CSL) | DNA-binding protein LAG-1 (CSL) | Nematode (Caenorhabditis elegans) [TaxId: 6239] | 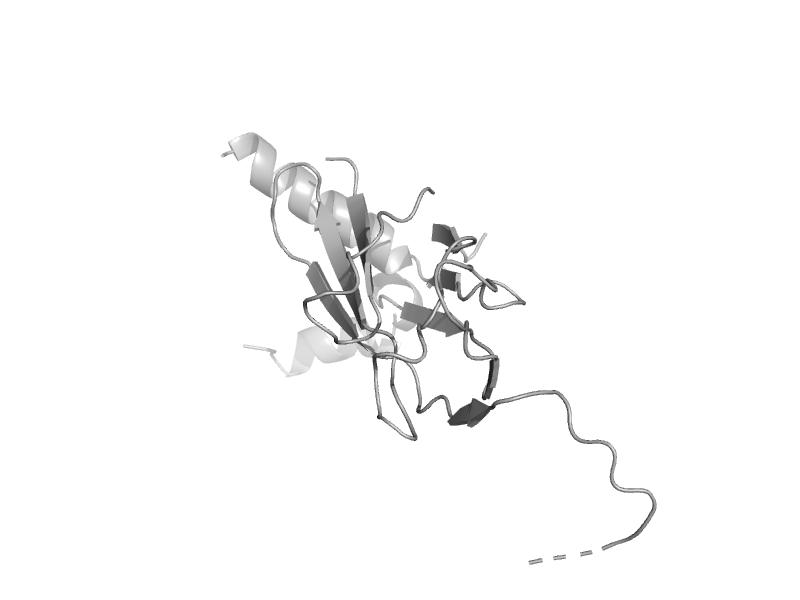 | view | |
| p53 tumor suppressor, DNA-binding domain | p53 DNA-binding domain-like | Human (Homo sapiens) [TaxId: 9606] | 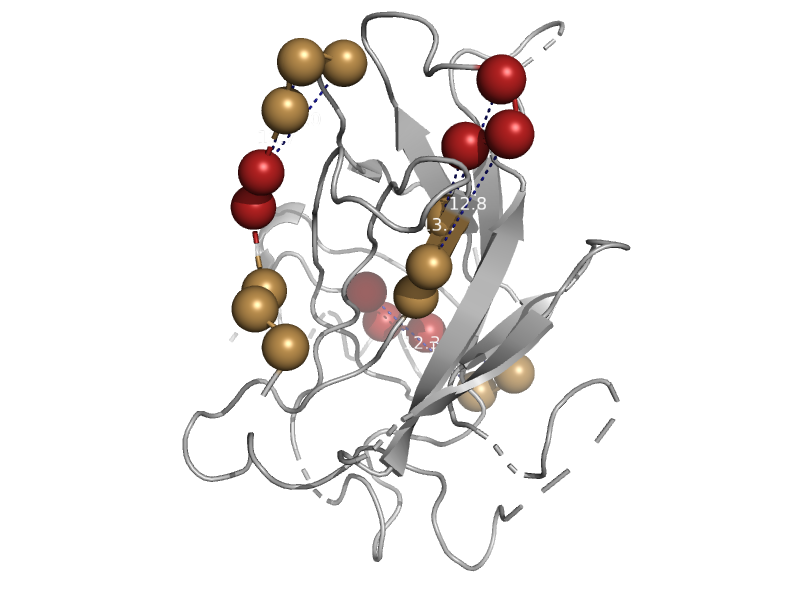 | view | |
| automated matches | automated matches | Human (Homo sapiens) [TaxId: 9606] | 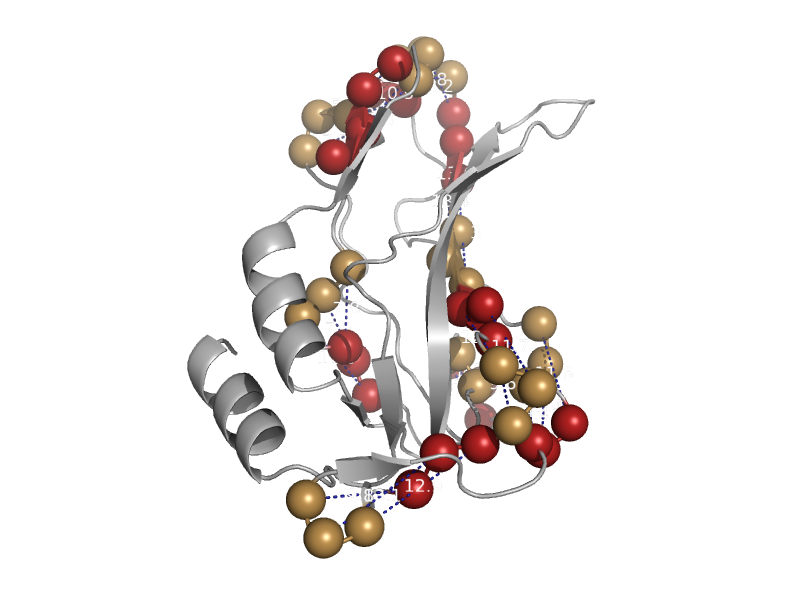 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | 230 | 240 | 250 | 260 | 270 | 280 | 290 | 300 | 310 | 320 | 330 | 340 | 350 | 360 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | P | Y | L | V | I | V | E | Q | P | K | Q | R | G | F | R | F | R | Y | - | - | G | - | - | - | - | - | - | - | C | E | G | P | S | H | G | G | L | P | G | A | S | S | E | K | - | - | - | - | - | - | - | - | G | R | K | T | Y | P | T | V | K | I | C | N | Y | E | - | - | - | - | - | - | G | P | A | K | I | E | V | D | L | V | T | H | S | D | - | - | - | - | P | P | R | A | H | A | - | - | - | H | S | L | V | G | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | C | S | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | G | I | C | A | V | S | V | G | P | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | M | T | A | Q | F | N | N | L | G | V | L | H | V | T | K | K | N | M | M | G | T | M | I | Q | K | L | Q | R | Q | R | L | R | S | R | P | Q | G | L | T | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | A | E | Q | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | L | E | Q | E | A | K | E | L | K | - | - | K | - | - | - | V | M | D | L | S | I | V | R | L | R | F | S | A | F | L | R | - | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | P | L | K | P | V | I | S | Q | P | - | I | H | D | S | K | S | P | G | A | S | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | V | V | E | - | R | Q | P | C | M | P | M | H | P | D | R | P | L | V | I | K | T | G | V | Q | F | T | T | K | V | R | L | L | V | K | F | P | E | L | N | - | - | - | Y | Q | L | K | I | K | V | C | I | D | K | - | - | - | - | - | - | - | D | S | G | D | V | A | - | - | - | - | - | - | - | - | - | A | L | R | G | S | R | K | F | N | I | L | G | T | N | T | K | V | M | N | M | E | E | S | N | N | G | S | L | S | A | E | F | K | - | H | L | T | L | R | E | Q | R | C | G | N | G | G | R | A | N | - | - | - | - | C | D | A | S | L | I | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | V | T | E | E | L | H | L | I | T | F | E | T | E | - | - | - | V | Y | H | Q | - | - | - | - | - | - | - | - | - | - | G | L | K | I | D | L | E | T | H | - | S | L | P | V | V | V | I | S | N | I | C | Q | M | P | N | A | W | A | S | I | L | W | Y | N | M | L | T | N | N | P | K | N | V | N | F | F | T | - | - | - | K | P | P | I | G | T | W | D | Q | V | A | - | - | E | V | L | S | W | Q | F | S | S | T | T | K | - | - | - | - | - | - | - | R | G | L | S | I | E | Q | L | T | T | L | A | E | K | L | L | G | P | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | V | N | Y | S | G | C | Q | I | T | W | A | K | F | C | K | E | N | M | A | - | - | - | - | - | - | - | - | - | G | K | G | F | S | F | W | V | W | L | D | N | I | I | D | L | V | K | K | Y |
| S | V | E | V | L | A | D | H | P | G | E | L | V | R | T | D | S | P | N | F | L | S | S | - | V | L | P | T | - | - | - | - | H | W | R | S | N | K | T | L | P | - | I | A | F | K | V | V | A | L | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | V | P | D | G | T | L | V | T | V | A | G | N | D | E | N | - | - | - | - | Y | S | A | E | L | R | - | N | A | T | A | A | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | Q | V | A | R | F | N | D | L | R | F | V | G | R | S | G | - | - | - | - | - | - | R | G | K | S | F | T | L | T | I | T | V | F | T | - | - | - | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | P | Q | V | A | T | Y | H | R | A | I | K | I | T | V | - | - | - | D | G | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | V | I | L | T | Q | L | N | E | D | G | T | T | S | N | Y | F | D | K | R | K | L | K | I | A | P | R | S | T | L | Q | F | K | V | G | P | P | F | E | L | V | R | - | - | - | - | - | - | - | - | - | - | - | D | Y | C | P | V | V | E | S | H | T | G | R | T | - | - | - | - | L | D | L | R | I | I | P | R | I | D | R | G | F | D | H | I | D | E | E | W | V | G | Y | K | R | N | Y | F | T | L | V | S | T | F | E | T | A | - | N | C | D | L | D | T | F | L | K | S | S | F | D | L | L | V | G | R | L | R | V | Q | - | - | - | - | - | - | - | - | - | - | - | - | Y | F | A | I | K | I | K | A | K | N | D | D | D | D | T | E | I | N | L | V | Q | H | T | A | K | R | D | K | G | P | Q | F | C | P | S | V | C | P | L | V | P | S | P | L | P | K | H | Q | T | I | R | E | A | S | N | V | N | I | T | K | M | K | K | Y | D | S | T | F | Y | L | H | R | D | H | V | N | Y | - | - | - | - | - | - | E | E | Y | G | V | D | S | L | L | F | S | Y | P | E | - | - | D | S | I | Q | K | V | A | R | Y | E | R | V | Q | F | A | S | S | I | S | V | K | K | P | - | - | S | - | - | - | Q | Q | N | K | H | F | S | L | H | V | I | L | G | A | V | V | - | D | P | G | I | P | Y | D | E | L | A | L | G | S | G | M | F | V | Y | L | Q | E | M | K | T | P | P | L | I | I | R | G | R | S | P | S | N | Y | A | S | S | Q | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Y | V | E | I | I | E | Q | P | K | Q | R | G | M | R | F | R | Y | - | - | K | - | - | - | - | - | - | - | C | E | G | R | S | A | G | S | I | P | G | E | R | S | T | D | - | - | - | - | - | - | - | - | T | T | K | T | H | P | T | I | K | I | N | G | Y | T | - | - | - | - | - | - | G | P | G | T | V | R | I | S | L | V | T | K | D | P | - | - | - | - | P | H | R | P | H | P | - | - | - | H | E | L | V | G | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | C | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | G | F | Y | E | A | E | L | C | P | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | C | I | H | S | F | Q | N | L | G | I | Q | C | V | K | K | R | D | L | E | Q | A | I | S | Q | R | I | Q | T | N | N | N | P | F | - | - | - | Q | V | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | I | E | E | Q | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | Y | D | L | N | A | V | R | L | C | F | Q | V | T | V | R | - | D | P | - | - | - | - | - | - | - | - | - | - | S | G | R | P | L | R | L | P | P | V | L | P | H | P | - | I | F | D | N | R | A | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | E | K | W | M | E | I | D | V | L | K | Q | K | V | A | - | K | S | S | D | M | A | F | A | I | S | S | E | H | E | K | Y | L | W | T | K | M | G | C | L | V | P | I | Q | V | K | W | K | L | D | K | R | H | F | N | - | - | - | S | N | L | S | L | R | I | R | F | V | K | Y | D | K | K | E | N | V | E | Y | A | I | R | N | P | R | S | D | V | M | K | C | R | S | H | T | E | R | E | Q | H | F | P | F | D | S | F | F | Y | I | R | - | - | - | - | N | S | E | - | H | E | F | S | Y | S | A | E | K | G | S | T | F | T | L | I | M | Y | P | - | - | G | A | - | - | - | - | - | - | - | V | Q | A | N | F | - | D | I | I | F | M | C | Q | E | K | C | L | D | L | D | D | R | R | K | T | M | C | L | A | V | F | L | D | D | - | - | - | - | - | E | N | - | - | - | - | - | - | - | - | - | - | G | N | - | - | E | I | L | H | A | Y | I | K | Q | V | R | I | V | A | - | - | - | Y | P | R | R | D | W | K | N | F | C | E | R | E | D | A | K | Q | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | N | V | A | L | V | L | K | - | S | Q | P | F | P | - | - | V | V | I | S | K | G | K | Q | L | G | E | N | Q | L | V | V | L | V | L | T | G | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | S | N | F | H | I | N | G | P | V | K | A | T | M | I | C | D | - | - | - | - | - | S | H | T | T | P | L | E | - | M | D | S | Q | P | I | Y | P | - | - | - | - | - | - | - | - | - | - | - | - | A | T | L | T | A | H | F | - | P | L | K | F | L | A | G | T | - | - | - | - | - | - | - | R | K | C | S | V | N | L | K | F | G | V | N | I | - | - | - | R | D | L | D | - | - | - | - | - | - | - | - | - | - | N | V | T | T | T | V | E | S | D | A | S | N | P | F | V | V | I | T | N | E | C | Q | W | E | G | S | A | G | V | L | L | K | K | D | A | F | D | G | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | L | E | I | T | W | A | Q | F | I | - | - | N | T | L | Q | R | H | F | L | I | A | T | K | Q | D | P | V | R | P | K | R | P | L | S | S | Y | D | L | K | Y | I | Q | T | H | F | F | G | N | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | - | - | - | - | - | I | I | H | Q | Q | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | F | D | K | F | W | V | W | F | G | K | S | M | Q | T | L | R | Y | Q |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | L | E | W | P | L | S | S | Q | S | G | S | Y | E | L | R | I | E | V | Q | P | K | P | - | H | H | R | A | H | Y | - | - | E | - | - | - | - | - | - | - | T | E | G | - | S | R | G | A | V | K | A | P | T | G | - | - | - | - | - | - | - | - | - | - | - | - | - | G | H | P | V | V | Q | L | H | G | Y | M | E | N | - | - | - | - | K | P | L | G | L | Q | I | F | I | G | T | A | D | E | R | - | - | - | I | L | K | P | H | A | - | - | F | Y | Q | V | H | R | I | T | G | - | - | - | - | - | - | - | - | K | T | V | T | T | T | S | Y | E | K | I | V | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | T | K | V | L | E | I | P | L | E | P | K | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | M | R | A | T | I | D | C | A | G | I | L | K | L | R | N | A | D | I | E | L | R | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | E | T | D | I | G | R | - | - | K | - | - | - | - | - | - | N | T | R | V | R | L | V | F | R | V | H | I | P | - | E | S | - | - | - | - | - | - | - | - | - | - | S | G | R | I | V | S | L | - | Q | T | A | S | N | P | - | I | E | C | S | Q | - | R | S | A | H | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | L | T | S | D | R | M | I | D | F | L | S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | K | E | K | Y | - | - | - | - | E | C | V | I | S | I | F | H | A | K | V | A | Q | K | S | Y | G | N | E | K | R | F | F | C | - | - | - | P | P | P | C | I | Y | L | I | G | Q | G | W | K | L | K | K | D | R | V | A | Q | L | Y | K | T | L | T | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | L | V | A | Y | I | G | I | G | S | D | - | - | - | - | T | S | E | R | Q | Q | L | D | F | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | I | Y | D | Y | C | A | A | K | T | L | Y | I | S | D | S | D | K | R | K | - | - | - | - | - | - | - | - | Y | F | D | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | A | Q | F | F | Y | G | C | G | M | E | I | G | G | F | V | S | Q | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | I | K | V | I | S | K | P | S | K | K | K | Q | S | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | V | P | S | Q | T | Y | G | S | Y | G | F | R | L | G | F | L | T | C | T | Y | S | P | A | L | N | K | M | F | C | Q | L | A | T | C | P | V | Q | L | W | V | D | S | T | P | P | P | - | - | - | - | - | - | - | - | G | T | R | V | R | A | M | A | I | Y | K | Q | S | Q | H | M | T | - | - | - | - | - | - | - | - | E | V | V | R | R | C | P | H | H | E | - | - | - | - | - | L | A | P | P | H | L | I | R | V | E | - | - | - | - | G | N | L | R | V | E | Y | L | D | D | N | T | F | R | H | S | V | V | V | P | Y | E | P | P | V | G | S | - | - | - | - | - | - | - | D | C | T | T | I | - | H | Y | N | Y | M | C | N | S | S | C | M | - | - | G | G | M | N | S | P | I | L | T | I | I | T | L | E | D | - | - | - | - | - | S | S | - | - | - | - | - | - | - | - | - | - | G | N | - | - | L | L | G | R | N | S | F | - | E | V | R | V | C | A | - | - | - | C | P | G | R | D | R | T | E | E | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | L | R | V | G | L | E | E | S | E | L | W | L | R | F | K | E | L | T | - | - | N | E | M | I | V | T | K | N | G | R | R | M | - | - | - | - | - | - | - | - | - | - | - | - | F | P | V | L | K | V | N | V | S | G | L | D | P | N | A | M | Y | S | F | L | L | D | F | V | A | A | D | N | H | R | W | K | Y | V | N | G | E | W | V | P | G | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | P | E | P | Q | A | P | S | C | V | Y | I | H | P | D | S | P | N | F | G | A | H | W | M | K | A | - | - | P | V | S | F | S | K | V | K | L | T | N | K | L | N | G | G | - | - | - | - | - | - | - | - | - | G | Q | I | M | L | N | S | L | H | K | Y | E | P | R | I | H | I | V | R | V | G | D | P | Q | R | M | I | T | S | H | C | F | P | E | T | - | - | - | - | - | - | Q | F | I | A | V | T | A | Y | Q | N | E | E | I | T | A | L | K | I | K | Y | N | - | - | - | - | - | - | - | - |
