solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1ecma- ( 5 )
d1ybza1 ( 2 )
d2d8da1 ( 3 )
d2fp1a- ( 35 )
d2gtvx1 ( 1 )
d3reta- ( 1 ) mktpedCtgladireaidridldivqalgrrmdyvkaasrfea-------
d4ppua- ( 79 ) rvdese----sltlegirnslirqEdsIIfgLleRAkycyNadTydptaf
d5csma- ( 1 ) Mdftkpe--tVlnlqnIrdelvrmEdsIIfkFieRshfAtCpSVyeanhp
d5ts9a- ( 18 )
d6cnza- ( 37 )
60 70 80 90 100
d1ecma- ( 5 )
d1ybza1 ( 2 )
d2d8da1 ( 3 )
d2fp1a- ( 35 )
d2gtvx1 ( 1 )
d3reta- ( 44 ) -------seaaIpapervaamlperarwAeen------------------
d4ppua- ( 125 ) --dmdgfngSlVe--ymVkgteklhAkvgrfkspDehpFfpddlpepmlp
d5csma- ( 49 ) gleipnfkgsfLd--waLsnlEiahSrirrfespdeTpFfpdkiqkSflp
d5ts9a- ( 18 )
d6cnza- ( 37 )
110 120 130 140 150
d1ecma- ( 5 ) npllalrekisal
d1ybza1 ( 2 ) ttlllreidi
d2d8da1 ( 3 ) eriqalrkevdrv
d2fp1a- ( 35 ) g-tSq
d2gtvx1 ( 1 ) m
d3reta- ( 69 ) --------------------------------------------------
d4ppua- ( 171 ) plqypkvLhfaAdsiniNkkIwnMYfrdLVprLVk-kgddg-nygsTAvc
d5csma- ( 97 ) sinypqILApyapeVnyNdkIkkvYiekIIplISkrdgddknnfgsVAtr
d5ts9a- ( 18 ) gaqda
d6cnza- ( 37 ) ta
160 170 180 190 200
d1ecma- ( 18 ) dekllallaerrelavevGkakllsh-----------------rpVrdid
d1ybza1 ( 15 ) dnqiisllkkrleiAqaiGikellp-------------------------
d2d8da1 ( 16 ) nreilrllsergrlvqeigrlqtelg-----------------lphydpk
d2fp1a- ( 39 ) LaeLVdAAAeRLevAdpvAAfkwraq-----------------lpIedsg
d2gtvx1 ( 2 ) ieklaeirkkideiDnkilkarwp-------------------waeklia
d3reta- ( 69 ) --------------------------------------------------
d4ppua- ( 219 ) DaiCLqcLSkrIHYGKFvAEakfqaspeayesaIkaqdkdaLmdmLtfpt
d5csma- ( 147 ) DieCLqsLSrrIHFGKFvAEAkFqsdiplYtklIkskdvegImknItnsa
d5ts9a- ( 24 ) FVpLVrsMAdrLntAdqvALskwdtg-----------------qpVyd--
d6cnza- ( 39 ) LtnLValASqrLalAepvAhwkwinr-----------------kpisdpp
aaaaaaaaaaaa aaaaaaaaaa a
210 220 230 240 250
d1ecma- ( 51 ) rerdllerlitlgkah-------hldahyItrlfqliiedsvltqqallq
d1ybza1 ( 45 ) dreeVlrragef------------------reifekilevSdvq
d2d8da1 ( 49 ) reeemLayltaenp-g-------pfpdetirklfkeiFkasl
d2fp1a- ( 72 ) rveqqLakLgedArsq-------hIdpdYVtrVFddQirAteaIEysrfs
d2gtvx1 ( 33 ) eRnslAkdvAeiKnql-------gip---in--dperekyiydrirklCk
d3reta- ( 69 ) -----------------------gldapfveglfaqiIhwYiaeqikywr
d4ppua- ( 269 ) vedaikkrVemkTrtygqevkvykisPilVGdLYgdwImplTkeVqVeYL
d5csma- ( 197 ) veekilerLtkkAevygvdpt--rIspeyLvkIYkeIVipItkeVEveYL
d5ts9a- ( 55 ) geaqvianAatmA-sy-------gLt-adAinIFsDQveAnkeVqyalLn
d6cnza- ( 72 ) reaalltdVe-rAtan-------gVdpaYArtFFddQiaAskqLQnalfa
aaaaaaaaaaaa aaaaaaaaaaaaaaaaaaaaaaaa
260 270 280 290 300
d1ecma- ( 94 ) qh
d1ybza1
d2d8da1
d2fp1a- ( 115 ) dwklnpasAppeppdls-asrsaidsLnnrMLsqIwshwslLsapsÇaaq
d2gtvx1 ( 71 ) ehn----------vden-igikiFqrliehnkalqkqyleet
d3reta- ( 96 ) qt
d4ppua- ( 336 ) lrrld
d5csma- ( 251 ) lrrlee
d5ts9a- ( 100 ) nwrrqgdApatprqslagvIrpild-lqasIMqnLqsVaplrsiadÇhal
d6cnza- ( 115 ) twrath-gpegpApdlatsTrpqldrLTqsLIaaLarVaplrdapdÇpsr
a
310 320 330
d1ecma-
d1ybza1
d2d8da1
d2fp1a- ( 164 ) ldrAkrdivrsrhLdslyqrALttATqsYçqalppa
d2gtvx1
d3reta-
d4ppua-
d5csma-
d5ts9a- ( 150 ) VasaVgqVa-qasldvlhraALdrAvarIÇv
d6cnza- ( 164 ) larsianwktltrydsaqkdALgtALshVÇaa
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Chorismate mutase domain of P-protein | Dimeric chorismate mutase | Escherichia coli [TaxId: 562] |  | view | |
| mono-domain chorismate mutase | Dimeric chorismate mutase | Pyrococcus furiosus [TaxId: 2261] |  | view | |
| Chorismate mutase domain of P-protein | Dimeric chorismate mutase | Thermus thermophilus [TaxId: 274] |  | view | |
| Secreted chorismate mutase | Secreted chorismate mutase-like | Mycobacterium tuberculosis [TaxId: 1773] |  | view | |
| Chorismate mutase-like protein MJ0246 | monomeric chorismate mutase | Methanococcus jannaschii [TaxId: 2190] | 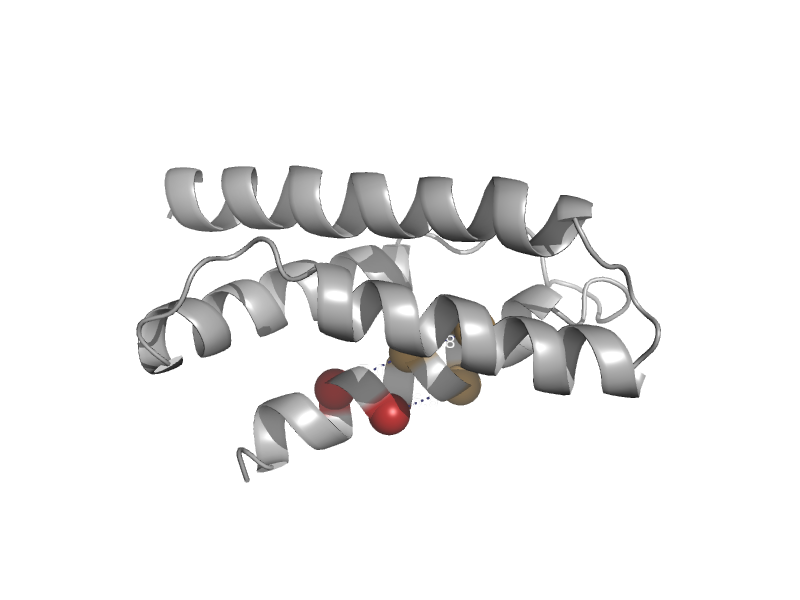 | view | |
| automated matches | Dimeric chorismate mutase | Pseudomonas aeruginosa [TaxId: 287] | 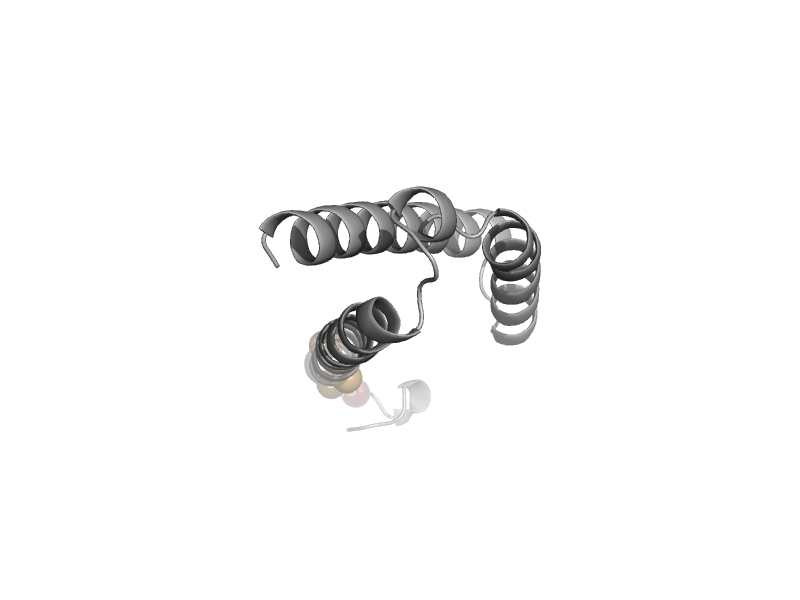 | view | |
| automated matches | automated matches | Thale cress (Arabidopsis thaliana) [TaxId: 3702] | 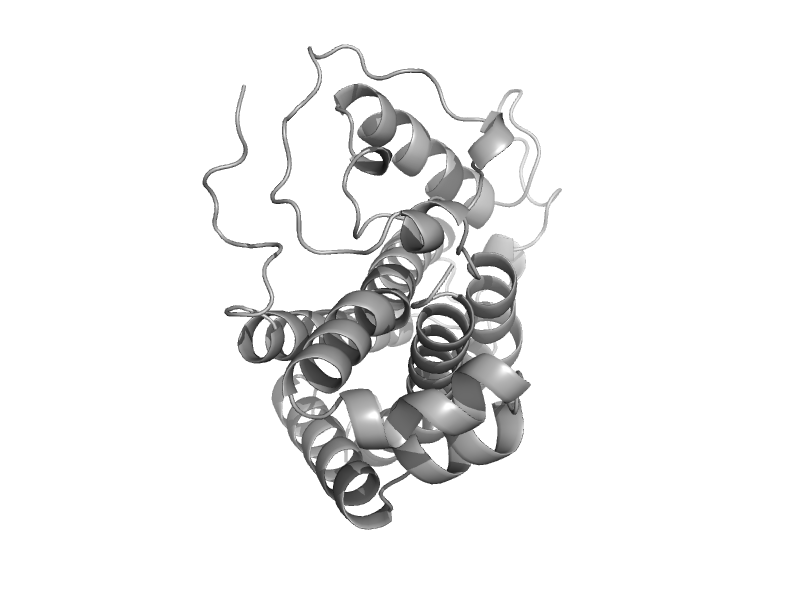 | view | |
| Allosteric chorismate mutase | Allosteric chorismate mutase | Baker's yeast (Saccharomyces cerevisiae) [TaxId: 4932] | 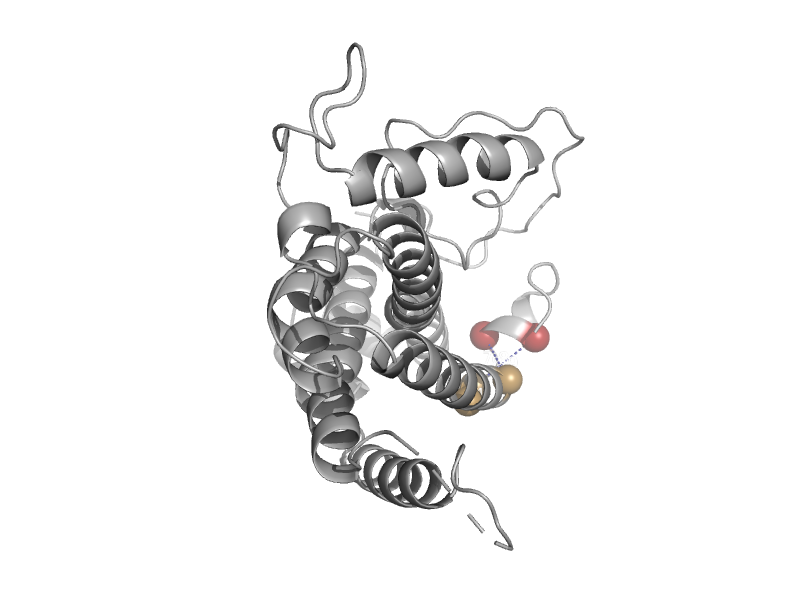 | view | |
| automated matches | automated matches | Paraburkholderia phymatum [TaxId: 391038] |  | view | |
| automated matches | automated matches | Burkholderia thailandensis [TaxId: 271848] |  | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | 230 | 240 | 250 | 260 | 270 | 280 | 290 | 300 | 310 | 320 | 330 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | P | L | L | A | L | R | E | K | I | S | A | L | D | E | K | L | L | A | L | L | A | E | R | R | E | L | A | V | E | V | G | K | A | K | L | L | S | H | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | P | V | R | D | I | D | R | E | R | D | L | L | E | R | L | I | T | L | G | K | A | H | - | - | - | - | - | - | - | H | L | D | A | H | Y | I | T | R | L | F | Q | L | I | I | E | D | S | V | L | T | Q | Q | A | L | L | Q | Q | H | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | T | T | L | L | L | R | E | I | D | I | D | N | Q | I | I | S | L | L | K | K | R | L | E | I | A | Q | A | I | G | I | K | E | L | L | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | R | E | E | V | L | R | R | A | G | E | F | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | E | I | F | E | K | I | L | E | V | S | D | V | Q | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | R | I | Q | A | L | R | K | E | V | D | R | V | N | R | E | I | L | R | L | L | S | E | R | G | R | L | V | Q | E | I | G | R | L | Q | T | E | L | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | P | H | Y | D | P | K | R | E | E | E | M | L | A | Y | L | T | A | E | N | P | - | G | - | - | - | - | - | - | - | P | F | P | D | E | T | I | R | K | L | F | K | E | I | F | K | A | S | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | - | T | S | Q | L | A | E | L | V | D | A | A | A | E | R | L | E | V | A | D | P | V | A | A | F | K | W | R | A | Q | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | P | I | E | D | S | G | R | V | E | Q | Q | L | A | K | L | G | E | D | A | R | S | Q | - | - | - | - | - | - | - | H | I | D | P | D | Y | V | T | R | V | F | D | D | Q | I | R | A | T | E | A | I | E | Y | S | R | F | S | D | W | K | L | N | P | A | S | A | P | P | E | P | P | D | L | S | - | A | S | R | S | A | I | D | S | L | N | N | R | M | L | S | Q | I | W | S | H | W | S | L | L | S | A | P | S | C | A | A | Q | L | D | R | A | K | R | D | I | V | R | S | R | H | L | D | S | L | Y | Q | R | A | L | T | T | A | T | Q | S | Y | C | Q | A | L | P | P | A |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | M | I | E | K | L | A | E | I | R | K | K | I | D | E | I | D | N | K | I | L | K | A | R | W | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | W | A | E | K | L | I | A | E | R | N | S | L | A | K | D | V | A | E | I | K | N | Q | L | - | - | - | - | - | - | - | G | I | P | - | - | - | I | N | - | - | D | P | E | R | E | K | Y | I | Y | D | R | I | R | K | L | C | K | E | H | N | - | - | - | - | - | - | - | - | - | - | V | D | E | N | - | I | G | I | K | I | F | Q | R | L | I | E | H | N | K | A | L | Q | K | Q | Y | L | E | E | T | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| M | K | T | P | E | D | C | T | G | L | A | D | I | R | E | A | I | D | R | I | D | L | D | I | V | Q | A | L | G | R | R | M | D | Y | V | K | A | A | S | R | F | E | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | S | E | A | A | I | P | A | P | E | R | V | A | A | M | L | P | E | R | A | R | W | A | E | E | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | L | D | A | P | F | V | E | G | L | F | A | Q | I | I | H | W | Y | I | A | E | Q | I | K | Y | W | R | Q | T | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| R | V | D | E | S | E | - | - | - | - | S | L | T | L | E | G | I | R | N | S | L | I | R | Q | E | D | S | I | I | F | G | L | L | E | R | A | K | Y | C | Y | N | A | D | T | Y | D | P | T | A | F | - | - | D | M | D | G | F | N | G | S | L | V | E | - | - | Y | M | V | K | G | T | E | K | L | H | A | K | V | G | R | F | K | S | P | D | E | H | P | F | F | P | D | D | L | P | E | P | M | L | P | P | L | Q | Y | P | K | V | L | H | F | A | A | D | S | I | N | I | N | K | K | I | W | N | M | Y | F | R | D | L | V | P | R | L | V | K | - | K | G | D | D | G | - | N | Y | G | S | T | A | V | C | D | A | I | C | L | Q | C | L | S | K | R | I | H | Y | G | K | F | V | A | E | A | K | F | Q | A | S | P | E | A | Y | E | S | A | I | K | A | Q | D | K | D | A | L | M | D | M | L | T | F | P | T | V | E | D | A | I | K | K | R | V | E | M | K | T | R | T | Y | G | Q | E | V | K | V | Y | K | I | S | P | I | L | V | G | D | L | Y | G | D | W | I | M | P | L | T | K | E | V | Q | V | E | Y | L | L | R | R | L | D | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| M | D | F | T | K | P | E | - | - | T | V | L | N | L | Q | N | I | R | D | E | L | V | R | M | E | D | S | I | I | F | K | F | I | E | R | S | H | F | A | T | C | P | S | V | Y | E | A | N | H | P | G | L | E | I | P | N | F | K | G | S | F | L | D | - | - | W | A | L | S | N | L | E | I | A | H | S | R | I | R | R | F | E | S | P | D | E | T | P | F | F | P | D | K | I | Q | K | S | F | L | P | S | I | N | Y | P | Q | I | L | A | P | Y | A | P | E | V | N | Y | N | D | K | I | K | K | V | Y | I | E | K | I | I | P | L | I | S | K | R | D | G | D | D | K | N | N | F | G | S | V | A | T | R | D | I | E | C | L | Q | S | L | S | R | R | I | H | F | G | K | F | V | A | E | A | K | F | Q | S | D | I | P | L | Y | T | K | L | I | K | S | K | D | V | E | G | I | M | K | N | I | T | N | S | A | V | E | E | K | I | L | E | R | L | T | K | K | A | E | V | Y | G | V | D | P | T | - | - | R | I | S | P | E | Y | L | V | K | I | Y | K | E | I | V | I | P | I | T | K | E | V | E | V | E | Y | L | L | R | R | L | E | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | A | Q | D | A | F | V | P | L | V | R | S | M | A | D | R | L | N | T | A | D | Q | V | A | L | S | K | W | D | T | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | P | V | Y | D | - | - | G | E | A | Q | V | I | A | N | A | A | T | M | A | - | S | Y | - | - | - | - | - | - | - | G | L | T | - | A | D | A | I | N | I | F | S | D | Q | V | E | A | N | K | E | V | Q | Y | A | L | L | N | N | W | R | R | Q | G | D | A | P | A | T | P | R | Q | S | L | A | G | V | I | R | P | I | L | D | - | L | Q | A | S | I | M | Q | N | L | Q | S | V | A | P | L | R | S | I | A | D | C | H | A | L | V | A | S | A | V | G | Q | V | A | - | Q | A | S | L | D | V | L | H | R | A | A | L | D | R | A | V | A | R | I | C | V | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | T | A | L | T | N | L | V | A | L | A | S | Q | R | L | A | L | A | E | P | V | A | H | W | K | W | I | N | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | P | I | S | D | P | P | R | E | A | A | L | L | T | D | V | E | - | R | A | T | A | N | - | - | - | - | - | - | - | G | V | D | P | A | Y | A | R | T | F | F | D | D | Q | I | A | A | S | K | Q | L | Q | N | A | L | F | A | T | W | R | A | T | H | - | G | P | E | G | P | A | P | D | L | A | T | S | T | R | P | Q | L | D | R | L | T | Q | S | L | I | A | A | L | A | R | V | A | P | L | R | D | A | P | D | C | P | S | R | L | A | R | S | I | A | N | W | K | T | L | T | R | Y | D | S | A | Q | K | D | A | L | G | T | A | L | S | H | V | C | A | A | - | - | - | - |
