solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1cmwa6 ( 174 ) lrpdqwadyraLtgdesdnLpgvkgigektA------------rkLle
d1ul1x1 ( 218 ) lnqeqfVDlcILlgsdyC--esIrgIg----------------p-kAv
d3h7ia2 ( 181 ) ksgsaeidcMtkILkGdkkdnvASVkvrsdFwftrvegertpsMktsiVe
d3orya2 ( 225 ) itlenlidigILlGtdyNp-dGfegIg----------------pkkAl
d5hmla2 ( 186 ) vd-dveqfiSlkAImGdlgdnirgVe----------------gIgakrGy
aaaaaaaaaaa a
60 70 80 90 100
d1cmwa6 ( 210 ) ewgsleALlknldrlkpAirekil----ahm------ddlklSwdlakvr
d1ul1x1 ( 248 ) dlIq-h---------ksIeeIvrrldpnkypvpenw-lh-eAhqlfl--p
d3h7ia2 ( 231 ) aIAn--------dreqAkvlLt-----------eseynrYkeNLvlidf-
d3orya2 ( 256 ) qlVkay---------ggIekIp----kpilkspiev-dviaikkyflq-p
d5hmla2 ( 219 ) nIIrefg-----nVldIidqlpLpgkqkyIqnLnaseelLfrNlilvdlp
aaa aaaaa
110 120 130 140 150
d1cmwa6 ( 250 ) tdlplevdfakrrepdrerlraflerle--fGsllhefgl----------
d1ul1x1 ( 287 ) evldpesvelkwsepneeelikfmCge--fse-rirsgv-rlsksrqgst
d3h7ia2 ( 261 ) dy-------------ipdniasnivnyynsyklpprgkiysyFvkaglsk
d3orya2 ( 291 ) qvtd--nyriewhtpdpdavkrilvdehdfsidrvstaleryvkafkeni
d5hmla2 ( 264 ) ty-------------cvdaiaavgqdvldk-------ftkdileiae
aaaaaaa a aaaa
160
d1cmwa6 ( 288 ) -----------------le
d1ul1x1 ( 337 ) qglddffkvtgslssakrk
d3h7ia2 ( 298 ) ltnsinef
d3orya2 ( 339 ) r
d5hmla2
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| 5' to 3' exonuclease domain of DNA polymerase Taq | 5' to 3' exonuclease, C-terminal subdomain | Thermus aquaticus [TaxId: 271] | 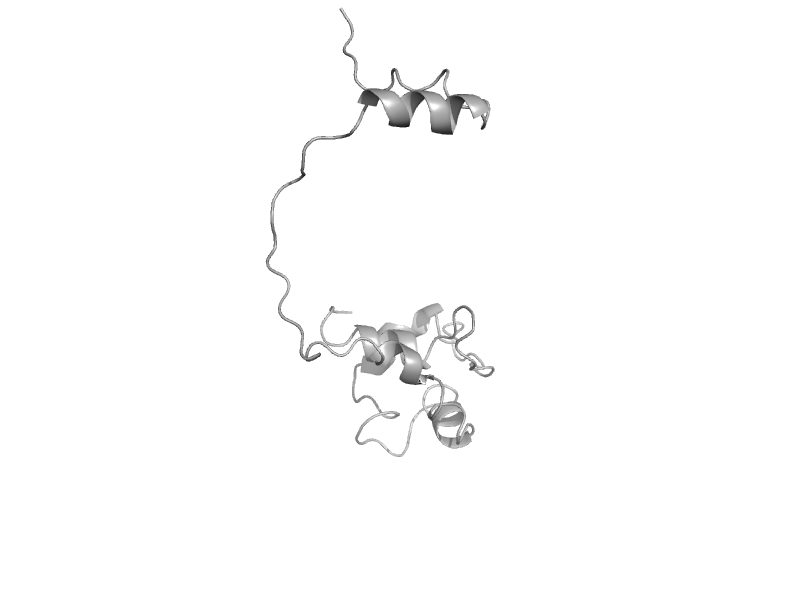 | view | |
| Flap endonuclease-1 (Fen-1 nuclease) | 5' to 3' exonuclease, C-terminal subdomain | Human (Homo sapiens) [TaxId: 9606] | 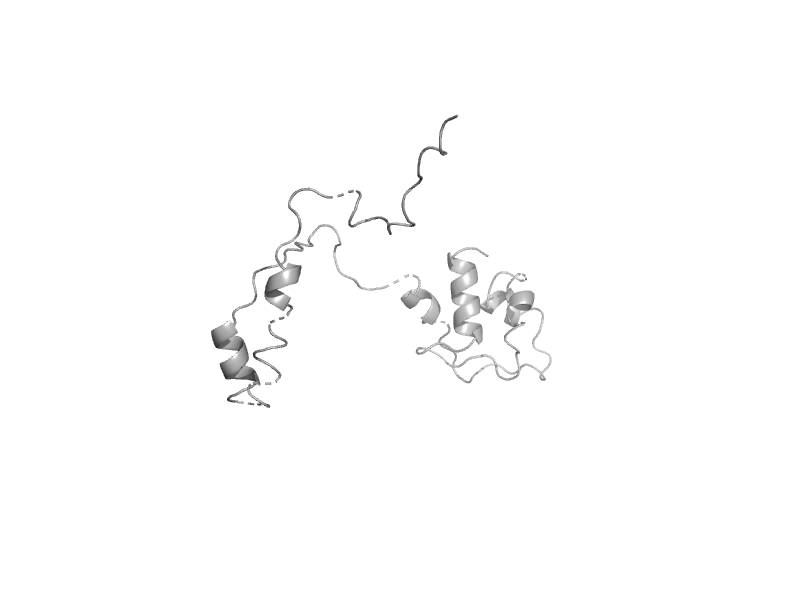 | view | |
| T4 RNase H | 5' to 3' exonuclease, C-terminal subdomain | Bacteriophage T4 [TaxId: 10665] | 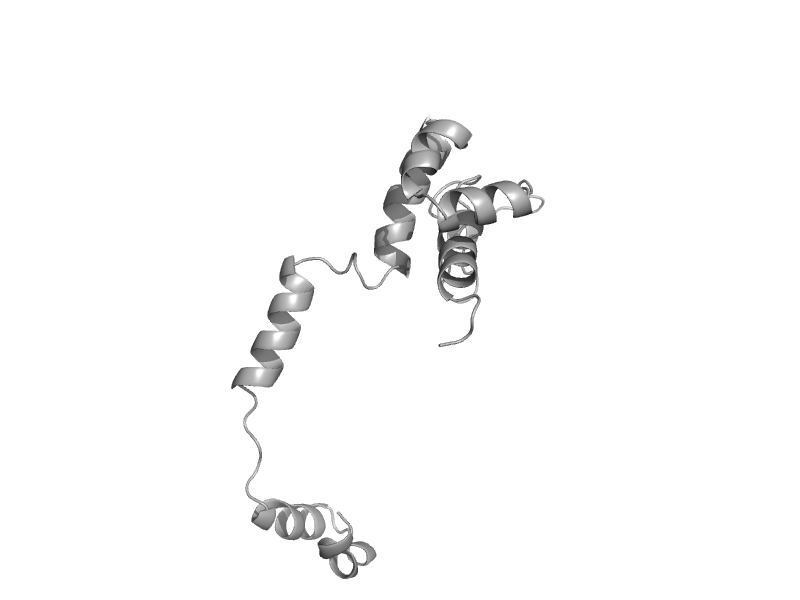 | view | |
| automated matches | automated matches | Desulfurococcus amylolyticus [TaxId: 94694] | 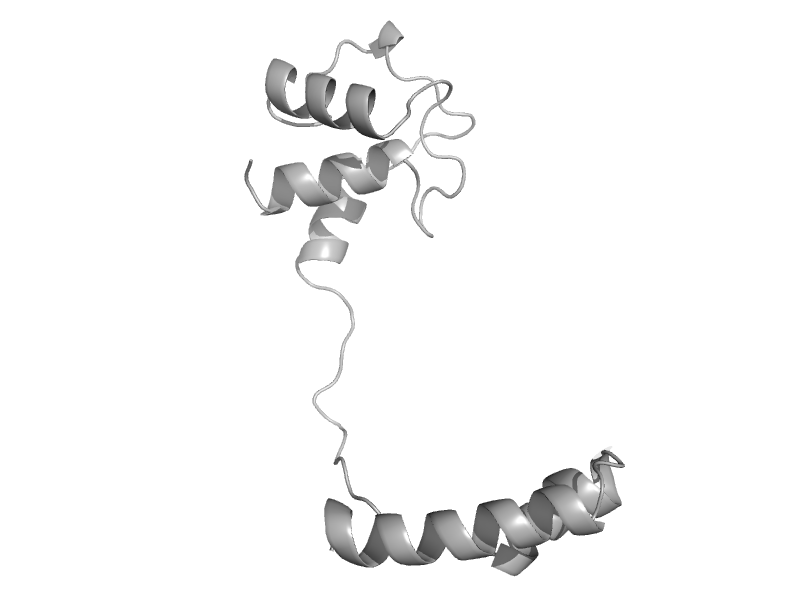 | view | |
| T5 5'-exonuclease | 5' to 3' exonuclease, C-terminal subdomain | Bacteriophage T5 [TaxId: 10726] |  | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | L | R | P | D | Q | W | A | D | Y | R | A | L | T | G | D | E | S | D | N | L | P | G | V | K | G | I | G | E | K | T | A | - | - | - | - | - | - | - | - | - | - | - | - | R | K | L | L | E | E | W | G | S | L | E | A | L | L | K | N | L | D | R | L | K | P | A | I | R | E | K | I | L | - | - | - | - | A | H | M | - | - | - | - | - | - | D | D | L | K | L | S | W | D | L | A | K | V | R | T | D | L | P | L | E | V | D | F | A | K | R | R | E | P | D | R | E | R | L | R | A | F | L | E | R | L | E | - | - | F | G | S | L | L | H | E | F | G | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | E |
| - | - | L | N | Q | E | Q | F | V | D | L | C | I | L | L | G | S | D | Y | C | - | - | E | S | I | R | G | I | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | - | K | A | V | D | L | I | Q | - | H | - | - | - | - | - | - | - | - | - | K | S | I | E | E | I | V | R | R | L | D | P | N | K | Y | P | V | P | E | N | W | - | L | H | - | E | A | H | Q | L | F | L | - | - | P | E | V | L | D | P | E | S | V | E | L | K | W | S | E | P | N | E | E | E | L | I | K | F | M | C | G | E | - | - | F | S | E | - | R | I | R | S | G | V | - | R | L | S | K | S | R | Q | G | S | T | Q | G | L | D | D | F | F | K | V | T | G | S | L | S | S | A | K | R | K |
| K | S | G | S | A | E | I | D | C | M | T | K | I | L | K | G | D | K | K | D | N | V | A | S | V | K | V | R | S | D | F | W | F | T | R | V | E | G | E | R | T | P | S | M | K | T | S | I | V | E | A | I | A | N | - | - | - | - | - | - | - | - | D | R | E | Q | A | K | V | L | L | T | - | - | - | - | - | - | - | - | - | - | - | E | S | E | Y | N | R | Y | K | E | N | L | V | L | I | D | F | - | D | Y | - | - | - | - | - | - | - | - | - | - | - | - | - | I | P | D | N | I | A | S | N | I | V | N | Y | Y | N | S | Y | K | L | P | P | R | G | K | I | Y | S | Y | F | V | K | A | G | L | S | K | L | T | N | S | I | N | E | F | - | - | - | - | - | - | - | - | - | - | - |
| - | - | I | T | L | E | N | L | I | D | I | G | I | L | L | G | T | D | Y | N | P | - | D | G | F | E | G | I | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | K | K | A | L | Q | L | V | K | A | Y | - | - | - | - | - | - | - | - | - | G | G | I | E | K | I | P | - | - | - | - | K | P | I | L | K | S | P | I | E | V | - | D | V | I | A | I | K | K | Y | F | L | Q | - | P | Q | V | T | D | - | - | N | Y | R | I | E | W | H | T | P | D | P | D | A | V | K | R | I | L | V | D | E | H | D | F | S | I | D | R | V | S | T | A | L | E | R | Y | V | K | A | F | K | E | N | I | R | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| V | D | - | D | V | E | Q | F | I | S | L | K | A | I | M | G | D | L | G | D | N | I | R | G | V | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | G | I | G | A | K | R | G | Y | N | I | I | R | E | F | G | - | - | - | - | - | N | V | L | D | I | I | D | Q | L | P | L | P | G | K | Q | K | Y | I | Q | N | L | N | A | S | E | E | L | L | F | R | N | L | I | L | V | D | L | P | T | Y | - | - | - | - | - | - | - | - | - | - | - | - | - | C | V | D | A | I | A | A | V | G | Q | D | V | L | D | K | - | - | - | - | - | - | - | F | T | K | D | I | L | E | I | A | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
