solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1bvsa2 ( 64 )
d1cuka2 ( 65 )
d1dgsa1 ( 401 ) rwpeacpecghrlvkegkvhrcpnplCpakrfeaIrhYAsrkAMdIegLg
d1kfta- ( 23 )
d1x2ia1 ( 2 )
d2a1ja1 ( 837 )
d2a1jb1 ( 220 )
d2csba1 ( 351 )
d2csba2 ( 294 )
d2csba3 ( 410 )
d2csba4 ( 465 )
d2duya1 ( 12 )
d2edua1 ( 8 )
d3bzca1 ( 474 )
d3bzca2 ( 564 )
60 70 80 90 100
d1bvsa2 ( 64 ) da
d1cuka2 ( 65 ) nk
d1dgsa1 ( 451 ) eklIerLlekglVrdVAdLYhLrkedlLglermgeksAqnLlrqIeeSkh
d1kfta- ( 23 )
d1x2ia1 ( 2 ) alt
d2a1ja1 ( 837 )
d2a1jb1 ( 220 ) adllmekleqd
d2csba1 ( 351 )
d2csba2 ( 294 )
d2csba3 ( 410 )
d2csba4 ( 465 )
d2duya1 ( 12 )
d2edua1 ( 8 ) ekaedcwelqispe
d3bzca1 ( 474 ) yqhdvs----qlklars
d3bzca2 ( 564 )
110 120 130 140 150
d1bvsa2 ( 66 ) enrdlflaLlsVs----gvgprlAmaTlavh--daaaLr-ala------s
d1cuka2 ( 67 ) qertLfkeLiktn----gVgpklAlaIlsgm--saqqFVnAVere----e
d1dgsa1 ( 501 ) rGLerLLyALgLp----gVgevlArnLArrFg-tMdrLleA--------s
d1kfta- ( 23 ) tssle-------tiegvgpkRrqmllkymg-glqglrnAs--------
d1x2ia1 ( 5 ) laerqrliVegLp----hVsatlArrLlkhfg-sverVftA--------s
d2a1ja1 ( 837 ) pqdfLlkMp----gVnakNcrsLmhhVk-niaeLaal--------s
d2a1jb1 ( 231 ) fvsrVtecLttVk----sVnktdSqtLLttfg-sleqLiaA--------s
d2csba1 ( 351 ) rtlatLideh----gLspdaAdeLiehfe-siagIlatd--------
d2csba2 ( 294 ) iveCalkLqdry----gIredVAlçLArafd-gsisIattp--------
d2csba3 ( 410 ) laeLtkke----gVgrktAerLlrafg-nperVkqLAref----e
d2csba4 ( 465 ) pgyasLisIr----gIdrerAerLlkkyg-gyskVrea--------g
d2duya1 ( 12 ) lpqaqt--pVSLneasleeLa-LpgIgpvlArrIvegr----pYar
d2edua1 ( 22 ) llahGrqkildl---lneGsardlrslqrigpkkAqlivgwrelhgpFsq
d3bzca1 ( 487 ) ldavvedcvnavgVdVntAsaalLarIsgLnstlAqnIvahrdangaFrt
d3bzca2 ( 564 ) dnpLdaSaVhpeT--ypLVqrIaadterdirsLigd---------
aaa aaaaaaaaaa aaaaa
160 170 180 190
d1bvsa2 ( 106 ) vasLt-Vp------------gIg-rgAerivle--ladkvgp--v
d1cuka2 ( 107 ) vgaLvklp------------gIg-ktAe-LIve--MkdFglgdlF
d1dgsa1 ( 538 ) leeLieveeVgeltAraIletLkdpaFrdLvrr--LkeAgVsmesk
d1kfta- ( 55 ) veeiakvp------------gisqglAekifwS--lkh
d1x2ia1 ( 42 ) vaeLmkVe------------gIgekiAkeIrrv--it---apy-ie
d2a1ja1 ( 870 ) qdeLtsiLg-------------naanAkqLydf--ih---tsfaevv
d2a1jb1 ( 268 ) redLalCp------------gLgpqkArrLfdv--lh---epflkv
d2csba1 ( 385 ) leeIeryee----------grLseeAyraAveiq
d2csba2 ( 331 ) yrtLkdvÇp-----------dLtleeAksVn
d2csba3 ( 446 ) iekLasvegVg----------------erVlrs--Lv
d2csba4 ( 499 ) veeLred-gLt----------------daqIre--Lkglk
d2duya1 ( 52 ) vedLlkVk------------gIgpatLerLrpyLrp
d2edua1 ( 69 ) vedlerve------------gitgkqMesFlka--nilglaa---gq
d3bzca1 ( 537 ) rdeLkkVs------------rLgektfeqAagfLrvmng
d3bzca2 ( 598 ) safLkrldpkkFtde-----tfglptVtdIlke--Ldkpgrdprp-e
aaaa aaaaaaaaa
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| DNA helicase RuvA subunit, middle domain | DNA helicase RuvA subunit, middle domain | Mycobacterium leprae [TaxId: 1769] | 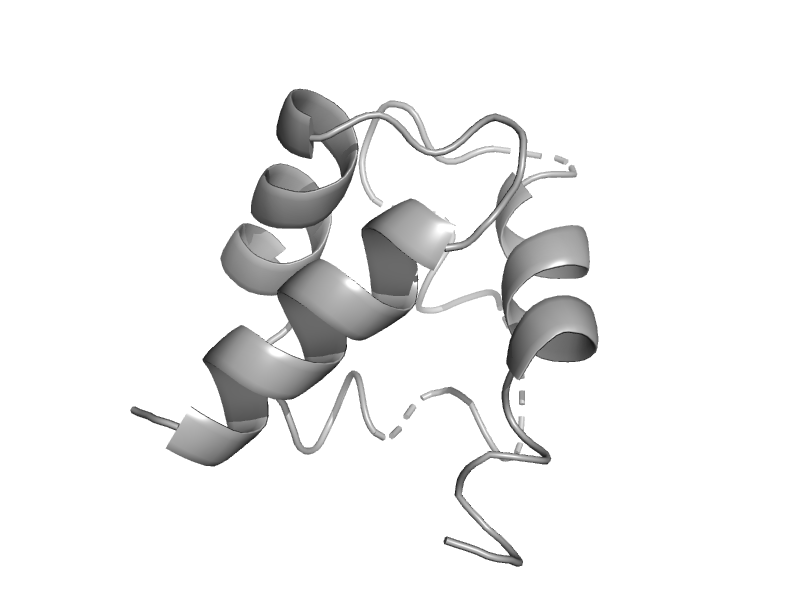 | view | |
| DNA helicase RuvA subunit, middle domain | DNA helicase RuvA subunit, middle domain | Escherichia coli [TaxId: 562] | 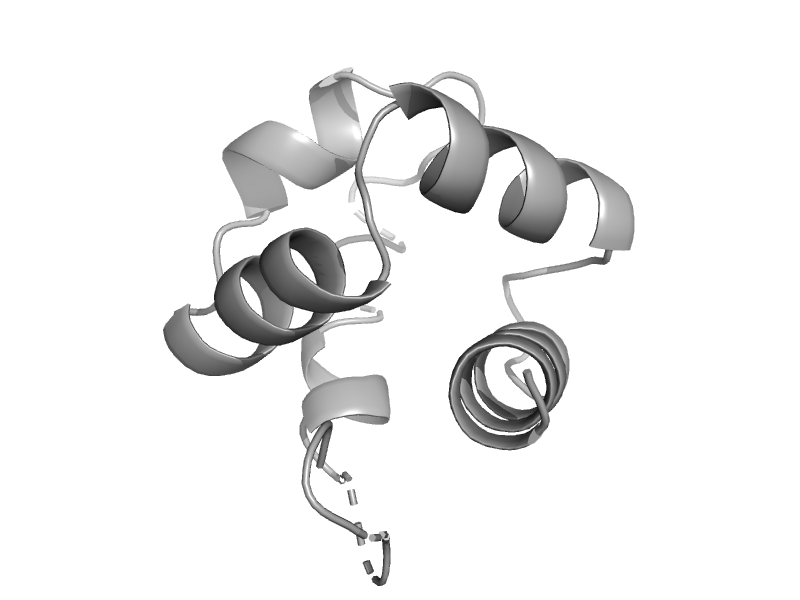 | view | |
| NAD+-dependent DNA ligase, domain 3 | NAD+-dependent DNA ligase, domain 3 | Thermus filiformis [TaxId: 276] | 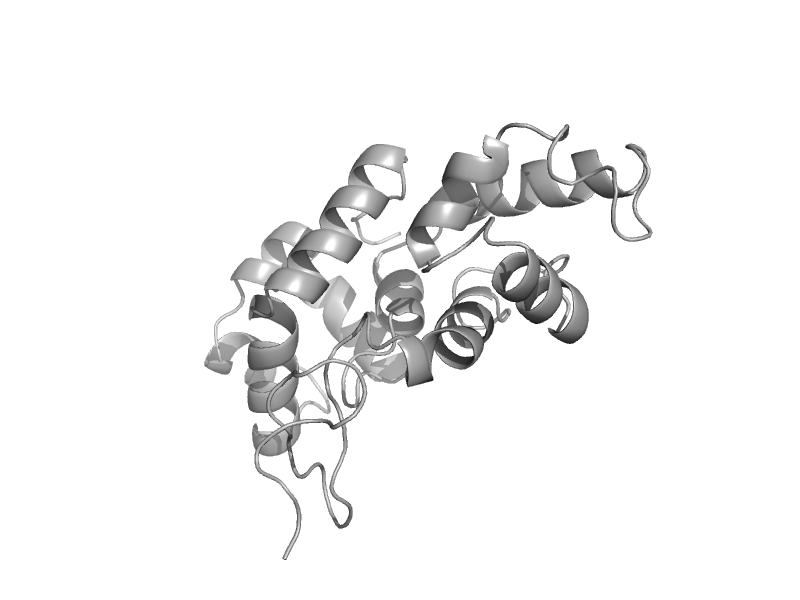 | view | |
| Excinuclease UvrC C-terminal domain | Excinuclease UvrC C-terminal domain | Escherichia coli [TaxId: 562] | 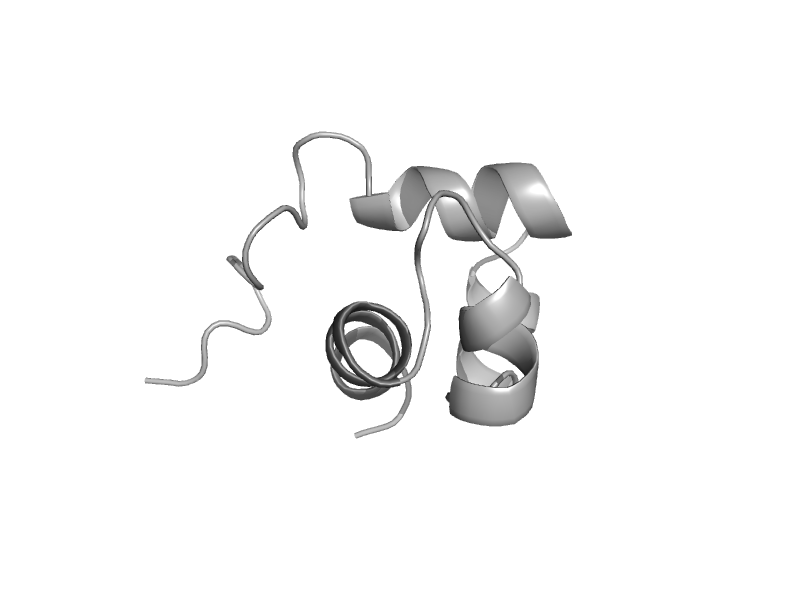 | view | |
| ATP-dependent RNA helicase PF2015 | Hef domain-like | Pyrococcus furiosus [TaxId: 2261] | 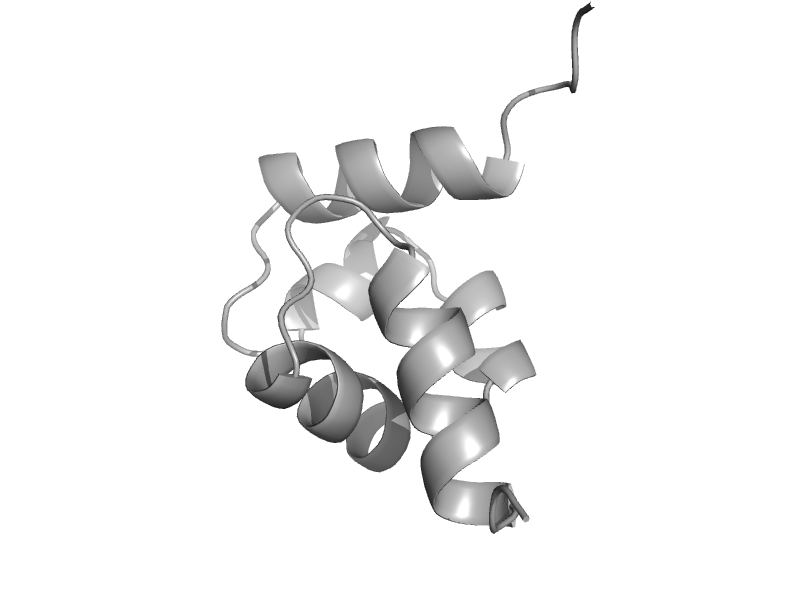 | view | |
| DNA repair endonuclease XPF | Hef domain-like | Human (Homo sapiens) [TaxId: 9606] | 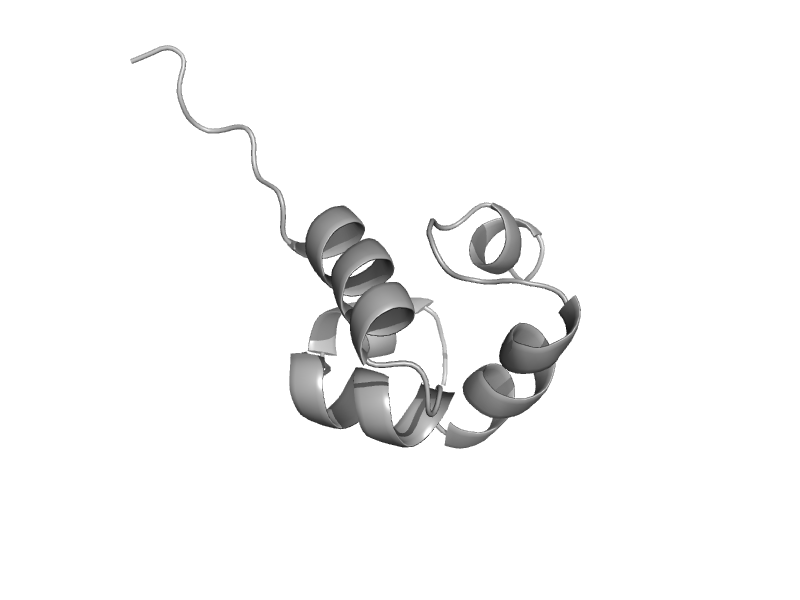 | view | |
| DNA excision repair protein ERCC-1 | Hef domain-like | Human (Homo sapiens) [TaxId: 9606] | 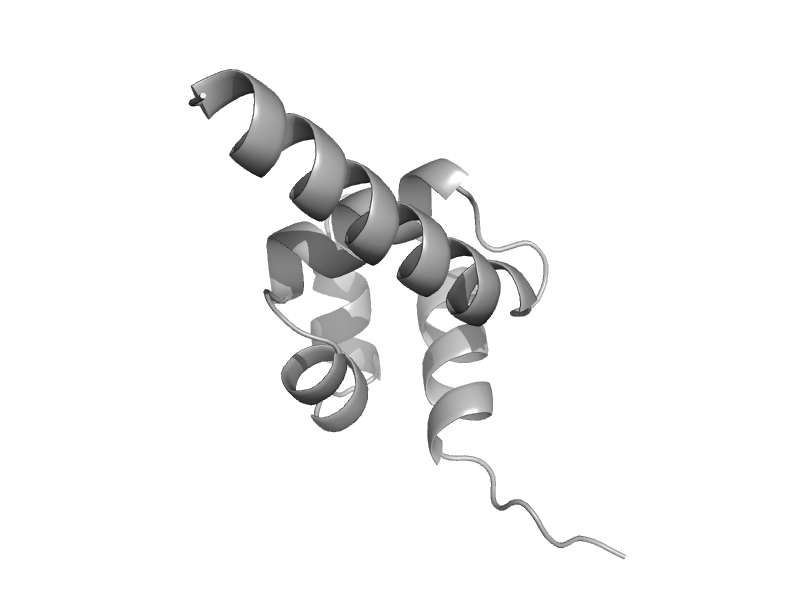 | view | |
| Topoisomerase V | Topoisomerase V repeat domain | Methanopyrus kandleri [TaxId: 2320] | 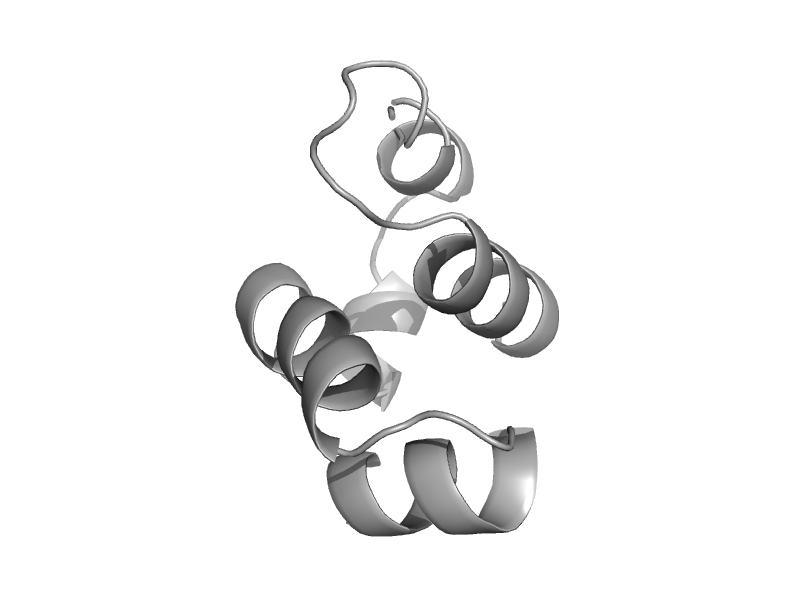 | view | |
| Topoisomerase V | Topoisomerase V repeat domain | Methanopyrus kandleri [TaxId: 2320] | 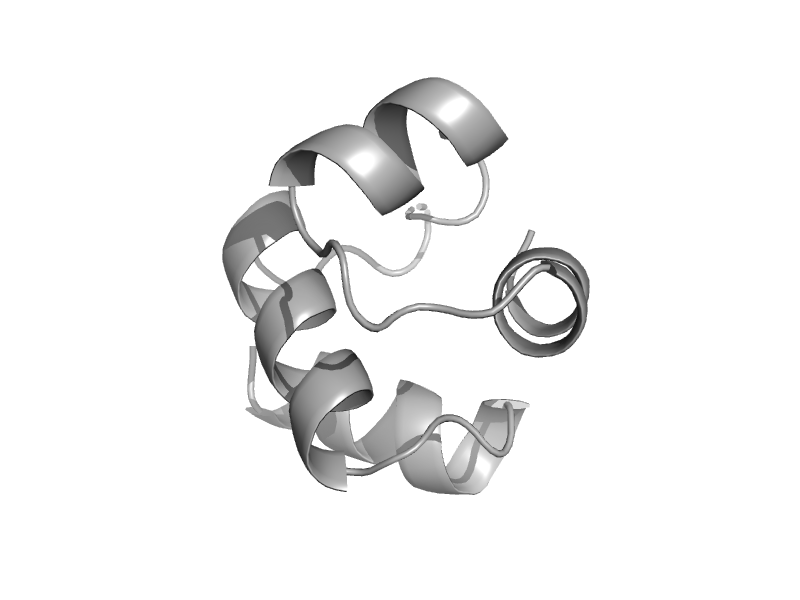 | view | |
| Topoisomerase V | Topoisomerase V repeat domain | Methanopyrus kandleri [TaxId: 2320] | 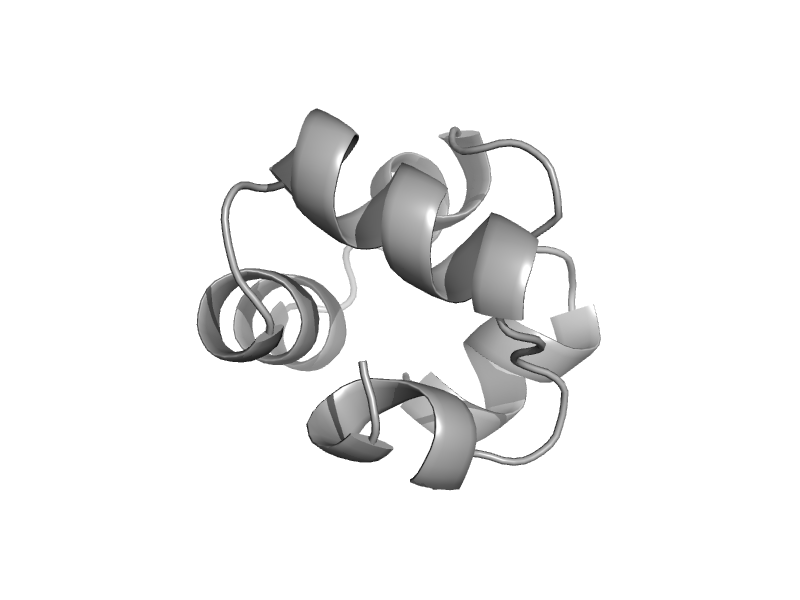 | view | |
| Topoisomerase V | Topoisomerase V repeat domain | Methanopyrus kandleri [TaxId: 2320] | 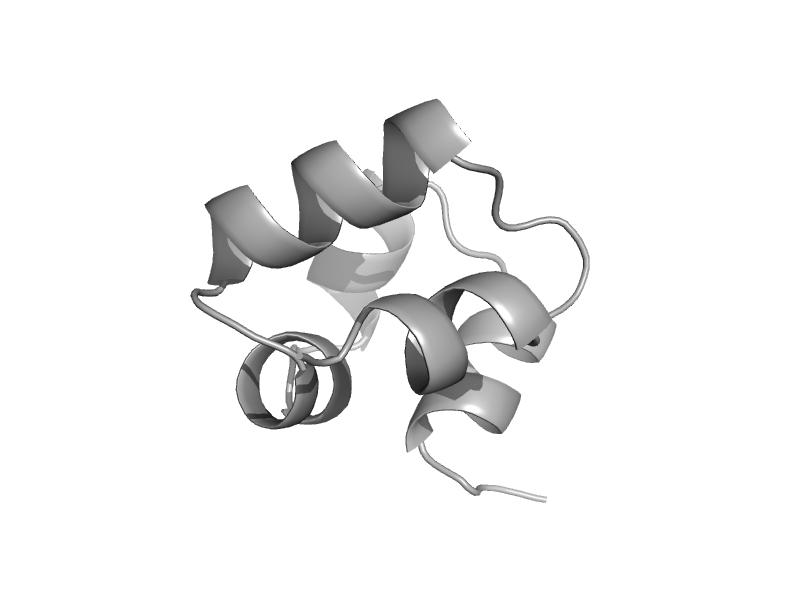 | view | |
| Uncharacterized protein TTHA1967 | ComEA-like | Thermus thermophilus [TaxId: 274] | 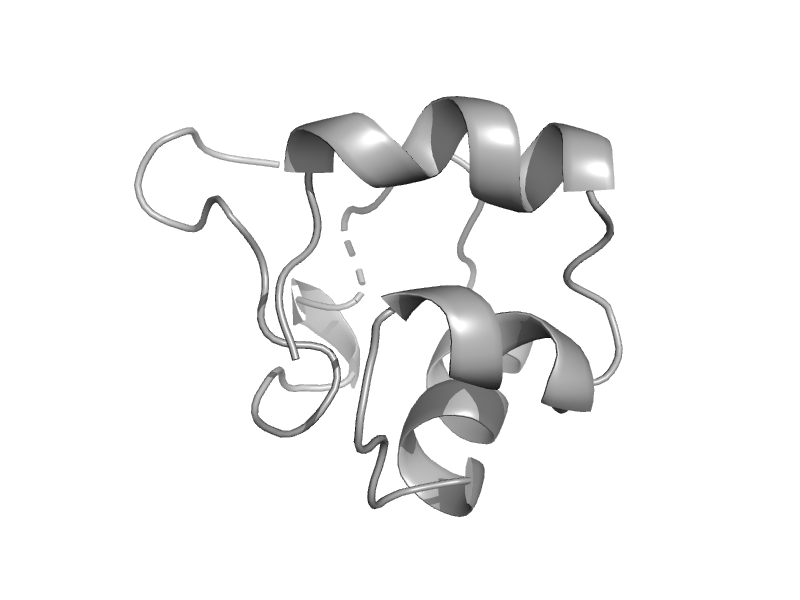 | view | |
| KIF22, C-terminal domain | ComEA-like | Human (Homo sapiens) [TaxId: 9606] | 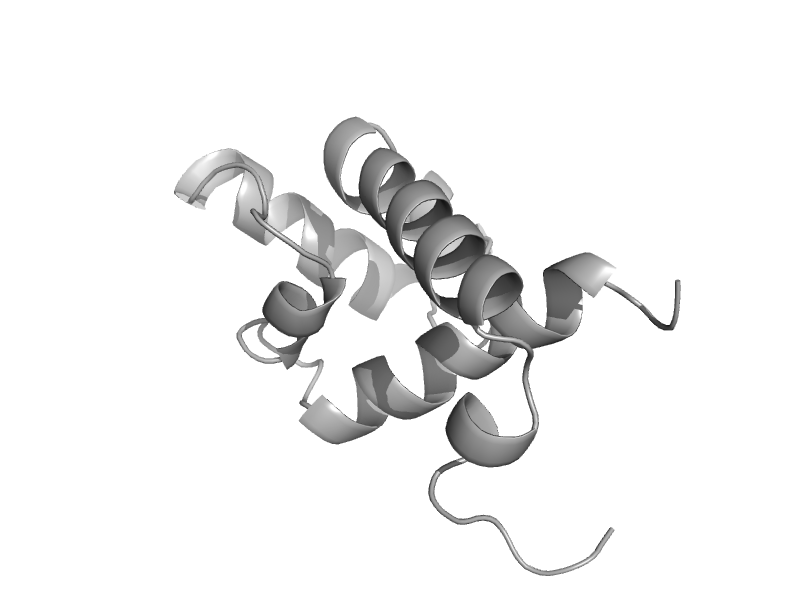 | view | |
| Transcriptional accessory factor Tex | Tex HhH-containing domain-like | Pseudomonas aeruginosa [TaxId: 287] | 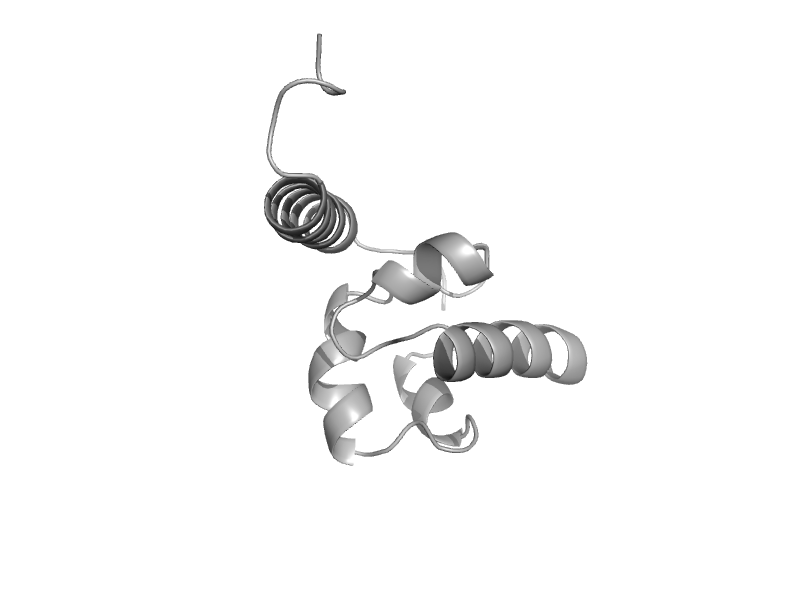 | view | |
| Transcriptional accessory factor Tex | Tex HhH-containing domain-like | Pseudomonas aeruginosa [TaxId: 287] | 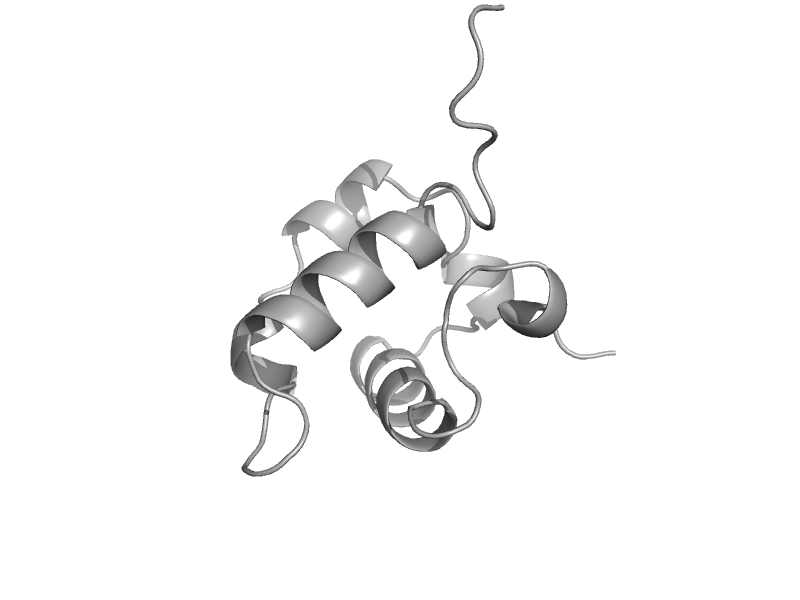 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | A | E | N | R | D | L | F | L | A | L | L | S | V | S | - | - | - | - | G | V | G | P | R | L | A | M | A | T | L | A | V | H | - | - | D | A | A | A | L | R | - | A | L | A | - | - | - | - | - | - | S | V | A | S | L | T | - | V | P | - | - | - | - | - | - | - | - | - | - | - | - | G | I | G | - | R | G | A | E | R | I | V | L | E | - | - | L | A | D | K | V | G | P | - | - | V | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | N | K | Q | E | R | T | L | F | K | E | L | I | K | T | N | - | - | - | - | G | V | G | P | K | L | A | L | A | I | L | S | G | M | - | - | S | A | Q | Q | F | V | N | A | V | E | R | E | - | - | - | - | E | V | G | A | L | V | K | L | P | - | - | - | - | - | - | - | - | - | - | - | - | G | I | G | - | K | T | A | E | - | L | I | V | E | - | - | M | K | D | F | G | L | G | D | L | F | - | - |
| R | W | P | E | A | C | P | E | C | G | H | R | L | V | K | E | G | K | V | H | R | C | P | N | P | L | C | P | A | K | R | F | E | A | I | R | H | Y | A | S | R | K | A | M | D | I | E | G | L | G | E | K | L | I | E | R | L | L | E | K | G | L | V | R | D | V | A | D | L | Y | H | L | R | K | E | D | L | L | G | L | E | R | M | G | E | K | S | A | Q | N | L | L | R | Q | I | E | E | S | K | H | R | G | L | E | R | L | L | Y | A | L | G | L | P | - | - | - | - | G | V | G | E | V | L | A | R | N | L | A | R | R | F | G | - | T | M | D | R | L | L | E | A | - | - | - | - | - | - | - | - | S | L | E | E | L | I | E | V | E | E | V | G | E | L | T | A | R | A | I | L | E | T | L | K | D | P | A | F | R | D | L | V | R | R | - | - | L | K | E | A | G | V | S | M | E | S | K | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | T | S | S | L | E | - | - | - | - | - | - | - | T | I | E | G | V | G | P | K | R | R | Q | M | L | L | K | Y | M | G | - | G | L | Q | G | L | R | N | A | S | - | - | - | - | - | - | - | - | V | E | E | I | A | K | V | P | - | - | - | - | - | - | - | - | - | - | - | - | G | I | S | Q | G | L | A | E | K | I | F | W | S | - | - | L | K | H | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | L | T | L | A | E | R | Q | R | L | I | V | E | G | L | P | - | - | - | - | H | V | S | A | T | L | A | R | R | L | L | K | H | F | G | - | S | V | E | R | V | F | T | A | - | - | - | - | - | - | - | - | S | V | A | E | L | M | K | V | E | - | - | - | - | - | - | - | - | - | - | - | - | G | I | G | E | K | I | A | K | E | I | R | R | V | - | - | I | T | - | - | - | A | P | Y | - | I | E | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | Q | D | F | L | L | K | M | P | - | - | - | - | G | V | N | A | K | N | C | R | S | L | M | H | H | V | K | - | N | I | A | E | L | A | A | L | - | - | - | - | - | - | - | - | S | Q | D | E | L | T | S | I | L | G | - | - | - | - | - | - | - | - | - | - | - | - | - | N | A | A | N | A | K | Q | L | Y | D | F | - | - | I | H | - | - | - | T | S | F | A | E | V | V |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | A | D | L | L | M | E | K | L | E | Q | D | F | V | S | R | V | T | E | C | L | T | T | V | K | - | - | - | - | S | V | N | K | T | D | S | Q | T | L | L | T | T | F | G | - | S | L | E | Q | L | I | A | A | - | - | - | - | - | - | - | - | S | R | E | D | L | A | L | C | P | - | - | - | - | - | - | - | - | - | - | - | - | G | L | G | P | Q | K | A | R | R | L | F | D | V | - | - | L | H | - | - | - | E | P | F | L | K | V | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | T | L | A | T | L | I | D | E | H | - | - | - | - | G | L | S | P | D | A | A | D | E | L | I | E | H | F | E | - | S | I | A | G | I | L | A | T | D | - | - | - | - | - | - | - | - | L | E | E | I | E | R | Y | E | E | - | - | - | - | - | - | - | - | - | - | G | R | L | S | E | E | A | Y | R | A | A | V | E | I | Q | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | I | V | E | C | A | L | K | L | Q | D | R | Y | - | - | - | - | G | I | R | E | D | V | A | L | C | L | A | R | A | F | D | - | G | S | I | S | I | A | T | T | P | - | - | - | - | - | - | - | - | Y | R | T | L | K | D | V | C | P | - | - | - | - | - | - | - | - | - | - | - | D | L | T | L | E | E | A | K | S | V | N | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | A | E | L | T | K | K | E | - | - | - | - | G | V | G | R | K | T | A | E | R | L | L | R | A | F | G | - | N | P | E | R | V | K | Q | L | A | R | E | F | - | - | - | - | E | I | E | K | L | A | S | V | E | G | V | G | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | R | V | L | R | S | - | - | L | V | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | G | Y | A | S | L | I | S | I | R | - | - | - | - | G | I | D | R | E | R | A | E | R | L | L | K | K | Y | G | - | G | Y | S | K | V | R | E | A | - | - | - | - | - | - | - | - | G | V | E | E | L | R | E | D | - | G | L | T | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | A | Q | I | R | E | - | - | L | K | G | L | K | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | L | P | Q | A | Q | T | - | - | P | V | S | L | N | E | A | S | L | E | E | L | A | - | L | P | G | I | G | P | V | L | A | R | R | I | V | E | G | R | - | - | - | - | P | Y | A | R | V | E | D | L | L | K | V | K | - | - | - | - | - | - | - | - | - | - | - | - | G | I | G | P | A | T | L | E | R | L | R | P | Y | L | R | P | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | K | A | E | D | C | W | E | L | Q | I | S | P | E | L | L | A | H | G | R | Q | K | I | L | D | L | - | - | - | L | N | E | G | S | A | R | D | L | R | S | L | Q | R | I | G | P | K | K | A | Q | L | I | V | G | W | R | E | L | H | G | P | F | S | Q | V | E | D | L | E | R | V | E | - | - | - | - | - | - | - | - | - | - | - | - | G | I | T | G | K | Q | M | E | S | F | L | K | A | - | - | N | I | L | G | L | A | A | - | - | - | G | Q |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Y | Q | H | D | V | S | - | - | - | - | Q | L | K | L | A | R | S | L | D | A | V | V | E | D | C | V | N | A | V | G | V | D | V | N | T | A | S | A | A | L | L | A | R | I | S | G | L | N | S | T | L | A | Q | N | I | V | A | H | R | D | A | N | G | A | F | R | T | R | D | E | L | K | K | V | S | - | - | - | - | - | - | - | - | - | - | - | - | R | L | G | E | K | T | F | E | Q | A | A | G | F | L | R | V | M | N | G | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | D | N | P | L | D | A | S | A | V | H | P | E | T | - | - | Y | P | L | V | Q | R | I | A | A | D | T | E | R | D | I | R | S | L | I | G | D | - | - | - | - | - | - | - | - | - | S | A | F | L | K | R | L | D | P | K | K | F | T | D | E | - | - | - | - | - | T | F | G | L | P | T | V | T | D | I | L | K | E | - | - | L | D | K | P | G | R | D | P | R | P | - | E |
