solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1hlra6 ( 81 ) qp-enlhpLqkAWvlhgGAqcGfcSpgFivsAkgLLdtnadPsredVrdw
d1n62a1 ( 82 ) apdgtlsaLqeGFrmmhGlqcgyctpgMimrShrLLqenpsPteaeIrfg
d3eubs2 ( 93 ) stktrlhpVqerIakshGsqcgfctpgIvmsMytllrnqpePtveeIeda
d5y6qa2 ( 83 ) sasgelhpVqrCFvendAfqcgyctpgQimsAvaCikeghagsddeIrey
aaaaaaaa aaaaaaaaaaaaa aaaaaaa
60 70 80 90 100
d1hlra6 ( 130 ) FqkhrnaCrcTgykpLVdAVmdAAaVingkkpetdLefkmpadgriwgsk
d1n62a1 ( 132 ) Ig--gnlCrcTgyqnIVkAIqyAaakingvpfee
d3eubs2 ( 143 ) fq--gnlCrcTgyrPIlqgFrtf
d5y6qa2 ( 133 ) Ms--gnlCrcgAyphIidAIkqAaaemak
aaaaaaaaaaaaaa
110
d1hlra6 ( 180 ) yprptAvakVTgtl
d1n62a1
d3eubs2
d5y6qa2
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| automated matches | automated matches | Desulfovibrio gigas [TaxId: 879] | 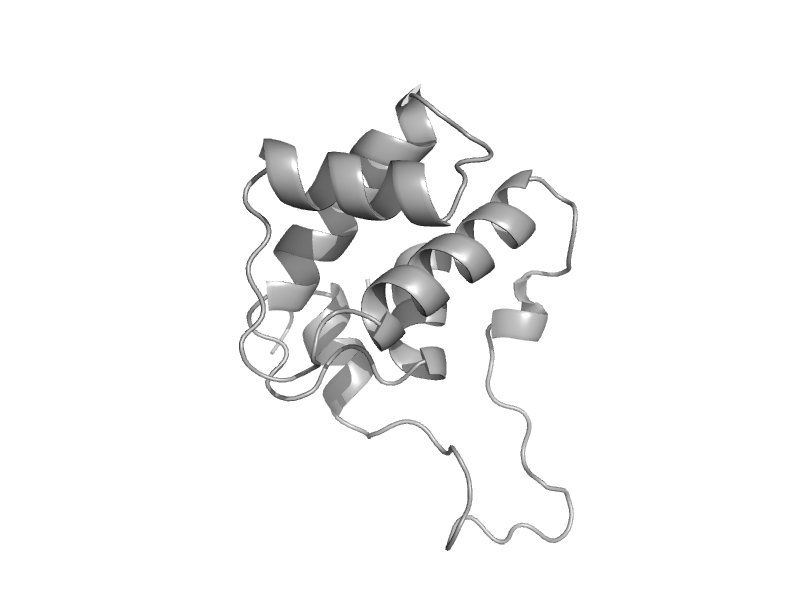 | view | |
| Carbon monoxide (CO) dehydrogenase iron-sulfur protein, C-domain | CO dehydrogenase ISP C-domain like | Oligotropha carboxidovorans, formerly Pseudomonas carboxydovorans [TaxId: 40137] | 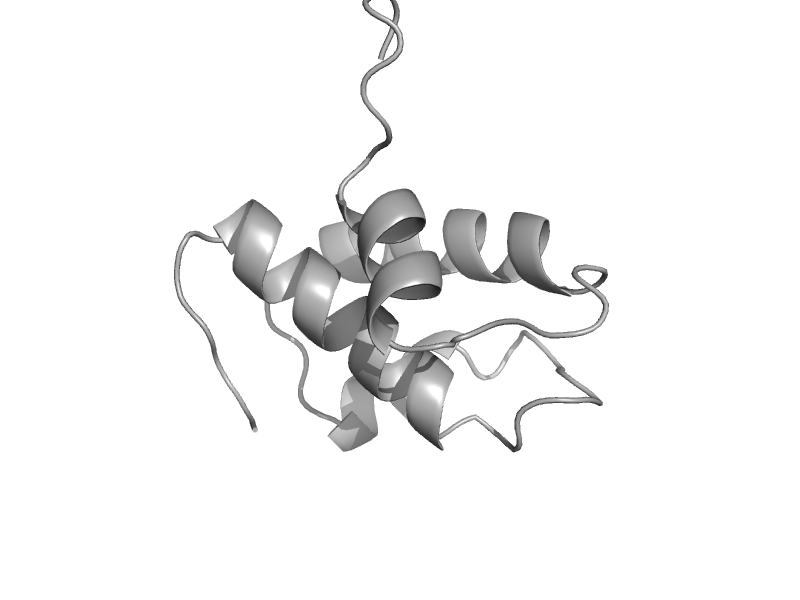 | view | |
| automated matches | CO dehydrogenase ISP C-domain like | Cow (Bos taurus) [TaxId: 9913] | 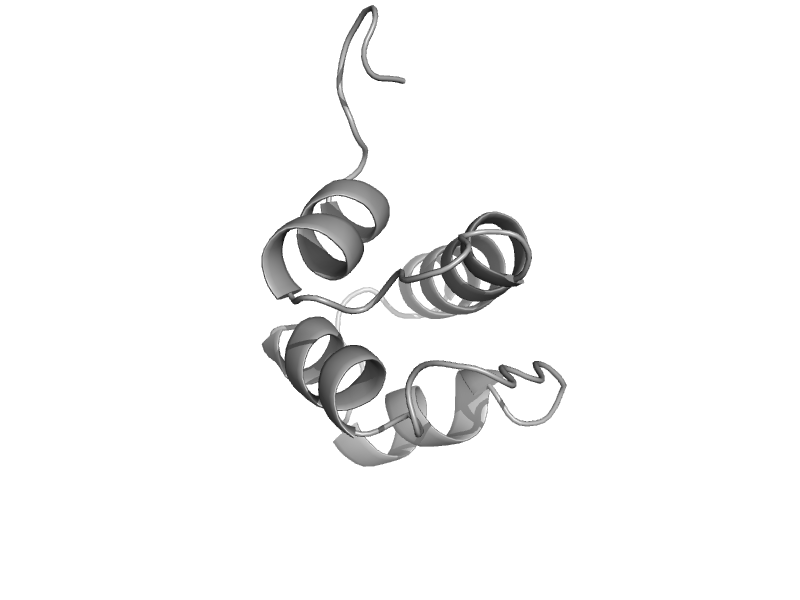 | view | |
| automated matches | automated matches | Methylobacillus sp. [TaxId: 194289] | 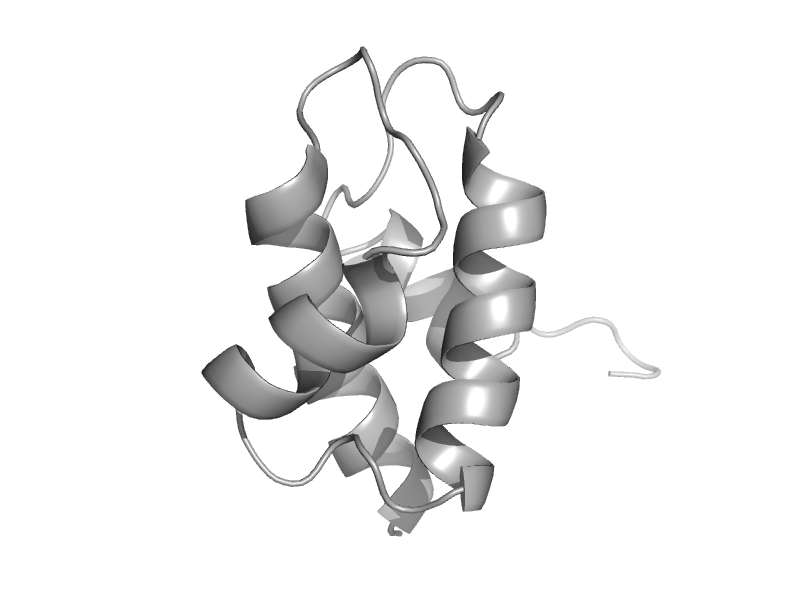 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Q | P | - | E | N | L | H | P | L | Q | K | A | W | V | L | H | G | G | A | Q | C | G | F | C | S | P | G | F | I | V | S | A | K | G | L | L | D | T | N | A | D | P | S | R | E | D | V | R | D | W | F | Q | K | H | R | N | A | C | R | C | T | G | Y | K | P | L | V | D | A | V | M | D | A | A | A | V | I | N | G | K | K | P | E | T | D | L | E | F | K | M | P | A | D | G | R | I | W | G | S | K | Y | P | R | P | T | A | V | A | K | V | T | G | T | L |
| A | P | D | G | T | L | S | A | L | Q | E | G | F | R | M | M | H | G | L | Q | C | G | Y | C | T | P | G | M | I | M | R | S | H | R | L | L | Q | E | N | P | S | P | T | E | A | E | I | R | F | G | I | G | - | - | G | N | L | C | R | C | T | G | Y | Q | N | I | V | K | A | I | Q | Y | A | A | A | K | I | N | G | V | P | F | E | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| S | T | K | T | R | L | H | P | V | Q | E | R | I | A | K | S | H | G | S | Q | C | G | F | C | T | P | G | I | V | M | S | M | Y | T | L | L | R | N | Q | P | E | P | T | V | E | E | I | E | D | A | F | Q | - | - | G | N | L | C | R | C | T | G | Y | R | P | I | L | Q | G | F | R | T | F | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| S | A | S | G | E | L | H | P | V | Q | R | C | F | V | E | N | D | A | F | Q | C | G | Y | C | T | P | G | Q | I | M | S | A | V | A | C | I | K | E | G | H | A | G | S | D | D | E | I | R | E | Y | M | S | - | - | G | N | L | C | R | C | G | A | Y | P | H | I | I | D | A | I | K | Q | A | A | A | E | M | A | K | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
