solvent inaccessible: UPPER CASE X
solvent accesible: lower case x
alpha helix: red x
solvent accesible: lower case x
alpha helix: red x
beta strand: blue x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
3 - 10 helix: maroon x
hydrogen bond to main chain amide: bold x
hydrogen bond to mainchain carbonyl: underline x
disulphide bond: cedilla
positive phi: italic x
disulphide bond: cedilla
positive phi: italic x
10 20 30 40 50
d1rcua- ( 2 )
d1t35a- ( 2 )
d1weha- ( 2 )
d2nx2a1 ( 2 )
d2q4oa- ( 9 ) q---------------------------
d3bq9a2 ( 139 ) rp---------------------------
d3imka1 ( 3 )
d3quaa- ( 16 )
d3uqza2 ( 72 )
d5wq3a- ( 42 ) wkhadpwrvlriqsefvaGfdaL---------------------------
d6bcoa1 ( 12 ) pkifrkkvcttfittekpTdAYGdLdFt
60 70 80 90 100
d1rcua- ( 2 ) kkVVVVGysg
d1t35a- ( 2 ) kticvfagsn
d1weha- ( 2 ) rlLAVFvssr
d2nx2a1 ( 2 ) kvLAITGYkp
d2q4oa- ( 10 ) ------------------------------------kSkFrrICVfcgss
d3bq9a2 ( 141 ) ------------------------------------qeePnMVVCWGghs
d3imka1 ( 3 )
d3quaa- ( 16 ) rqwAVCVYCAsg
d3uqza2 ( 72 ) psFsilddcYpwdLseIydaPvlLfykgnldLLkfpkVAVVGsra
d5wq3a- ( 65 ) ------------------------------------heMpkAVTVfGsar
d6bcoa1 ( 83 ) ysgrkhsnFLrLSdrtdpatVyslVtr------sWgfraPnlVVSVlGgs
bbbbb
110 120 130 140 150
d1rcua- ( 12 ) pv------nkspVselrdiCleLGrtLAkkg-ylVfngGr-dGV-eLVSq
d1t35a- ( 12 ) pg---------gneaykrkaaeLGvy-Aeqg-igLVyggsrvGL-gtiAd
d1weha- ( 12 ) ls------ped---plyarWvrYGeVLAeeg-FgLAcgGY-qGG-eALAr
d2nx2a1 ( 12 ) felgifkqddkaLyyIkkaIknrLiafLdegLewILISGq-lGVELWAAe
d2q4oa- ( 24 ) qg---------kkssyqdaAvdLGneLvsrn-IdLVygggsiGL-glVSq
d3bq9a2 ( 155 ) In------e-----ieykYTkdVGyhIGlrg-LnICTGCG-pgAmkGPMk
d3imka1 ( 3 ) epaItkIISGGq-tGAdraALd
d3quaa- ( 28 ) pt----------hpeLleLAaeVGssIAarg-wtLVSgGGnvSAMgaVAq
d3uqza2 ( 117 ) cs-----------kqgaksVekVIqgL--eneLVIVSGLa-kgIDtaAHA
d5wq3a- ( 79 ) ik------edh---pyYkaGveLGekLvaad-YAVVTGGg-pGLmeAPNk
d6bcoa1 ( 127 ) gg------pvlqtwLqdlLrrgLVraAqstg-AWIVTgGlhtgiGrHVGv
aaaaaaaaaaaaaa bbbb aaaa
160 170 180 190 200
d1rcua- ( 54 ) GVreag--------gtVvGILPd-----eeaGNp---yLsvavktgldf-
d1t35a- ( 52 ) aI-eng--------gtaiGV-psglfsgev-vhq---nLtelievngh--
d1weha- ( 51 ) GVkakg--------glVvGVTApaffperrgpNp---fVdlelpAatlp-
d2nx2a1 ( 61 ) AAyd---lqeeypdLkVaVitPFyeqe---------knwk-------epn
d2q4oa- ( 64 ) aVhdgg--------rhVIGIipkt-------------lvgevravadh--
d3bq9a2 ( 192 ) gAtiGHakqrv-eggrylGLTepgiiaae-ppnp---iVnelvilpdie-
d3imka1 ( 25 ) fAik---h-----hipyGGwVPkgRlAeggrVpetyqLqep-----tsdy
d3quaa- ( 67 ) aArakg--------ghTVGVIPkalvhrel-aDv---dAaelivtdtm--
d3uqza2 ( 154 ) Alqn---------ggkTIAVIGTG-ld---------vfyp-------kan
d5wq3a- ( 118 ) gAsean-------glSVGLgielph-----hlnp---yVdlglnFryff-
d6bcoa1 ( 170 ) AVrdhqtastgsskVvAMGvApWGvVrnrdmLinpkgsfparYrwrgdpe
aaaa bbbb b
210 220 230 240 250
d1rcua- ( 87 ) -qrSfvLLrnAdVVVSIGGe------igTaieIlgAYal-----------
d1t35a- ( 89 ) -erkaks-elAdgfIS-pgg------fgTyeelfevlcwaqig------i
d1weha- ( 89 ) -qrigrLldlGaGYLALpgg------vgTlaeLvlAwnllylr---rg--
d2nx2a1 ( 92 ) keqYeaVlaqAdyeaSlth-rpyesplQfkqKnqfFIdkS----------
d2q4oa- ( 101 ) -qrkaea-khSdAFIALPgg------ygTleeLleVitwaqlg------i
d3bq9a2 ( 236 ) -kRLeaFVrcAhGIVIFPgg------agTaeeLlyLLgiLmhp---dNqr
d3imka1 ( 63 ) skrtekNVldSdGTLIISh-g----ilggsalTeffAeqy----------
d3quaa- ( 103 ) -rRkreMehrSdAFIALPGg------igTleeFfeawtagylg------m
d3uqza2 ( 178 ) krlQdyIGndHLVLSeygp-geqplkfHfpaRnrIIaGLC----------
d5wq3a- ( 154 ) -ArktMFlkySqAFVCLPGg------fgTldELfeVlcmvq-----tgkv
d6bcoa1 ( 220 ) dgvefpLDynYsAFFLVDdGtygrlggenrFrlrFEsyVaqqktgvggtg
aaaaaaa bbbb aaaaaaaaaaaaa
260 270 280 290 300
d1rcua- ( 120 ) -gkpVILLr--gTggwTdrISqvLidgkyLdnrrive--IhqAw---tVe
d1t35a- ( 126 ) hqkpIglYnVngyFep---kvkysiqegfsneshlkl--ihsss---rPd
d1weha- ( 127 ) vGrpLAVd--pyWlgllkah-------geiapedvgl--LrvVa---dee
d2nx2a1 ( 131 ) --dGLLLLYdpekegsPkyMlgtAekrreq---dg---YpIyfIt-----
d2q4oa- ( 138 ) hdkpVGLLnVdgyYnsllsfidkaveegfisptarei--IvsAp---tAk
d3bq9a2 ( 276 ) QsLpVILTGpasSrdyFealdefIga--tigdeArql--YkiiId--dpa
d3imka1 ( 100 ) --kpclhIdLdr--isiedAAtlInswTvs---hhIqvLNIaGpragkdp
d3quaa- ( 141 ) hdkpLILLDpfghYdglLtwLrglvptgyvsqramds--Lvvvd---nv-
d3uqza2 ( 217 ) --rGVIVAeAk------rsgslitCerAee----g---rdvFAipgsild
d5wq3a- ( 192 ) tnFpIVLIgtefWaglvdwIrhrlveegmidekdVdr--M-lvtd--dld
d6bcoa1 ( 270 ) idIpVLLLLIdG-dekmLkRIedAtqa--------qL--pcLLVagsggA
bbbb aaaaaa bbb
310 320 330 340 350
d1rcua- ( 162 ) eAVqiIeqi
d1t35a- ( 171 ) eLieqqny
d1weha- ( 163 ) dLrrFLrsl
d2nx2a1 ( 168 ) --mddlrvtvee
d2q4oa- ( 183 ) eLVkkLee
d3bq9a2 ( 320 ) aVAqhMhagmaaVkqyRrdsgDayyfNwtL
d3imka1 ( 143 ) eIyqaTdLLevFla
d3quaa- ( 186 ) aALeaCap
d3uqza2 ( 254 ) glSdgChhliqega-------klVtsGqdVlaeFef
d5wq3a- ( 237 ) qAvkfIvdaha---------g--------l
d6bcoa1 ( 309 ) aDClvetleeardriRryfpkgdpevLqaqverImtrkeLltvYssedgs
aaaaaa
360
d1rcua-
d1t35a-
d1weha-
d2nx2a1
d2q4oa-
d3bq9a2
d3imka1
d3quaa-
d3uqza2
d5wq3a-
d6bcoa1 ( 372 ) eeFetIvlrAlvkacgs
| Domain ID | Name | Family | Source | Domain | STRING-DB |
|---|---|---|---|---|---|
| Hypothetical protein TM1055 | MoCo carrier protein-like | Thermotoga maritima [TaxId: 2336] | 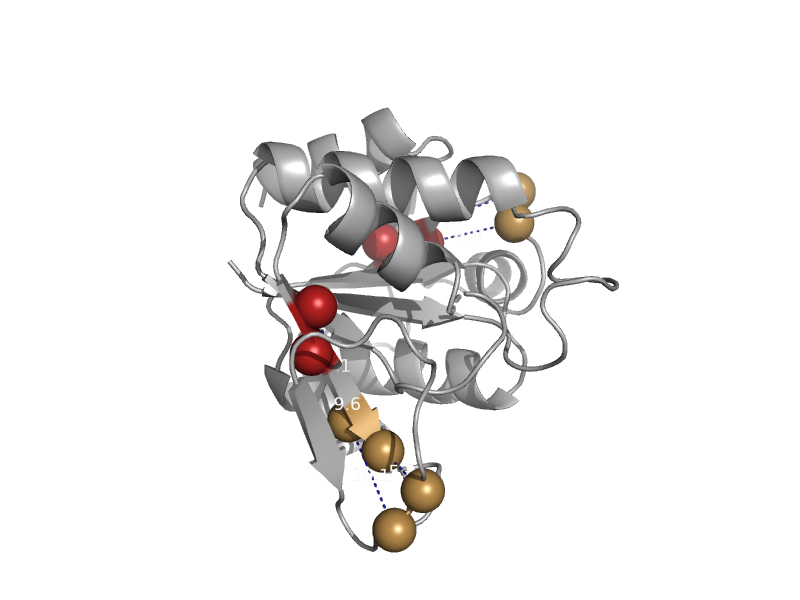 | view | |
| Hypothetical protein YvdD | MoCo carrier protein-like | Bacillus subtilis [TaxId: 1423] | 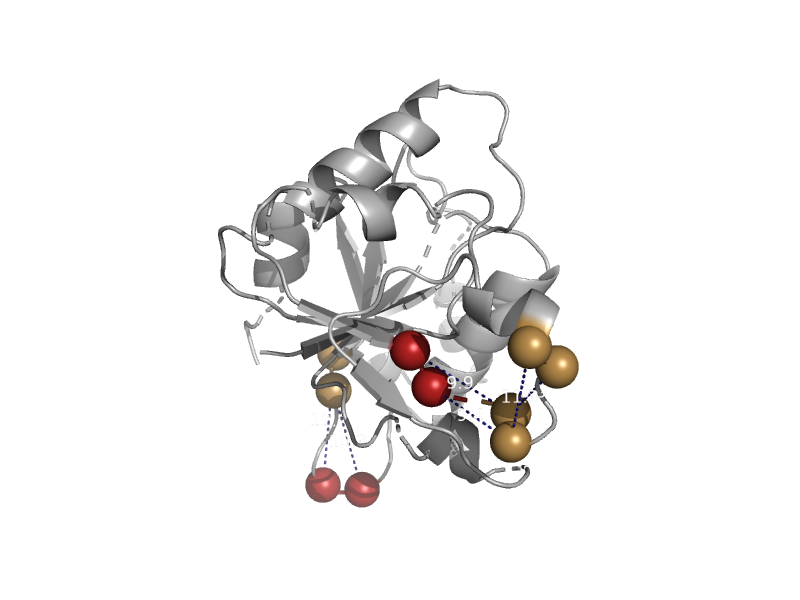 | view | |
| Hypothetical protein TT1887 (TTHA0294) | MoCo carrier protein-like | Thermus thermophilus [TaxId: 274] | 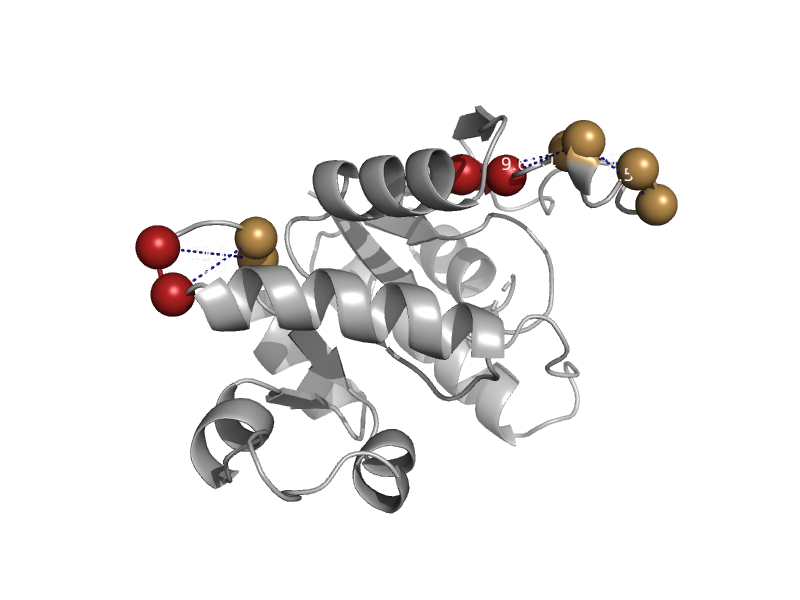 | view | |
| Hypothetical protein YpsA | YpsA-like | Bacillus subtilis [TaxId: 1423] | 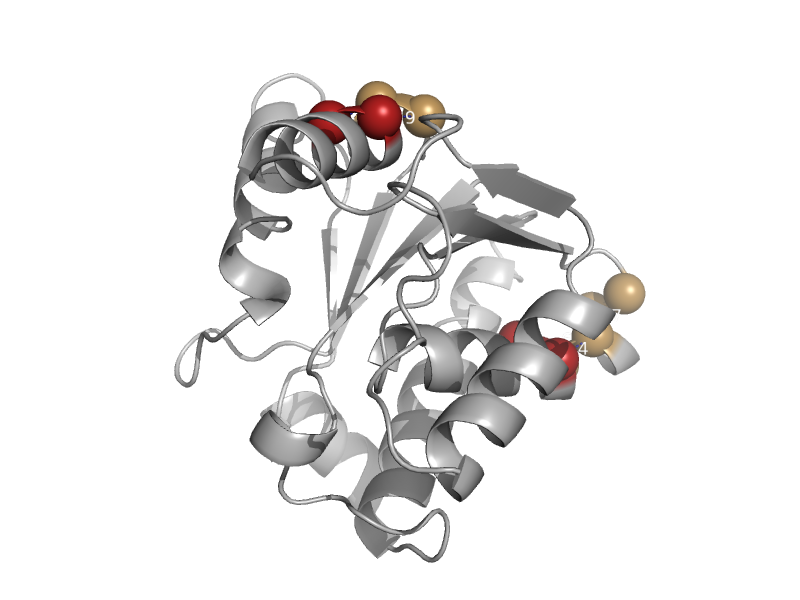 | view | |
| Hypothetical protein At2g37210/T2N18.3 | MoCo carrier protein-like | Thale cress (Arabidopsis thaliana) [TaxId: 3702] | 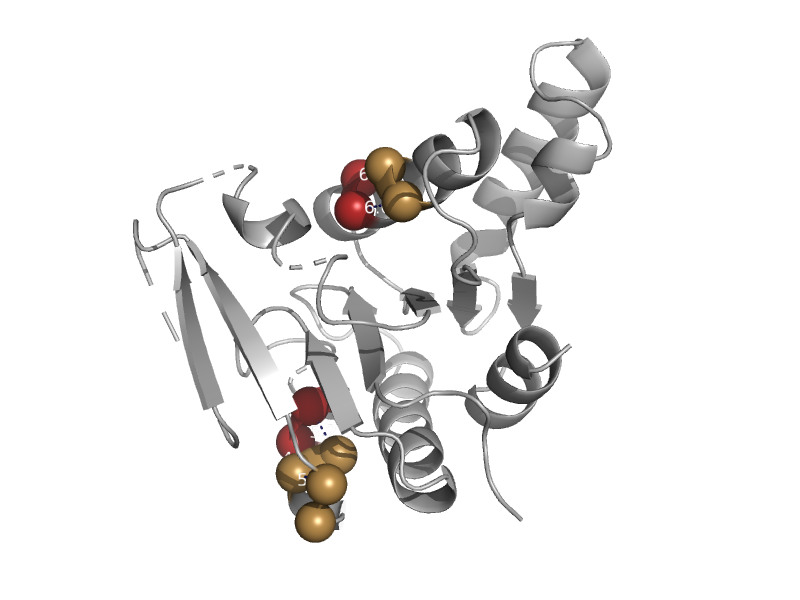 | view | |
| automated matches | automated matches | Idiomarina baltica [TaxId: 314276] | 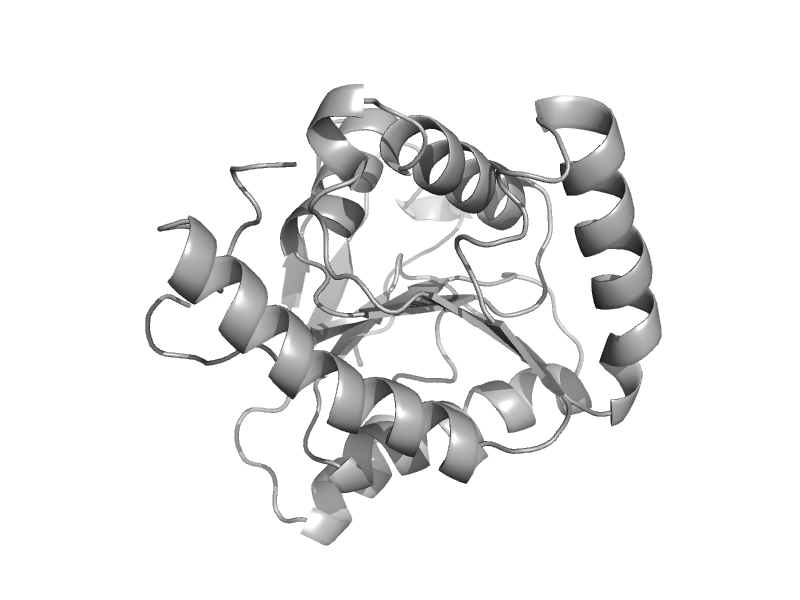 | view | |
| Putative molybdenum carrier protein (YP_461806.1) | Circularly perumited SLOG domain | Syntrophus aciditrophicus SB [TaxId: 56780] | 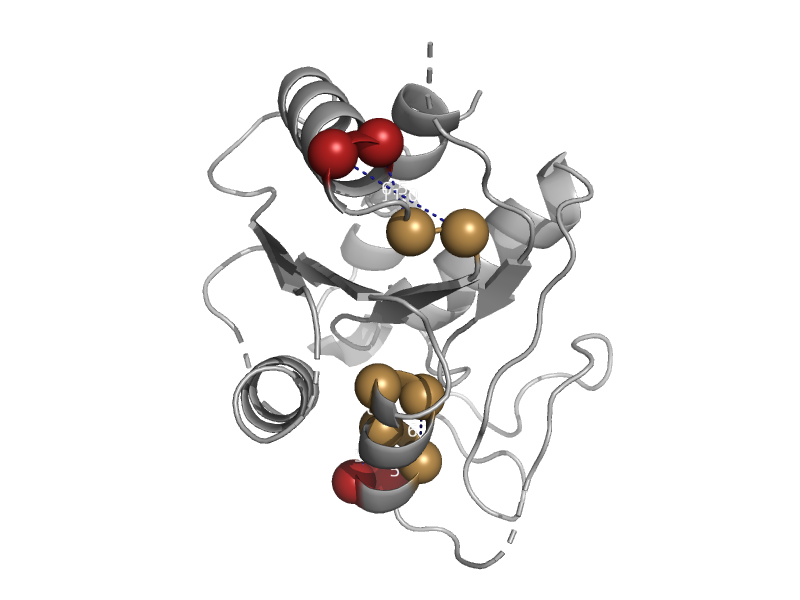 | view | |
| automated matches | automated matches | Mycobacterium smegmatis [TaxId: 246196] | 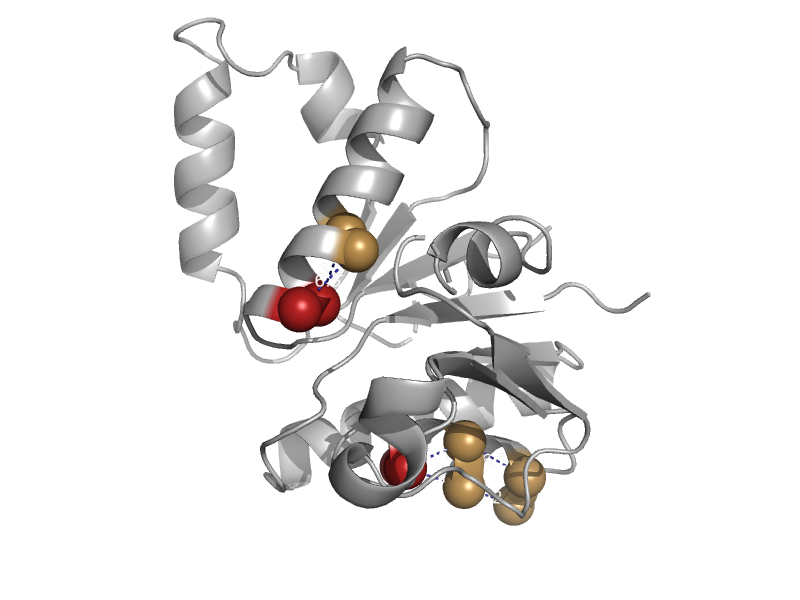 | view | |
| DNA processing protein A (DprA) | SLOG domain from DNA-Processing proteins | Pneumococcus (Streptococcus pneumoniae) [TaxId: 1313] | 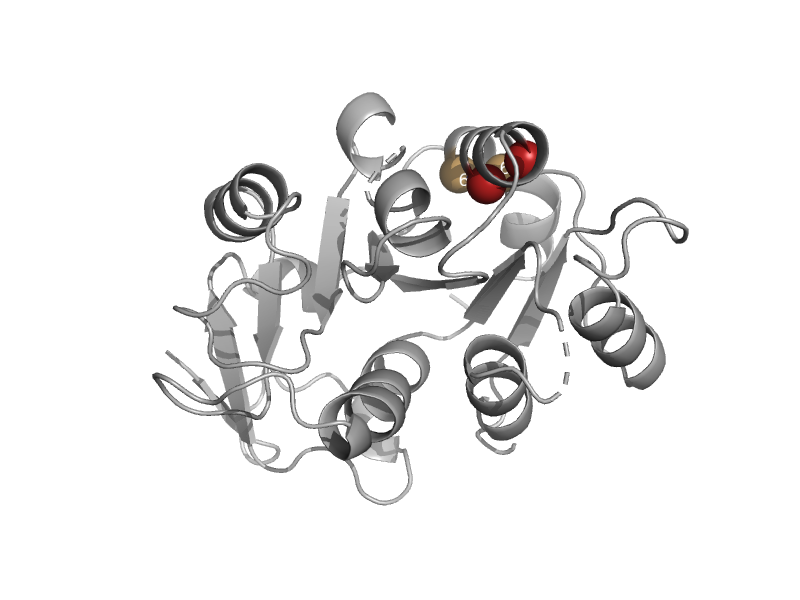 | view | |
| automated matches | automated matches | Corynebacterium glutamicum [TaxId: 1718] | 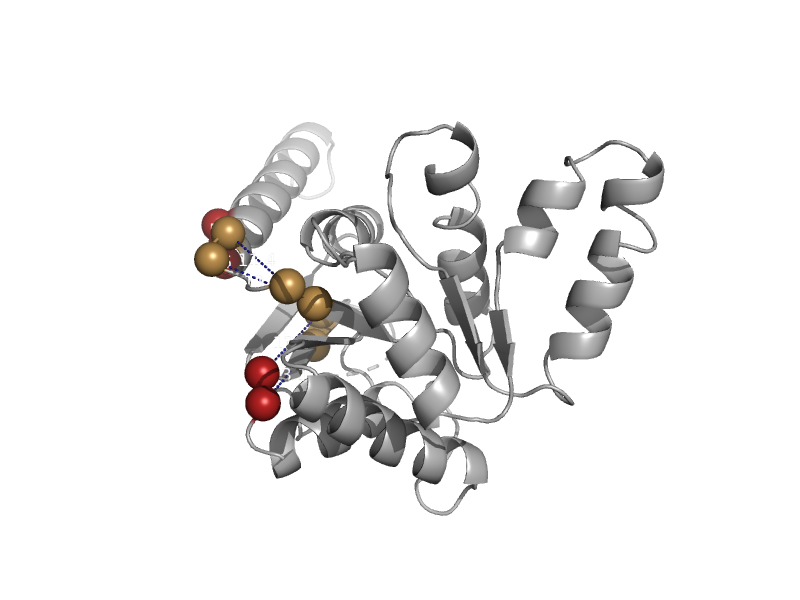 | view | |
| Calcium-activated non-selective cation channel TRPM4 | SMF/DprA-LOG (SLOG) domain from TPRM channels | Mouse (Mus musculus) [TaxId: 10090] | 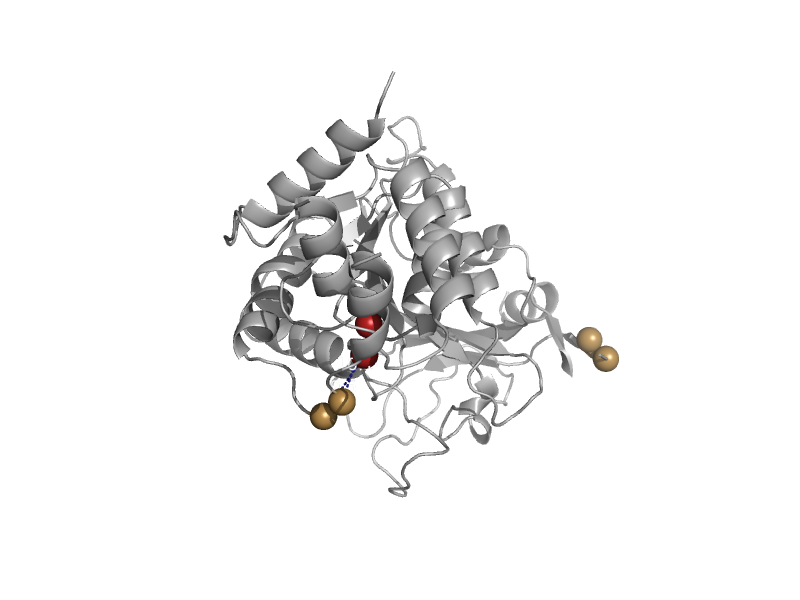 | view |
No outliers
| 10 | 20 | 30 | 40 | 50 | 60 | 70 | 80 | 90 | 100 | 110 | 120 | 130 | 140 | 150 | 160 | 170 | 180 | 190 | 200 | 210 | 220 | 230 | 240 | 250 | 260 | 270 | 280 | 290 | 300 | 310 | 320 | 330 | 340 | 350 | 360 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | K | V | V | V | V | G | Y | S | G | P | V | - | - | - | - | - | - | N | K | S | P | V | S | E | L | R | D | I | C | L | E | L | G | R | T | L | A | K | K | G | - | Y | L | V | F | N | G | G | R | - | D | G | V | - | E | L | V | S | Q | G | V | R | E | A | G | - | - | - | - | - | - | - | - | G | T | V | V | G | I | L | P | D | - | - | - | - | - | E | E | A | G | N | P | - | - | - | Y | L | S | V | A | V | K | T | G | L | D | F | - | - | Q | R | S | F | V | L | L | R | N | A | D | V | V | V | S | I | G | G | E | - | - | - | - | - | - | I | G | T | A | I | E | I | L | G | A | Y | A | L | - | - | - | - | - | - | - | - | - | - | - | - | G | K | P | V | I | L | L | R | - | - | G | T | G | G | W | T | D | R | I | S | Q | V | L | I | D | G | K | Y | L | D | N | R | R | I | V | E | - | - | I | H | Q | A | W | - | - | - | T | V | E | E | A | V | Q | I | I | E | Q | I | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | T | I | C | V | F | A | G | S | N | P | G | - | - | - | - | - | - | - | - | - | G | N | E | A | Y | K | R | K | A | A | E | L | G | V | Y | - | A | E | Q | G | - | I | G | L | V | Y | G | G | S | R | V | G | L | - | G | T | I | A | D | A | I | - | E | N | G | - | - | - | - | - | - | - | - | G | T | A | I | G | V | - | P | S | G | L | F | S | G | E | V | - | V | H | Q | - | - | - | N | L | T | E | L | I | E | V | N | G | H | - | - | - | E | R | K | A | K | S | - | E | L | A | D | G | F | I | S | - | P | G | G | - | - | - | - | - | - | F | G | T | Y | E | E | L | F | E | V | L | C | W | A | Q | I | G | - | - | - | - | - | - | I | H | Q | K | P | I | G | L | Y | N | V | N | G | Y | F | E | P | - | - | - | K | V | K | Y | S | I | Q | E | G | F | S | N | E | S | H | L | K | L | - | - | I | H | S | S | S | - | - | - | R | P | D | E | L | I | E | Q | Q | N | Y | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | L | L | A | V | F | V | S | S | R | L | S | - | - | - | - | - | - | P | E | D | - | - | - | P | L | Y | A | R | W | V | R | Y | G | E | V | L | A | E | E | G | - | F | G | L | A | C | G | G | Y | - | Q | G | G | - | E | A | L | A | R | G | V | K | A | K | G | - | - | - | - | - | - | - | - | G | L | V | V | G | V | T | A | P | A | F | F | P | E | R | R | G | P | N | P | - | - | - | F | V | D | L | E | L | P | A | A | T | L | P | - | - | Q | R | I | G | R | L | L | D | L | G | A | G | Y | L | A | L | P | G | G | - | - | - | - | - | - | V | G | T | L | A | E | L | V | L | A | W | N | L | L | Y | L | R | - | - | - | R | G | - | - | V | G | R | P | L | A | V | D | - | - | P | Y | W | L | G | L | L | K | A | H | - | - | - | - | - | - | - | G | E | I | A | P | E | D | V | G | L | - | - | L | R | V | V | A | - | - | - | D | E | E | D | L | R | R | F | L | R | S | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | V | L | A | I | T | G | Y | K | P | F | E | L | G | I | F | K | Q | D | D | K | A | L | Y | Y | I | K | K | A | I | K | N | R | L | I | A | F | L | D | E | G | L | E | W | I | L | I | S | G | Q | - | L | G | V | E | L | W | A | A | E | A | A | Y | D | - | - | - | L | Q | E | E | Y | P | D | L | K | V | A | V | I | T | P | F | Y | E | Q | E | - | - | - | - | - | - | - | - | - | K | N | W | K | - | - | - | - | - | - | - | E | P | N | K | E | Q | Y | E | A | V | L | A | Q | A | D | Y | E | A | S | L | T | H | - | R | P | Y | E | S | P | L | Q | F | K | Q | K | N | Q | F | F | I | D | K | S | - | - | - | - | - | - | - | - | - | - | - | - | D | G | L | L | L | L | Y | D | P | E | K | E | G | S | P | K | Y | M | L | G | T | A | E | K | R | R | E | Q | - | - | - | D | G | - | - | - | Y | P | I | Y | F | I | T | - | - | - | - | - | - | - | M | D | D | L | R | V | T | V | E | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | K | S | K | F | R | R | I | C | V | F | C | G | S | S | Q | G | - | - | - | - | - | - | - | - | - | K | K | S | S | Y | Q | D | A | A | V | D | L | G | N | E | L | V | S | R | N | - | I | D | L | V | Y | G | G | G | S | I | G | L | - | G | L | V | S | Q | A | V | H | D | G | G | - | - | - | - | - | - | - | - | R | H | V | I | G | I | I | P | K | T | - | - | - | - | - | - | - | - | - | - | - | - | - | L | V | G | E | V | R | A | V | A | D | H | - | - | - | Q | R | K | A | E | A | - | K | H | S | D | A | F | I | A | L | P | G | G | - | - | - | - | - | - | Y | G | T | L | E | E | L | L | E | V | I | T | W | A | Q | L | G | - | - | - | - | - | - | I | H | D | K | P | V | G | L | L | N | V | D | G | Y | Y | N | S | L | L | S | F | I | D | K | A | V | E | E | G | F | I | S | P | T | A | R | E | I | - | - | I | V | S | A | P | - | - | - | T | A | K | E | L | V | K | K | L | E | E | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | Q | E | E | P | N | M | V | V | C | W | G | G | H | S | I | N | - | - | - | - | - | - | E | - | - | - | - | - | I | E | Y | K | Y | T | K | D | V | G | Y | H | I | G | L | R | G | - | L | N | I | C | T | G | C | G | - | P | G | A | M | K | G | P | M | K | G | A | T | I | G | H | A | K | Q | R | V | - | E | G | G | R | Y | L | G | L | T | E | P | G | I | I | A | A | E | - | P | P | N | P | - | - | - | I | V | N | E | L | V | I | L | P | D | I | E | - | - | K | R | L | E | A | F | V | R | C | A | H | G | I | V | I | F | P | G | G | - | - | - | - | - | - | A | G | T | A | E | E | L | L | Y | L | L | G | I | L | M | H | P | - | - | - | D | N | Q | R | Q | S | L | P | V | I | L | T | G | P | A | S | S | R | D | Y | F | E | A | L | D | E | F | I | G | A | - | - | T | I | G | D | E | A | R | Q | L | - | - | Y | K | I | I | I | D | - | - | D | P | A | A | V | A | Q | H | M | H | A | G | M | A | A | V | K | Q | Y | R | R | D | S | G | D | A | Y | Y | F | N | W | T | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | E | P | A | I | T | K | I | I | S | G | G | Q | - | T | G | A | D | R | A | A | L | D | F | A | I | K | - | - | - | H | - | - | - | - | - | H | I | P | Y | G | G | W | V | P | K | G | R | L | A | E | G | G | R | V | P | E | T | Y | Q | L | Q | E | P | - | - | - | - | - | T | S | D | Y | S | K | R | T | E | K | N | V | L | D | S | D | G | T | L | I | I | S | H | - | G | - | - | - | - | I | L | G | G | S | A | L | T | E | F | F | A | E | Q | Y | - | - | - | - | - | - | - | - | - | - | - | - | K | P | C | L | H | I | D | L | D | R | - | - | I | S | I | E | D | A | A | T | L | I | N | S | W | T | V | S | - | - | - | H | H | I | Q | V | L | N | I | A | G | P | R | A | G | K | D | P | E | I | Y | Q | A | T | D | L | L | E | V | F | L | A | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | R | Q | W | A | V | C | V | Y | C | A | S | G | P | T | - | - | - | - | - | - | - | - | - | - | H | P | E | L | L | E | L | A | A | E | V | G | S | S | I | A | A | R | G | - | W | T | L | V | S | G | G | G | N | V | S | A | M | G | A | V | A | Q | A | A | R | A | K | G | - | - | - | - | - | - | - | - | G | H | T | V | G | V | I | P | K | A | L | V | H | R | E | L | - | A | D | V | - | - | - | D | A | A | E | L | I | V | T | D | T | M | - | - | - | R | R | K | R | E | M | E | H | R | S | D | A | F | I | A | L | P | G | G | - | - | - | - | - | - | I | G | T | L | E | E | F | F | E | A | W | T | A | G | Y | L | G | - | - | - | - | - | - | M | H | D | K | P | L | I | L | L | D | P | F | G | H | Y | D | G | L | L | T | W | L | R | G | L | V | P | T | G | Y | V | S | Q | R | A | M | D | S | - | - | L | V | V | V | D | - | - | - | N | V | - | A | A | L | E | A | C | A | P | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | S | F | S | I | L | D | D | C | Y | P | W | D | L | S | E | I | Y | D | A | P | V | L | L | F | Y | K | G | N | L | D | L | L | K | F | P | K | V | A | V | V | G | S | R | A | C | S | - | - | - | - | - | - | - | - | - | - | - | K | Q | G | A | K | S | V | E | K | V | I | Q | G | L | - | - | E | N | E | L | V | I | V | S | G | L | A | - | K | G | I | D | T | A | A | H | A | A | L | Q | N | - | - | - | - | - | - | - | - | - | G | G | K | T | I | A | V | I | G | T | G | - | L | D | - | - | - | - | - | - | - | - | - | V | F | Y | P | - | - | - | - | - | - | - | K | A | N | K | R | L | Q | D | Y | I | G | N | D | H | L | V | L | S | E | Y | G | P | - | G | E | Q | P | L | K | F | H | F | P | A | R | N | R | I | I | A | G | L | C | - | - | - | - | - | - | - | - | - | - | - | - | R | G | V | I | V | A | E | A | K | - | - | - | - | - | - | R | S | G | S | L | I | T | C | E | R | A | E | E | - | - | - | - | G | - | - | - | R | D | V | F | A | I | P | G | S | I | L | D | G | L | S | D | G | C | H | H | L | I | Q | E | G | A | - | - | - | - | - | - | - | K | L | V | T | S | G | Q | D | V | L | A | E | F | E | F | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| W | K | H | A | D | P | W | R | V | L | R | I | Q | S | E | F | V | A | G | F | D | A | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | H | E | M | P | K | A | V | T | V | F | G | S | A | R | I | K | - | - | - | - | - | - | E | D | H | - | - | - | P | Y | Y | K | A | G | V | E | L | G | E | K | L | V | A | A | D | - | Y | A | V | V | T | G | G | G | - | P | G | L | M | E | A | P | N | K | G | A | S | E | A | N | - | - | - | - | - | - | - | G | L | S | V | G | L | G | I | E | L | P | H | - | - | - | - | - | H | L | N | P | - | - | - | Y | V | D | L | G | L | N | F | R | Y | F | F | - | - | A | R | K | T | M | F | L | K | Y | S | Q | A | F | V | C | L | P | G | G | - | - | - | - | - | - | F | G | T | L | D | E | L | F | E | V | L | C | M | V | Q | - | - | - | - | - | T | G | K | V | T | N | F | P | I | V | L | I | G | T | E | F | W | A | G | L | V | D | W | I | R | H | R | L | V | E | E | G | M | I | D | E | K | D | V | D | R | - | - | M | - | L | V | T | D | - | - | D | L | D | Q | A | V | K | F | I | V | D | A | H | A | - | - | - | - | - | - | - | - | - | G | - | - | - | - | - | - | - | - | L | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | P | K | I | F | R | K | K | V | C | T | T | F | I | T | T | E | K | P | T | D | A | Y | G | D | L | D | F | T | Y | S | G | R | K | H | S | N | F | L | R | L | S | D | R | T | D | P | A | T | V | Y | S | L | V | T | R | - | - | - | - | - | - | S | W | G | F | R | A | P | N | L | V | V | S | V | L | G | G | S | G | G | - | - | - | - | - | - | P | V | L | Q | T | W | L | Q | D | L | L | R | R | G | L | V | R | A | A | Q | S | T | G | - | A | W | I | V | T | G | G | L | H | T | G | I | G | R | H | V | G | V | A | V | R | D | H | Q | T | A | S | T | G | S | S | K | V | V | A | M | G | V | A | P | W | G | V | V | R | N | R | D | M | L | I | N | P | K | G | S | F | P | A | R | Y | R | W | R | G | D | P | E | D | G | V | E | F | P | L | D | Y | N | Y | S | A | F | F | L | V | D | D | G | T | Y | G | R | L | G | G | E | N | R | F | R | L | R | F | E | S | Y | V | A | Q | Q | K | T | G | V | G | G | T | G | I | D | I | P | V | L | L | L | L | I | D | G | - | D | E | K | M | L | K | R | I | E | D | A | T | Q | A | - | - | - | - | - | - | - | - | Q | L | - | - | P | C | L | L | V | A | G | S | G | G | A | A | D | C | L | V | E | T | L | E | E | A | R | D | R | I | R | R | Y | F | P | K | G | D | P | E | V | L | Q | A | Q | V | E | R | I | M | T | R | K | E | L | L | T | V | Y | S | S | E | D | G | S | E | E | F | E | T | I | V | L | R | A | L | V | K | A | C | G | S |
| Member | Chain | UniProtKB | GO term |
|---|---|---|---|
| d1rcua_ | A | Q9X0E5 | GO:0005829 |
| d1rcua_ | A | Q9X0E5 | GO:0005829 |
| d1t35a_ | A | O06986 | GO:0005829 |
| d1t35a_ | A | O06986 | GO:0009691 |
| d1t35a_ | A | O06986 | GO:0016787 |
| d1t35a_ | A | O06986 | GO:0016799 |
| d1t35a_ | A | O06986 | GO:0005829 |
| d1t35a_ | A | O06986 | GO:0009691 |
| d1t35a_ | A | O06986 | GO:0016787 |
| d1t35a_ | A | O06986 | GO:0016799 |
| d1weha_ | A | Q5SLJ9 | GO:0005829 |
| d1weha_ | A | Q5SLJ9 | GO:0016020 |
| d1weha_ | A | Q5SLJ9 | GO:0005829 |
| d1weha_ | A | Q5SLJ9 | GO:0016020 |
